时空转录组FF V1.3在保持Stereo-seq技术“纳米级分辨率”和“厘米级全景视场”的两大优势下,通过优化时空试剂、酶、探针设计以及算法软件完成性能优化与提升。升级后,其捕获效率大幅提升,工作流程更快速简便,用户友好性显著增强,可助力研究人员轻松获得每个细胞内的更多高质量转录本数据,通过华大自主研发的CellBin(点击了解详情>>>Stereo-seq细胞分割CellBin,精准单细胞研究利器)细胞分割算法精准刻画组织结构空间特征,为各种应用场景下的科学研究提供无与伦比的洞察力。
01 不同版本数据对比,基因捕获能力显著提升
表1:时空转录组FF V1.2与V1.3的数据情况对比
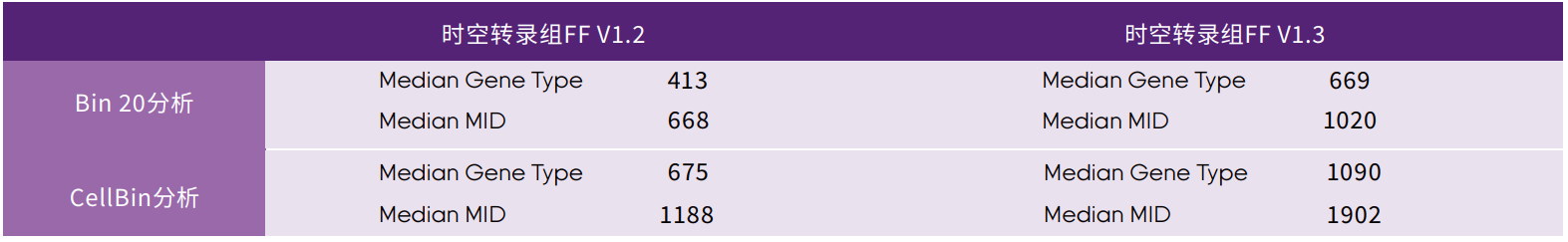
从表1可以看出,同样在2G reads左右的测序量下,Bin20和CellBin水平分析中,V1.3中MID与Gene的捕获中位数均明显优于V1.2。
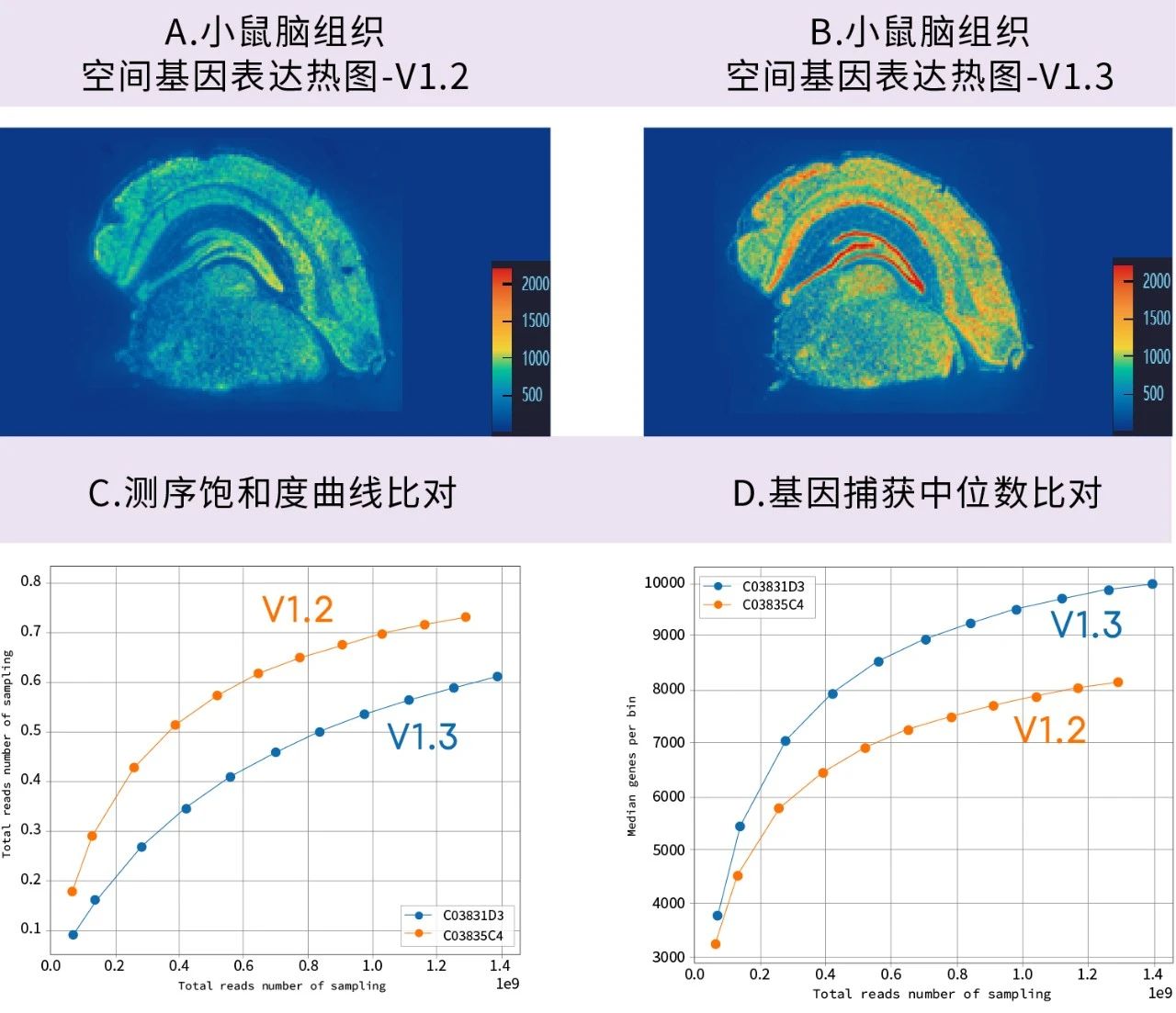
小鼠脑组织中时空转录组FFV1.2和V1.3捕获结果对比
对小鼠脑组织的相邻切片,分别使用时空转录组FF V1.2(A)和V1.3(B)进行转录组捕获,测序量均为2G左右,V1.3的测序饱和度更低(C),且在Bin20水平捕获的基因中位数明显高于V1.2(D)。
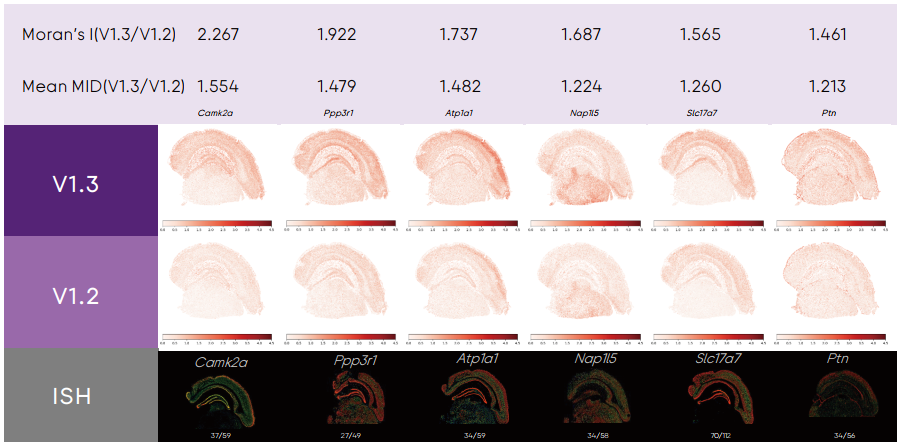
V1.3与V1.2版本对具有空间特异性的基因捕获一致
通过采用 Moran's I统计方法,对 V1.3和V1.2版本产出的基因表达数据进行空间自相关度分析,以识别在各版本中具有显著空间特征的基因。结果表明,两个版本均能有效捕获空间特异性基因。在V1.3和V1.2中,这些基因的空间分布规律一致,并且与原位杂交(ISH)[1]结果吻合。
表2:相同小鼠脑样本使用不同版本试剂进行实验的数据产出情况

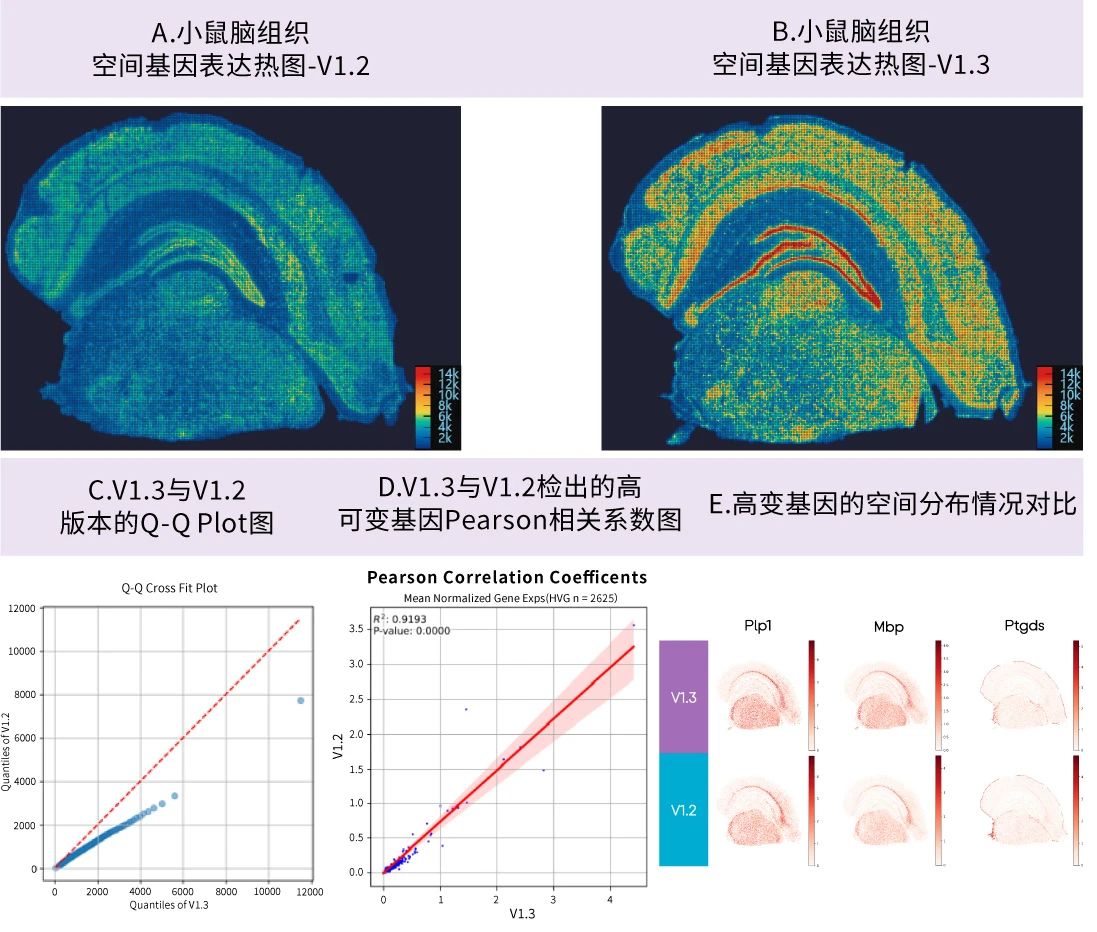
不同版本试剂产出的数据结果比对
从空间基因表达热图可见V1.3(B)产出数据的基因表达丰度明显高于V1.2(A);利用小鼠脑相邻切片的CellBin水平下Total MID Counts的分位数,我们绘制了Q-Q Cross Fit Plot图(C),结果可见,V1.3的捕获能力更强;虽然两个版本的捕获能力存在差异,但是高变基因的检出一致性较好(D);此外,选取了3个高变基因,分析表明,同一高变基因在不同版本中具有一致的空间分布规律(E)。
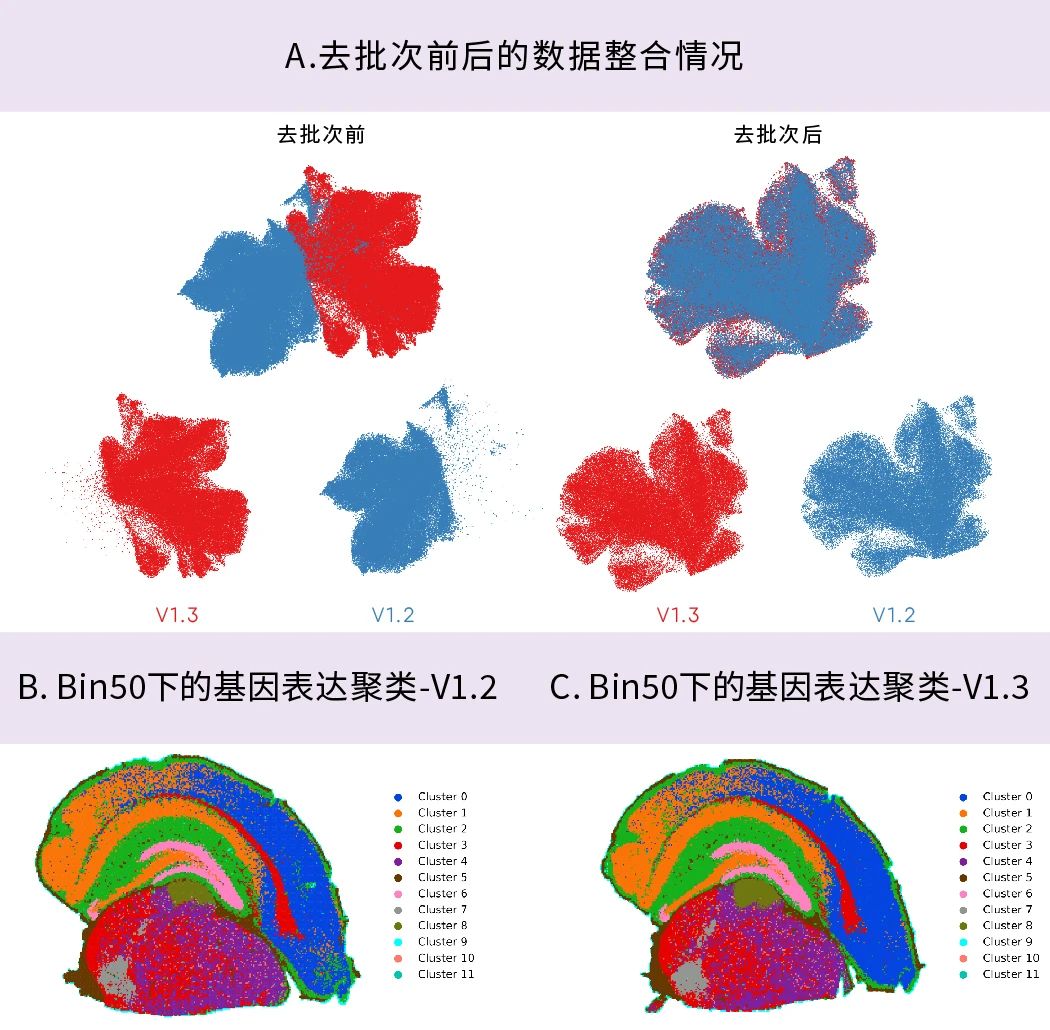
两个版本数据的去批次整合情况
使用小鼠脑组织相邻切片分布进行V1.3和V1.2实验,通过分析结果可以看到,V1.2和V1.3产出的数据在未去批次前(A 左图)存在数据差异,通过Stereopy(https:/stereopy.readthedocs.io/)的Harmony去批次算法[2]分析,两个版本的数据即可顺利整合分析(A 右图);其Bin50水平的细胞聚类结果,在V1.2(B)和V1.3(C)中高度吻合。
时空转录组FF V1.3不仅可应用于人和小鼠,也可对其他物种进行时空全转录组信息挖掘。目前,时空转录组FF V1.3已测试30多种人、小鼠以及其他物种的组织类型。点击查看测试数据:






