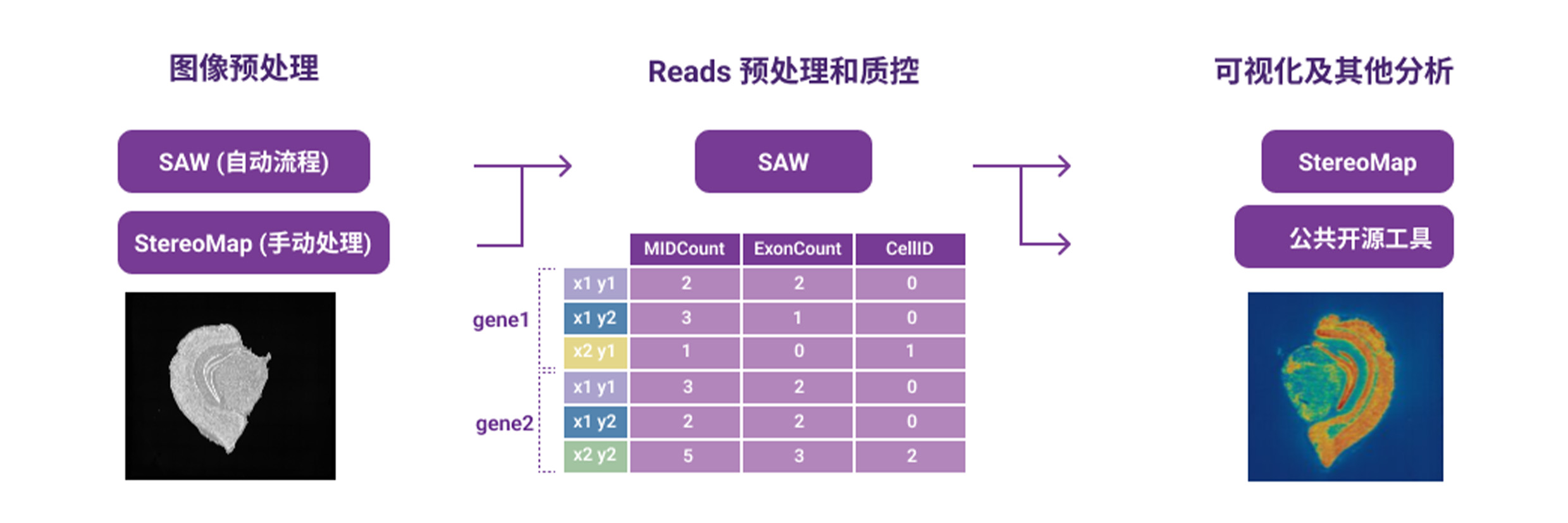
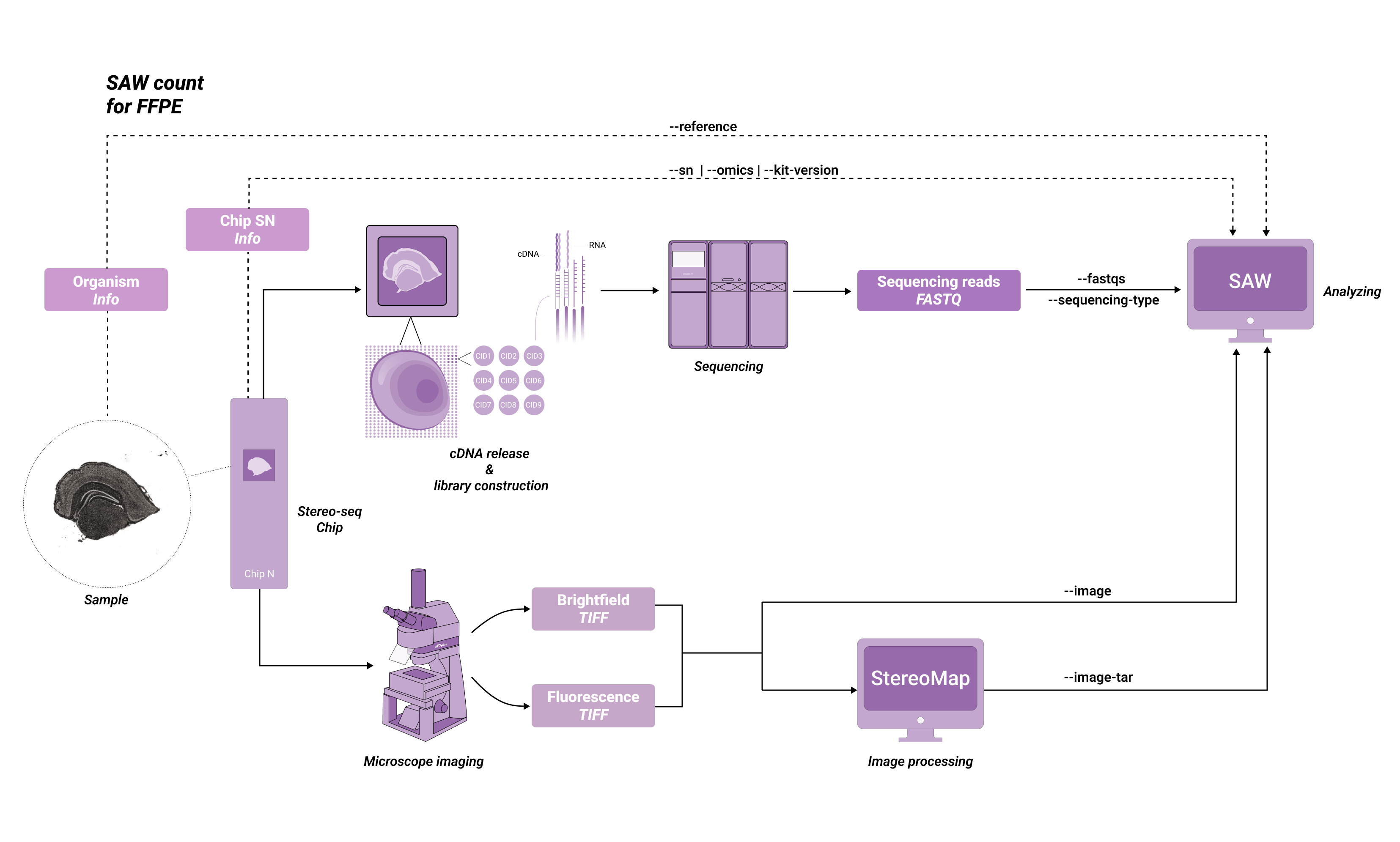
需要注意:一、新版本软件可兼容旧版本功能;二、三个软件必须配套使用,即软件的不同版本号之间需要配套。现有不同版本号间的配套规则如下:
全部
搜产品
搜资源
搜新闻
搜问答
搜索