1. 文件格式:
○ GFF文件 或者 GTF文件,文件后缀名支持 gtf/gtf.gz,gff/gff.gz,gff3/gff3.gz
2. GTF文件格式:
○ 注释行以 # 开始
○ 主体部分共 9列,以tab作为分隔符:seqname source feature start end score strand frame attributes
▪ type:注释信息的类型必须含有gene, transcript 和 exon
▪ start/end:最大值需小于2^31
▪ strand:链的正向与负向,分别用加号+和减号-表示。
▪ 第9列为attributes,格式为tag "value"(标签“值”),不同属性之间以空格相隔 ; 必须要有以下4个
• gene_name value
• gene_id value: 表示转录本在基因组上的基因座的唯一的ID。gene_id与value值用空格分开,如果值为空,则表示没有对应的基因。
• transcript_name value
• transcript_id value: 预测的转录本的唯一ID。transcript_id与value值用空格分开,空表示没有转录本。
○ 目前最大有效基因数必须小于2^20,即1048576
○ 不可以乱序,即同一个gene的transcript/exons需按顺序排列
3. GFF文件格式:
○ 注释行以 # 开始
○ 主体部分共 9列,以tab作为分隔符:seqid source type start end score strand phase attributes
▪ type:注释信息的类型必须含有gene, mRNA和 exon
▪ start/end:最大值需小于2^31
▪ strand:“+”表示正链,“-”表示负链,“.”表示不需要指定正负链,“?” 表示未知
▪ 第9列为attributes,格式为tag=value (标签=值),不同属性之间以分号相隔
• 需要存在ID Name Parent(对gene无需判断Parent)
• 对于第三列的命名规则请务必仔细研究 ⇒ “树状分级” (不能只列出child行 而没有parent行!)示例如下:
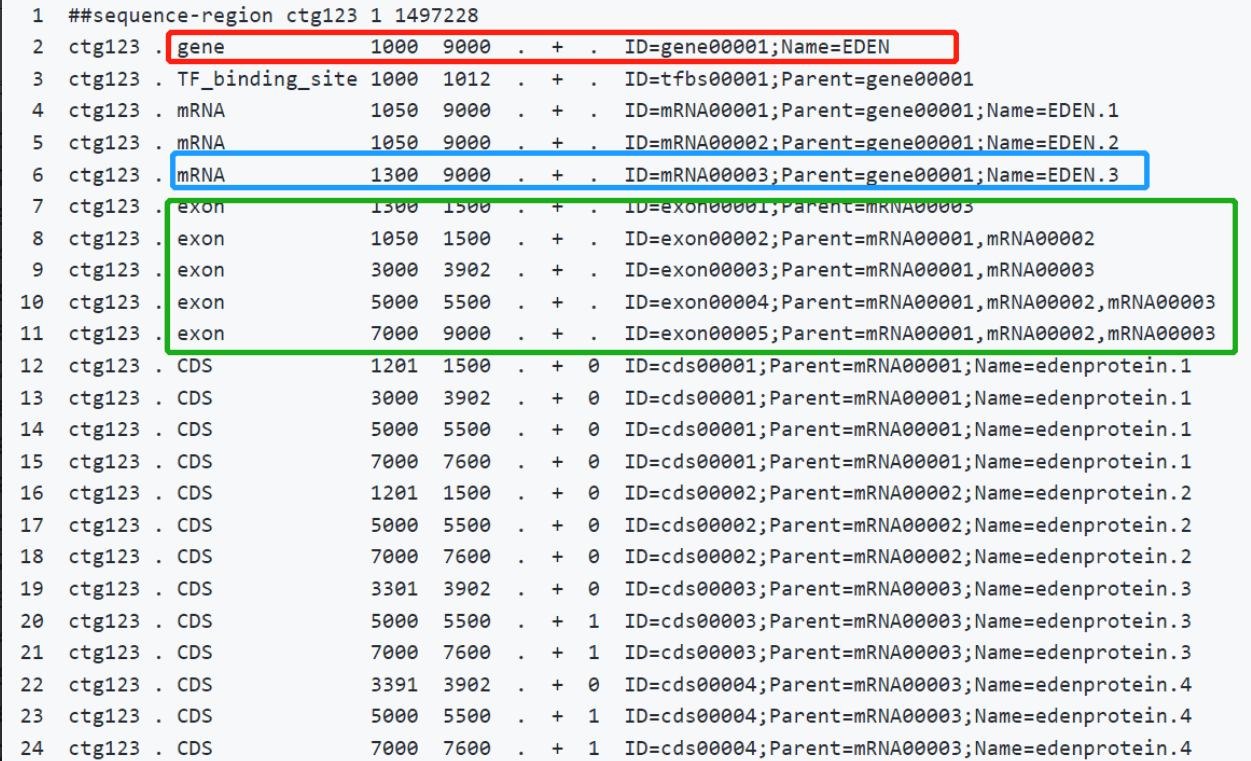
○ 目前最大有效基因数必须小于2^20,即1048576
○ 可以乱序,但仍需满足 gene必须出现在对应mRNA之前,mRNA必须出现在对应的exon之前的规则
4. 其他注意事项:
○ gene/gene_name(基因的名字) 值不含有特殊符号(空格,各类型括号,引号,<>,%等)。支持使用的常见特殊符号有”_“,"."。
○ gene/gene_name(基因的名字) 值长度小于64个字符
○ 虽然GFF文件现在大部分使用的都是第三版(GFF3),但是文件命名时请命名为.gff ;同理对于GTF文件也请文件命名时采用.gtf