华大时空组学技术Stereo-seq是一项基于DNA纳米球测序芯片自主研发、可实现超高通量和超高精度的全景式时空转录组技术,它将认识生命空间的分辨率提升至500 nm的亚细胞层级,相比国际同类技术分辨率提升了40,000倍。Stereo-seq目前已被成功应用到动物胚胎发育、植物研究、脑科学、肿瘤等多个领域,并助力研究人员在Cell、Science、Nature、Cell Research等顶级学术期刊上发表了40余篇文章。
近日,华大时空组学STOmics与百奥智汇达成战略合作,百奥智汇正式成为华大时空组学STOmics生态合作伙伴。百奥智汇在单细胞及空间转录组平台积累了丰富的服务经验,搭建了完善的单细胞及空间组学服务平台,仪器设备先进齐全,提供从样品制备到数据分析的一站式服务,尤其在数据分析方面,业内首创定制化分析服务模式,专业的科学家团队为优质高效的项目产出保驾护航。
此次百奥智汇全面引进华大时空组学技术,将为研究人员提供更高精度的空转检测服务,助力广大科研工作者在生命结构、疾病机制、生长发育机制等研究领域获得新的认知。

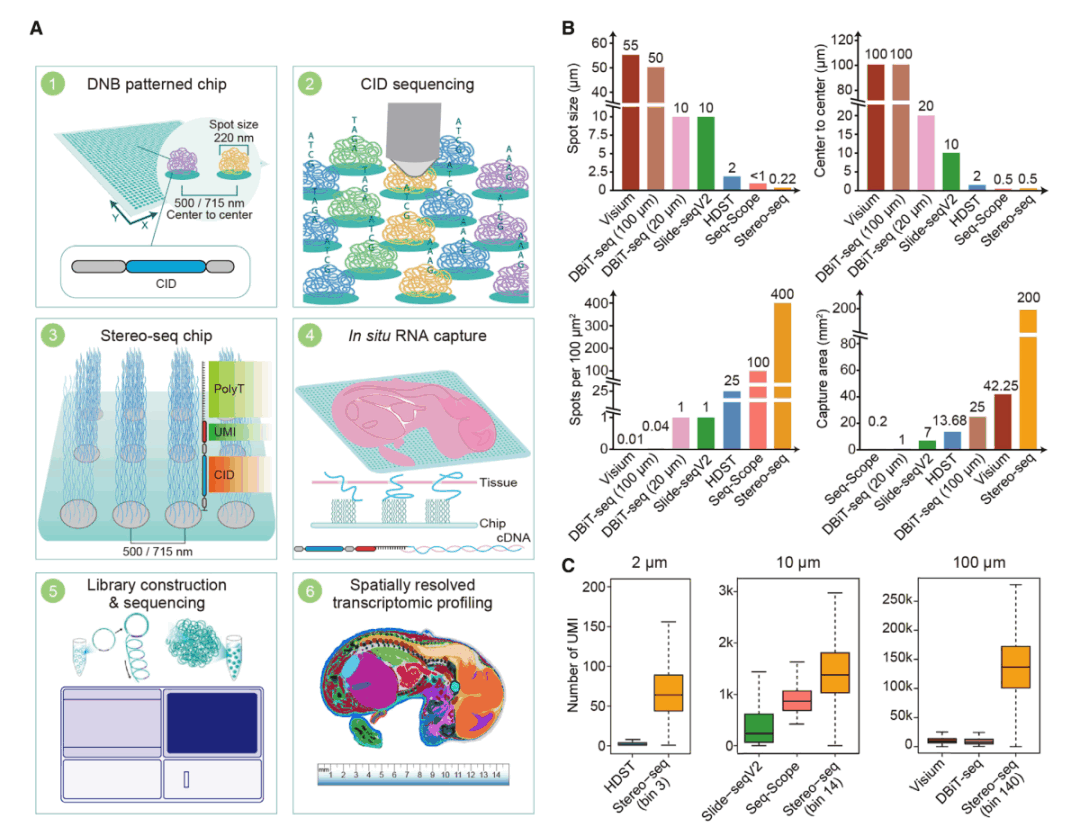
01 纳米级分辨率,实现亚细胞级分子定位
Stereo-seq的检测分辨率可达到500 nm,能实现对单个细胞及分子信息进行空间定位和检测。
02 厘米级全景视场,检测组织大小可定制
Stereo-seq设计的常规芯片大小为1 cm×1 cm,同时可依据组织大小定制不同尺寸的芯片。华大时空组学现已推出时空芯片S0.5,该芯片尺寸为0.5 cm×0.5 cm,可灵活兼容现有技术流程,极大地提升芯片利用率,同时依旧可以实现高水平的细胞分割。未来,华大时空组学还将根据实际科研需求推出大尺寸芯片。
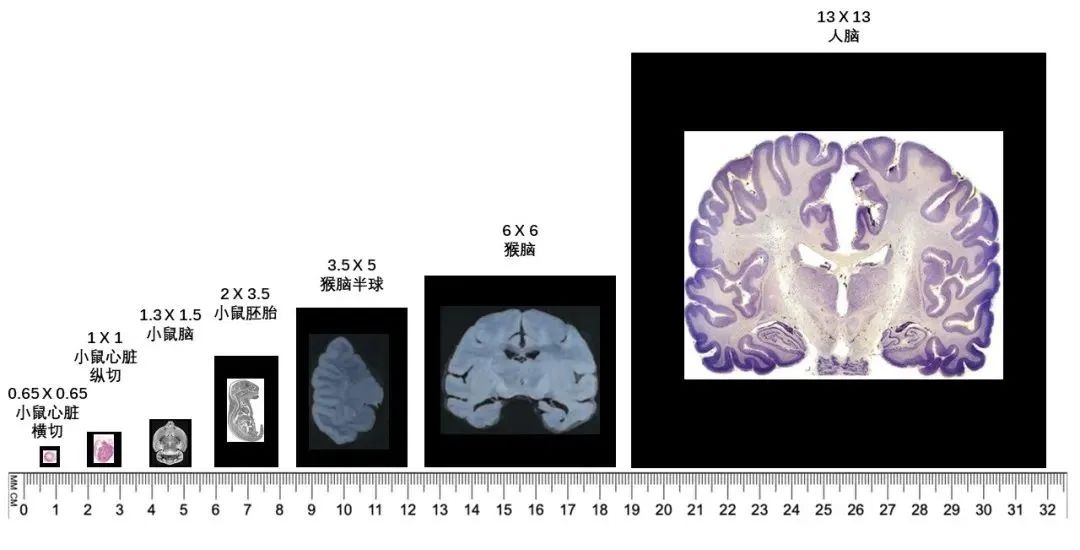
03 物种组织不设限,人、动物及植物多覆盖
Stereo-seq技术目前已研发了针对人、不同动物及植物的各部位组织器官的检测方法,完成了多种动植物的组织器官研究。
04 自主研发时空云平台STOmics Cloud,加速科研转化
Stereo-seq配套搭建了领先的多组学数据分析平台时空云平台STOmics Cloud,支持用户在线进行时空组学数据挖掘和分析结果可视化,全链助力时空科研成果产出。
同时,时空云平台STOmics Cloud基于大语言模型,创新建立了人工智能应用StereoCopilot,可以灵活辅助研究人员进行科研设计、数据分析及文章撰写等。
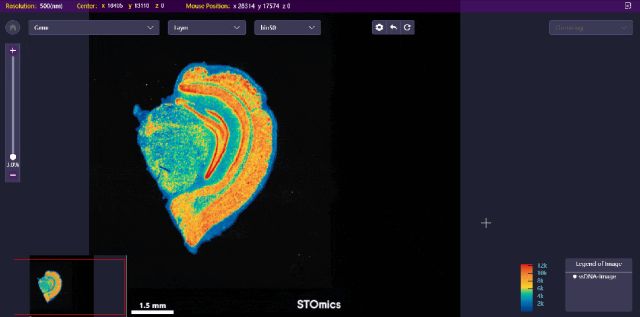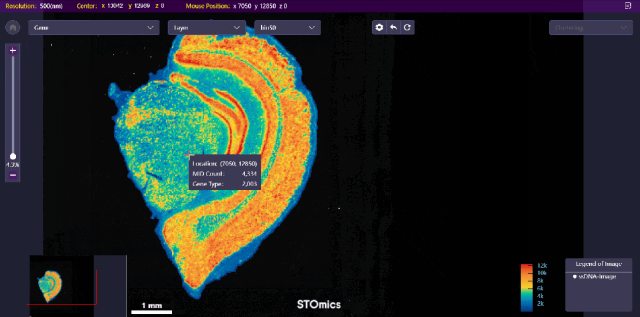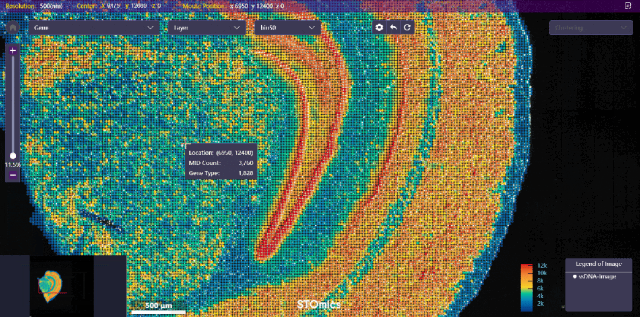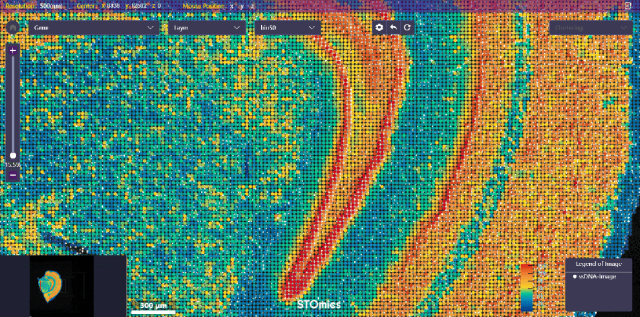
小鼠大脑演示数据
关于百奥智汇
百奥智汇成立于2018年,是一家拥有全球视野的生命科学技术公司,致力于将单细胞机理研究平台和生物信息学大数据/AI平台充分应用于癌症等重大人类疾病的诊断和治疗中,充分利用自身强大的科研能力、自有平台和数据,创建人类疾病的精准细胞图谱,寻找具突破性的疾病诊断和治疗靶标、开发颠覆性的治疗方法。
目前公司为生命科学和医学研究者提供国际领先的单细胞及空间多组学测序、定制化生物信息分析、生物信息可视化平台搭建、治疗靶标和生物标志物挖掘等技术服务和全流程解决方案。公司成立以来已与300余家医院、药企及科研院所建立合作关系,已支持客户在Cell、Science、Cancer Cell、European Heart Journal、Cell Discovery、Gut等期刊发表多篇高质量文章,单篇平均影响因子超过32。
关于华大时空
华大作为时空组学领域的重要推动者,在2020年发布了自主研发的时空组学技术Stereo-seq,华大时空组学STOmics应运而生,助力生老病死、意识起源、万物生长、生命起源等重大科学问题的研究与应用。
2022年,凭借能同时实现“纳米级分辨率”和“厘米级全景视场”的技术优势,Stereo-seq助力研究人员陆续在Cell、Nature和Science三大顶级学术期刊发表胚胎发育、器官再生、疾病等领域的重要成果。
秉承华大集团“基因科技造福人类”的使命,华大时空组学STOmics将积极推进全球科研合作,同时致力于促进时空组学技术的产业化应用,为科研和临床领域带来新的认知及价值。
参考文献:
Chen A, Liao S, Cheng M, et al. Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays[J].Cell, 2022,185(10):1777-1792.e21. doi:10.1016/j.cell.2022.04.003.