近日,德克萨斯大学安德森癌症中心(The University of Texas MD Anderson Cancer Center)研究团队通过整合华大自主研发的时空组学技术Stereo-seq、空间质谱和免疫组化等检测方法,开发出了一种能够有机结合多种空间组学数据的分析流程。
研究人员使用该分析流程检测了两种福尔马林固定的石蜡包埋(formalin-fixed paraffin embedded,FFPE)肿瘤样本的空间多组学数据,构建了具有单细胞分辨率的三维(three-dimensional,3D)空间多组学图谱。随后,研究人员通过该图谱识别了FFPE肿瘤样本中的3D肿瘤-免疫微环境(tumor-immune microenvironment,TIME)中各种细胞类型和细胞外基质(extracellular matrix,ECM)之间的串扰信号网络,揭示了TIME中各种细胞类型和空间分布以及细胞的激活通路和电生理状态。该图谱不仅提供了TIME中细胞的空间异质性的分子见解,还将为开发新的肿瘤预测标志物和治疗靶点提供有价值的参考。
该研究涉及的空间多组学平台包括非靶向质谱成像(包括聚糖、代谢物和肽)、Stereo-seq(时空组学)和靶向seqIF(免疫组化蛋白质组学)。相关成果于2024年2月20日发表在Cancers,以下是文章的详细解读。

文章题目:Molecular, metabolic, and subcellular mapping of the tumor immune microenvironment via 3D targeted and non-targeted multiplex multi-omics analyses
发表时间:2024-02-20
发表期刊:Cancers
主要研究团队:德克萨斯大学安德森癌症中心等
影响因子:5.200
DOI:10.3390/cancers16050846
大多数用于实体肿瘤免疫微环境(tumor–immune microenvironment,TIME)分子重建的平台无法以单细胞分辨率探索三维(three-dimensional,3D)空间背景,因此缺乏关于细胞间或细胞与细胞外基质(cell–extracellular matrix,ECM)的相互作用信息。
最近,已有多种空间多组学平台可对新鲜冷冻样本和福尔马林固定石蜡包埋(formalin-fixed paraffin embedded,FFPE)的样本中的RNA、代谢产物和蛋白进行靶向或非靶向分析。其中包括空间转录组学(spatial transcriptomics,ST)技术、成像质谱流式细胞术(imaging mass cytometry,IMC)、质谱成像(mass spectrometry imaging,MSI)和多重连续免疫荧光(multiplex sequential immunofluorescence,seqIF)。
然而,大多数使用空间非靶向检测方法的平台,不提供真正的单细胞水平分辨率空间多组学数据。此外,大多数实验在特定肿瘤样本的单个组织切片上进行,其中的细胞间相互作用只能在二维平面中观察到。在三维立体空间中,这些细胞可能比在二维平面中观察到的距离更近。因此,采用多切面对空间多组学平台生成的数据进行整合,构建组织的多组学三维空间图谱,可以排除二维平面数据的误导,构建更贴近真实组织形态的空间图谱,进而探索新的细胞间或细胞与ECM间的串扰信号网络,并通过这些机制上的见解推动对肿瘤的研究。
FFPE组织切片来源:高级别浆液性卵巢癌(high-grade serous ovarian cancer,HGSOC)和非典型子宫内膜增生(atypical endometrial hyperplasia,AEH);
分析数据来源:空间多组学平台,包括Stereo-seq、MSI和seqIF。
为生成一个全面的肿瘤组织三维空间多组学图谱,研究人员把厚度85微米肿瘤组织分为3层(layers),在每一层中分别使用三种空间组学技术(MIS、seqIF和Stereo-seq)对组织切片进行检测分析(图1)。通过MIS技术,研究人员依次绘制了来自同一组织切片的代谢物、聚糖和胰蛋白酶肽的定位图,检测到了948种代谢物、164种聚糖和586种肽。通过seqIF分析,研究人员使用Keratin8/18和Col1A作为标记,定义了HGSOC样本的肿瘤、基质和界面区域,并使用泛角蛋白和CD10作为组织分割标记识别了AEH样本的腺上皮、间质和管腔区域。
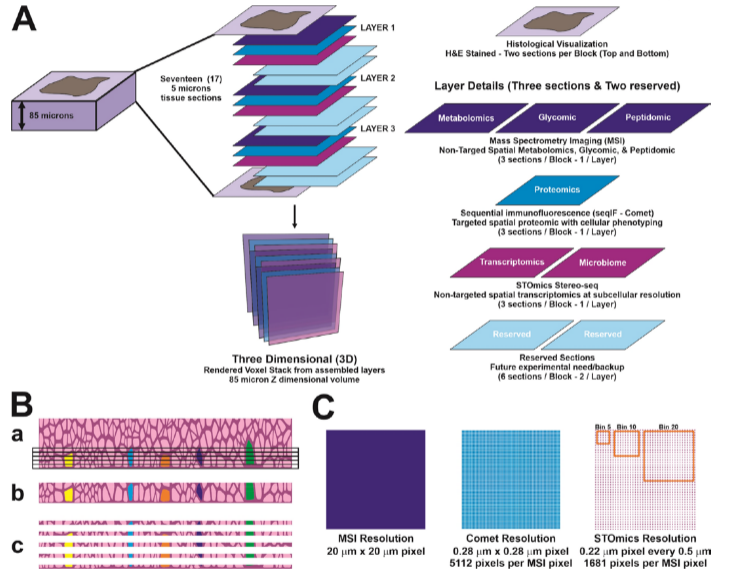
接下来,研究人员使用Stereo-seq N 芯片(1 cm× 1 cm,FFPE)检测了HGSOC和AEH组织样本的3个FFPE切片的空间转录组数据,得到了该切片的全转录组空间表达图谱(图1)。在HGSOC样本的3个FFPE切片中,Stereo-seq捕获的平均基因种类数范围在每100 µm2(bin 20, 10 µm× 10 µm)59.44个到每100 µm2 (bin 20)97.85个;而在AEH样本的3个FFPE切片中,Stereo-seq捕获的平均基因种类数范围在每100 µm2(bin 20)91.64-102个(表1和2)。
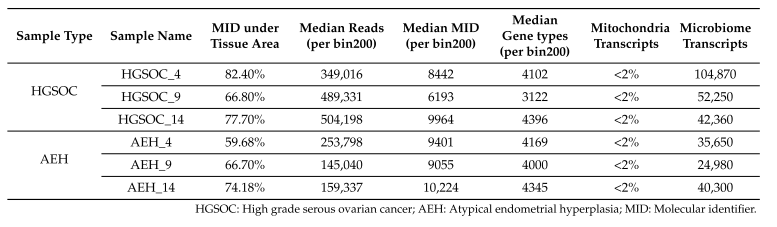
表1 Stereo-seq N芯片(1 cm× 1 cm,FFPE)分析结果
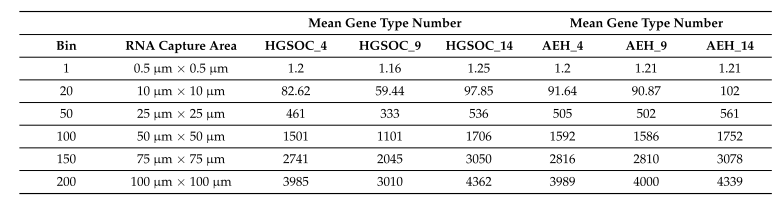
表2 不同bin size的每个bin的平均基因种类数
研究人员通过对Stereo-seq得到的空间转录组数据进行聚类分析,发现肿瘤样本中存在较高的异质性,且在同一个肿瘤组织样本的三个layers间没有明显的批次效应,聚类结果一致性高(图2)。
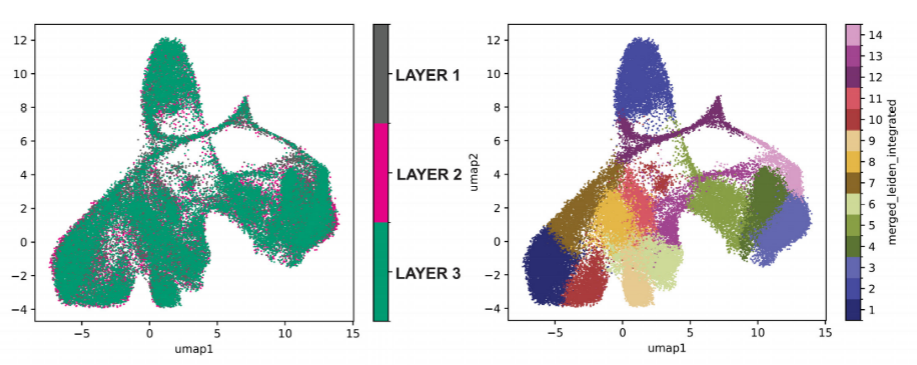
图2 Stereo-seq分析HGSOC样本的UMAP可视化
此外,Stereo-seq还同时检测到了同一组织切片中的微生物空间转录组。每张组织切片中总微生物组转录本数范围在24,980-104,870(表1),其中鉴定出了多种细菌、真菌和病毒,大部分属于放线菌纲(Actinomycetia)和伽马变形菌纲(Gammaproteobacteria)(图3和4)。
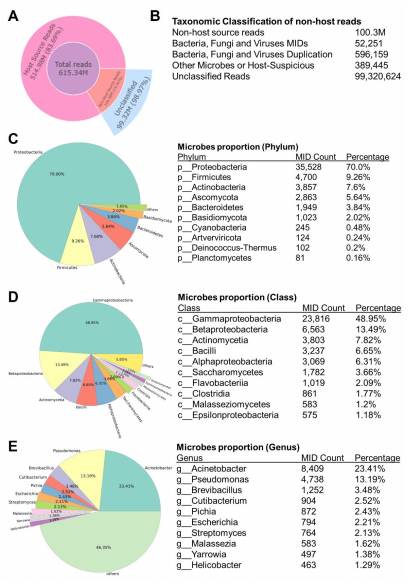
图3 Stereo-seq分析HGSOC样本的微生物空间转录组结果
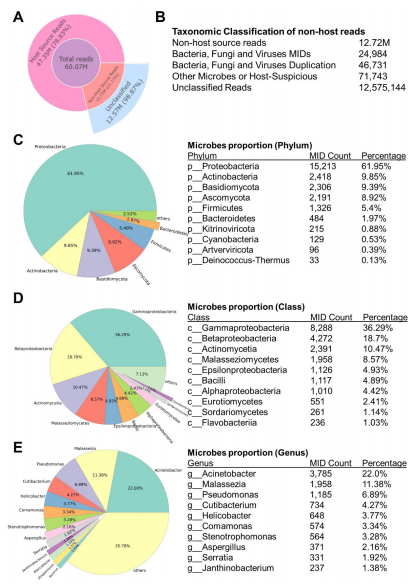
图4 Stereo-seq分析AEH样本的微生物空间转录组结果
该研究文章将Stereo-seq FFPE技术与其他非靶向空间转录组检测方法比较总结,Stero-seq具有以下几点优势:
✔Stereo-seq拥有迄今为止最高的分辨率,且至少高一个数量级,可以对面积在bin 5(2.5μm × 2.5μm)和bin 10(5μm × 5μm)的目标区域进行高分辨率空间转录组检测;
✔Stereo-seq FFPE技术采用随机探针的策略,可以非靶向捕获组织中的RNA,能够检测到mRNA及非mRNA的信息,例如上述对微生物转录组的检测;
✔Stereo-seq还可能在3D空间中检测和量化突变的和可变剪切转录本。
该研究的数据整合流程为:利用Stereo-seq得到的空间转录组表达矩阵作为多模态数据整合框架,并与MIS和seqIF数据进行对齐,从而完成了多模态数据的整合。这种跨模态比较有助于在每个细胞类群基础上调查它们之间的差异基因表达模式,或者在Stereo-seq生成的表达矩阵中重新以更高的分辨率采样,得到与其他空间组学相映射的单细胞水平的转录本数据。
该研究通过整合FFPE样本不同切面的空间多组学(包括Stereo-seq、MIS和seqIF)数据,生成了不同组织的三维多组学空间图谱。这种利用不同的空间组学检测技术构建的TIME空间多组学图谱,可以弥补单一检测技术的局限性。利用该图谱,研究人员探索了肿瘤组织的三维空间结构并深入了解了其细胞邻域的构建方式,这些结果将有助于对TIME建立更全面的了解。该方法未来将在癌症研究多个层面发挥作用,不仅有助于开发更好的生物学模型来检测肿瘤治疗方法,而且还可能为新的诊疗方案的开发提供线索。







