[1] Chen A, Liao S, Cheng M, et al. Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays. Cell. 2022;185(10):1777-1792.e21. doi:10.1016/j.cell.2022.04.003
[2] Liu C, Li R, Li Y, et al. Spatiotemporal mapping of gene expression landscapes and developmental trajectories during zebrafish embryogenesis. Dev Cell. 2022;57(10):1284-1298.e5. doi:10.1016/j.devcel.2022.04.009
[3] Wang M, Hu Q, Lv T, et al. High-resolution 3D spatiotemporal transcriptomic maps of developing Drosophila embryos and larvae. Dev Cell. 2022;57(10):1271-1283.e4. doi:10.1016/j.devcel.2022.04.006
[4] Xia K, Sun HX, Li J, et al. The single-cell stereo-seq reveals region-specific cell subtypes and transcriptome profiling in Arabidopsis leaves. Dev Cell. 2022;57(10):1299-1310.e4. doi:10.1016/j.devcel.2022.04.011
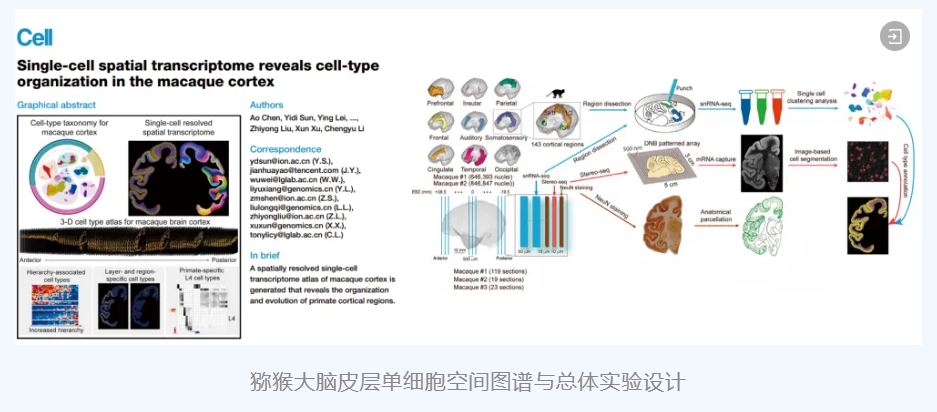
[5] Chen A, Sun Y, Lei Y, et al. Single-cell spatial transcriptome reveals cell-type organization in the macaque cortex. Cell. 2023;186(17):3726-3743.e24. doi:10.1016/j.cell.2023.06.009
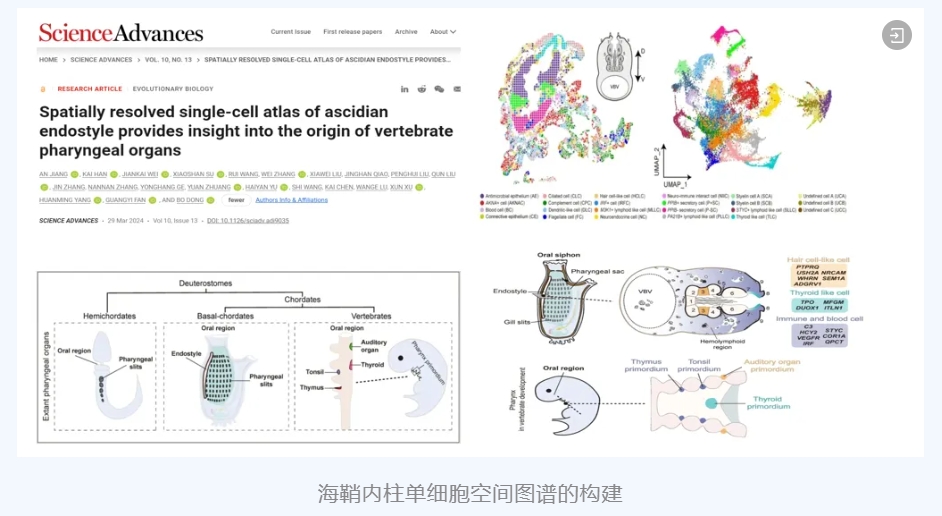
[6] Jiang A, Han K, Wei J, et al. Spatially resolved single-cell atlas of ascidian endostyle provides insight into the origin of vertebrate pharyngeal organs. Sci Adv. 2024;10(13):eadi9035. doi:10.1126/sciadv.adi9035
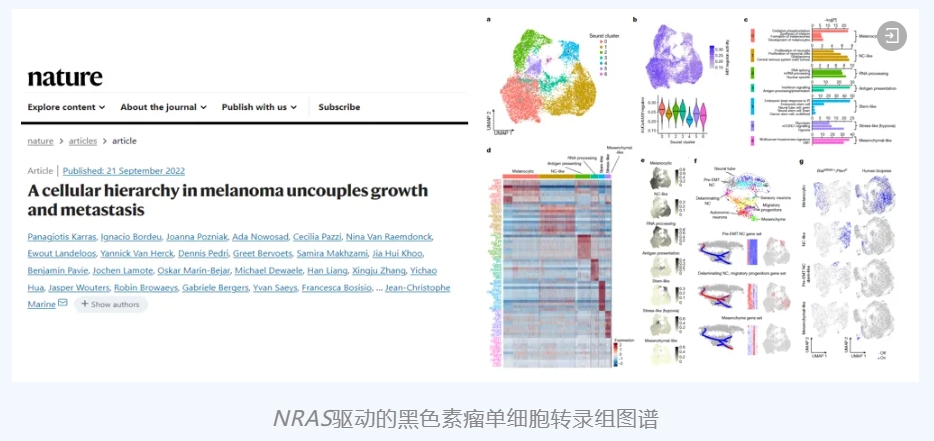
[7] Karras P, Bordeu I, Pozniak J, et al. A cellular hierarchy in melanoma uncouples growth and metastasis. Nature. 2022;610(7930):190-198. doi:10.1038/s41586-022-05242-7
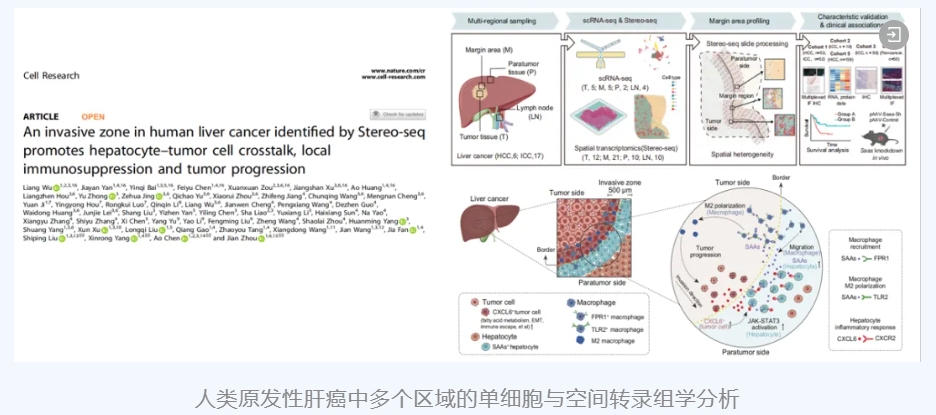
[8] Wu L, Yan J, Bai Y, et al. An invasive zone in human liver cancer identified by Stereo-seq promotes hepatocyte-tumor cell crosstalk, local immunosuppression and tumor progression. Cell Res. 2023;33(8):585-603. doi:10.1038/s41422-023-00831-1
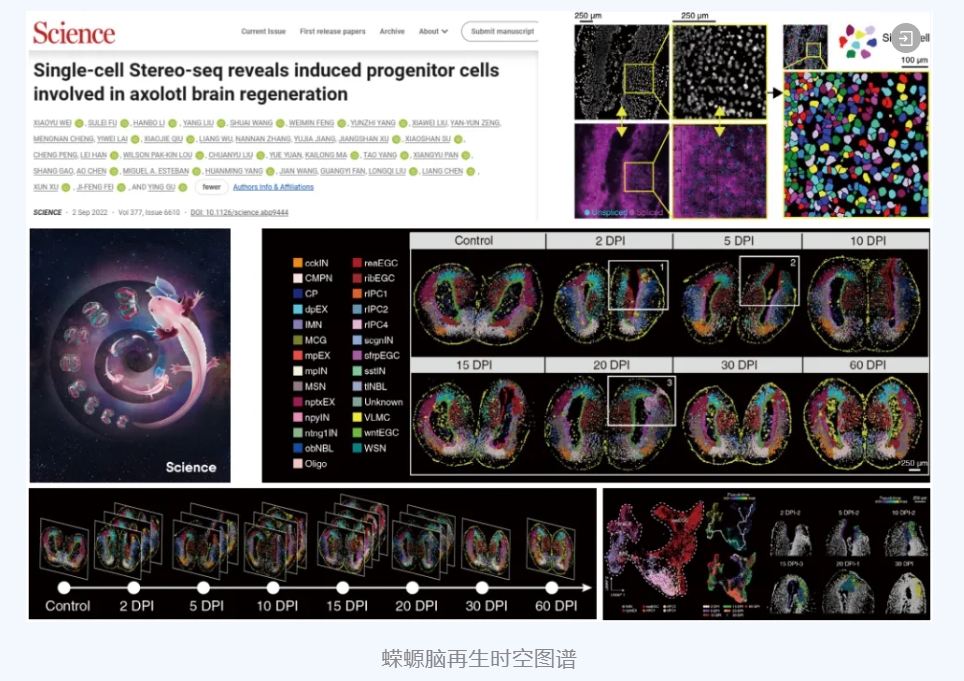
[9] Wei X, Fu S, Li H, et al. Single-cell Stereo-seq reveals induced progenitor cells involved in axolotl brain regeneration. Science. 2022;377(6610):eabp9444. doi:10.1126/science.abp9444
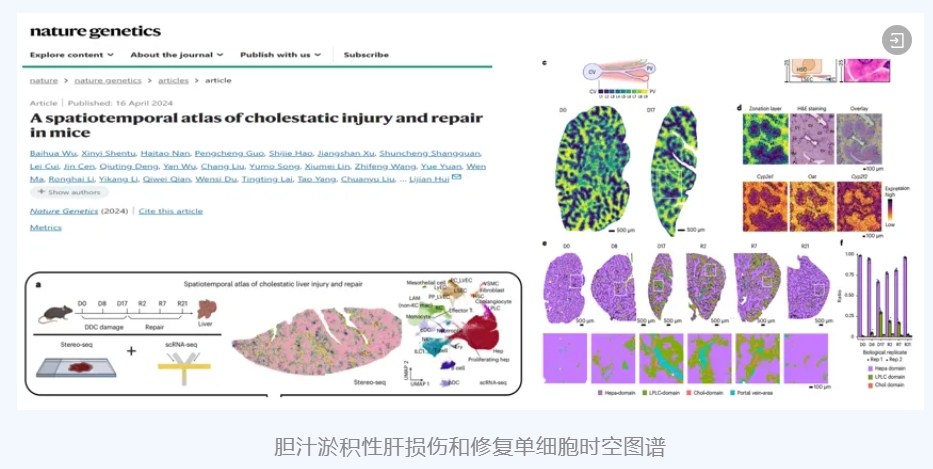
[10] Wu B, Shentu X, Nan H, et al. A spatiotemporal atlas of cholestatic injury and repair in mice. Nat Genet. 2024;56(5):938-952. doi:10.1038/s41588-024-01687-w
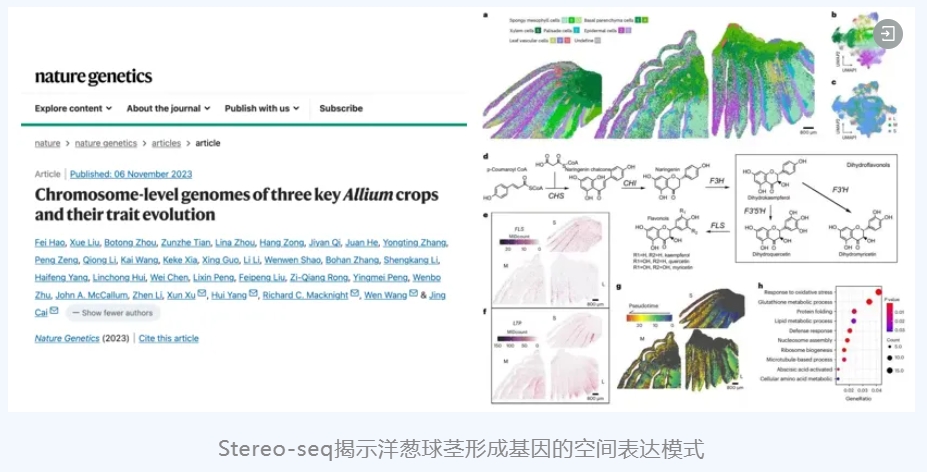
[11] Hao F, Liu X, Zhou B, et al. Chromosome-level genomes of three key Allium crops and their trait evolution. Nat Genet. 2023;55(11):1976-1986. doi:10.1038/s41588-023-01546-0
[12] Wang Y, Luo Y, Guo X, et al. A spatial transcriptome map of the developing maize ear. Nat Plants. 2024;10(5):815-827. doi:10.1038/s41477-024-01683-2