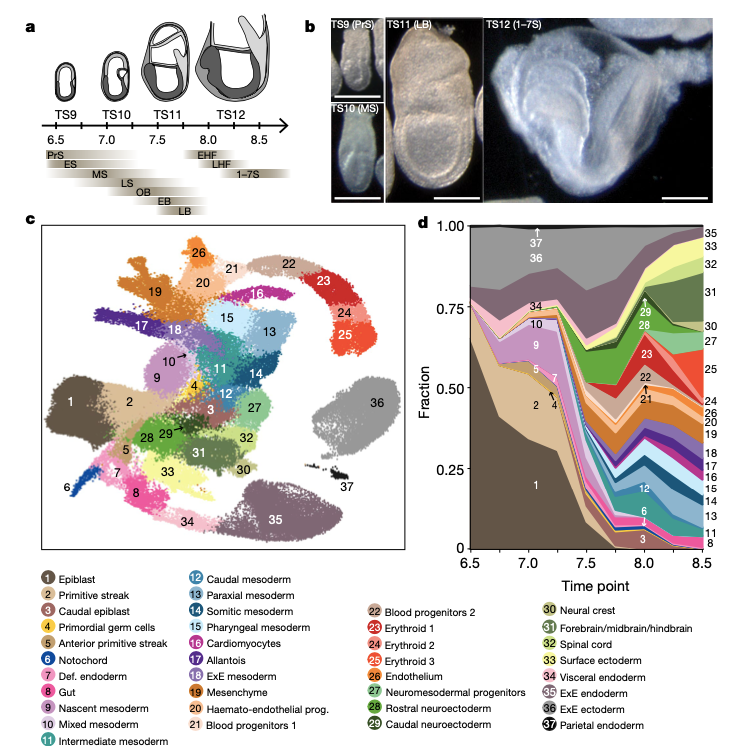
1. Pijuan-Sala B, Griffiths J A, Guibentif C, et al. A single-cell molecular map of mouse gastrulation and early organogenesis[J]. Nature, 2019, 566(7745): 490-495.
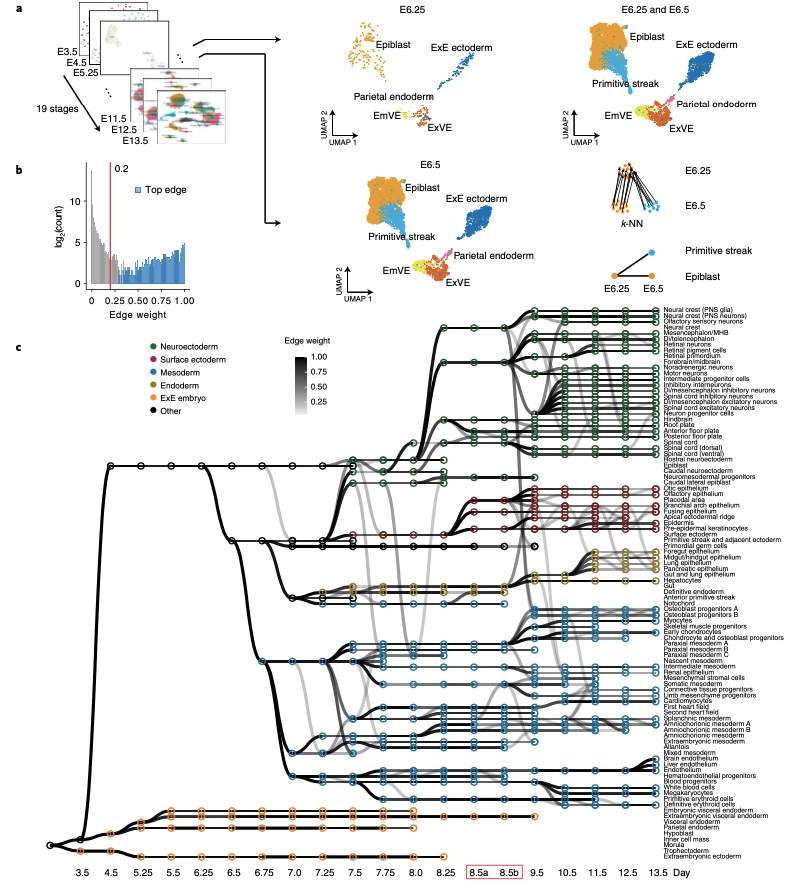
2. Qiu C, Cao J, Martin B K, et al. Systematic reconstruction of cellular trajectories across mouse embryogenesis[J]. Nature genetics, 2022, 54(3): 328-341.
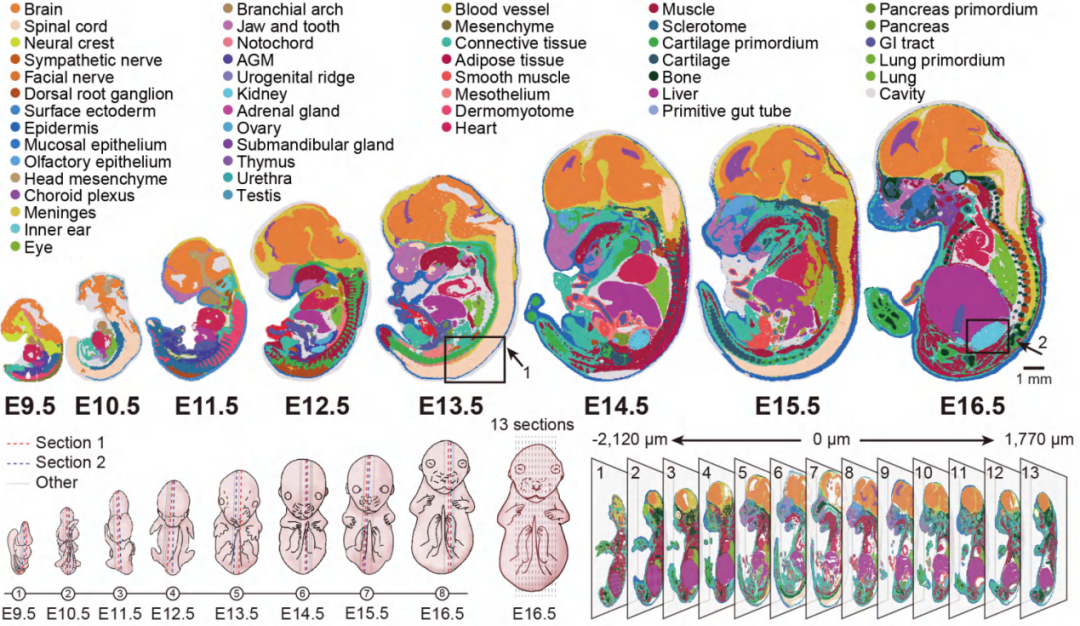
3. Chen A, Liao S, Cheng M, et al. Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays[J]. Cell, 2022, 185(10): 1777-1792. e21.
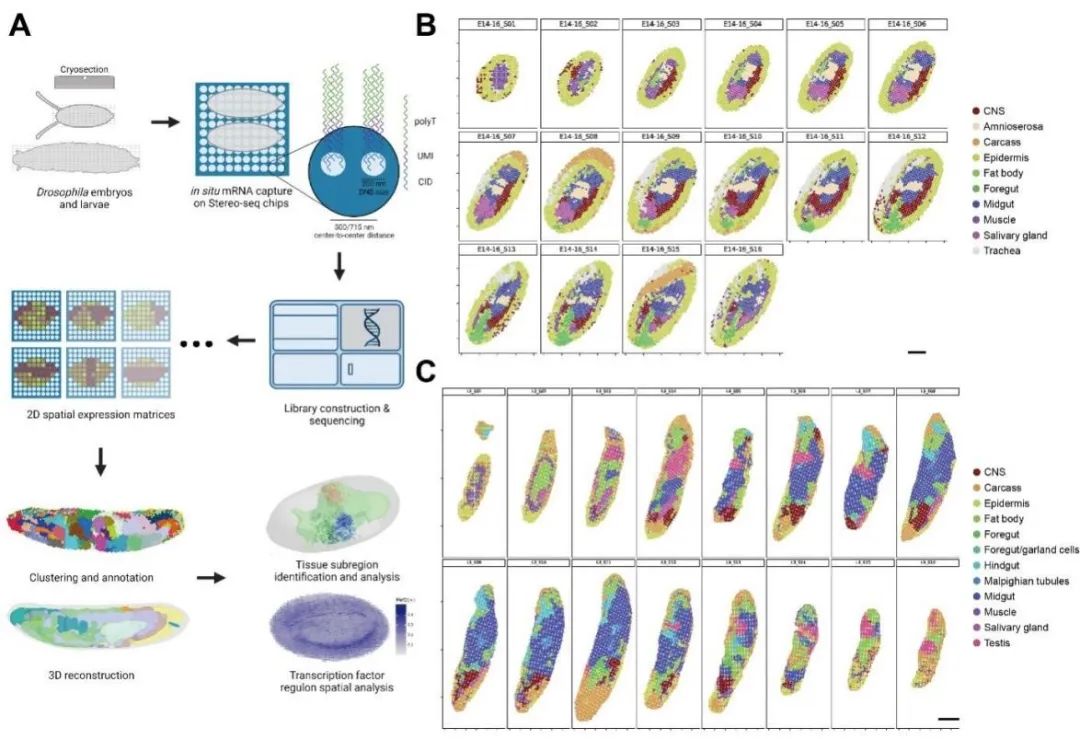
4. Wang M, Hu Q, Lv T, et al. High-resolution 3D spatiotemporal transcriptomic maps of developing Drosophila embryos and larvae[J]. Developmental Cell, 2022, 57(10): 1271-1283. e4.
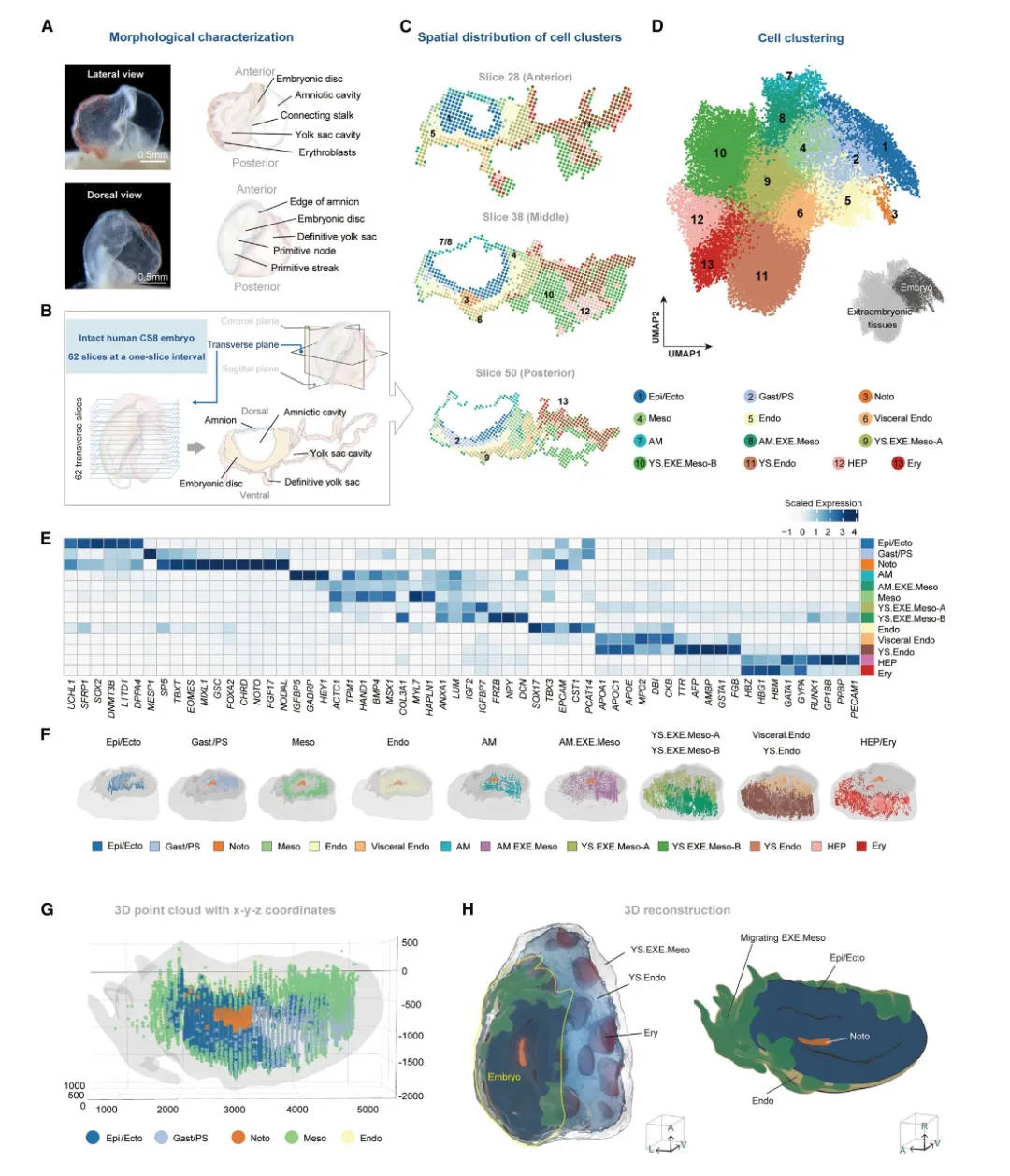
5. Xiao Z, Cui L, Yuan Y, et al. 3D reconstruction of a gastrulating human embryo[J]. Cell, 2024, 187(11): 2855-2874. e19.
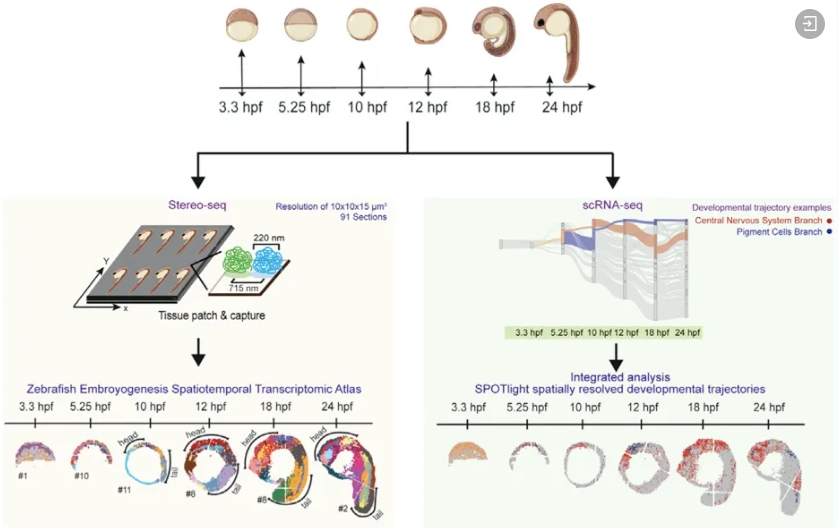
6. Liu C, Li R, Li Y, et al. Spatiotemporal mapping of gene expression landscapes and developmental trajectories during zebrafish embryogenesis[J]. Developmental Cell, 2022, 57(10): 1284-1298. e5.
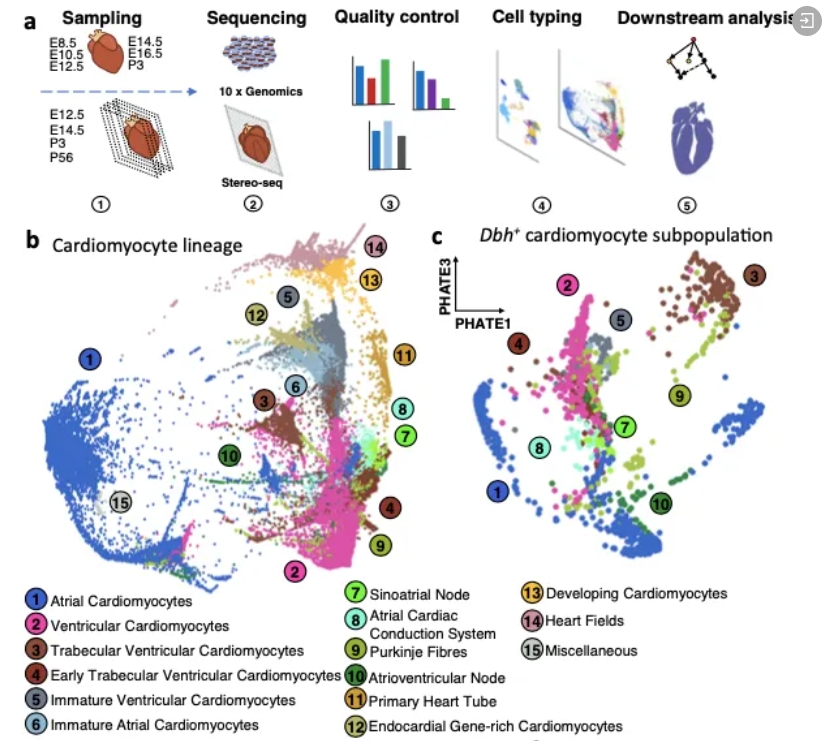
7. Sun T, Grassam-Rowe A, Pu Z, et al. Dbh+ catecholaminergic cardiomyocytes contribute to the structure and function of the cardiac conduction system in murine heart[J]. Nature communications, 2023, 14(1): 7801.