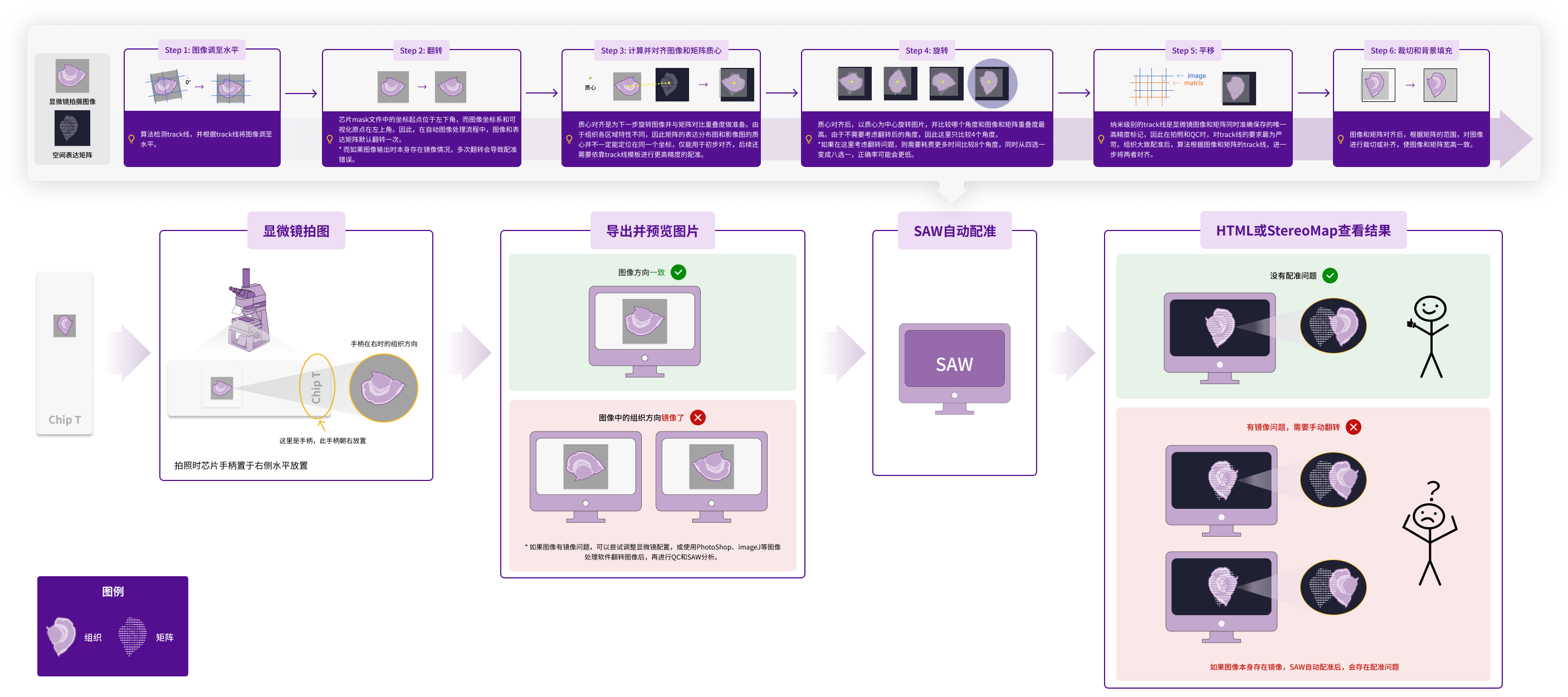
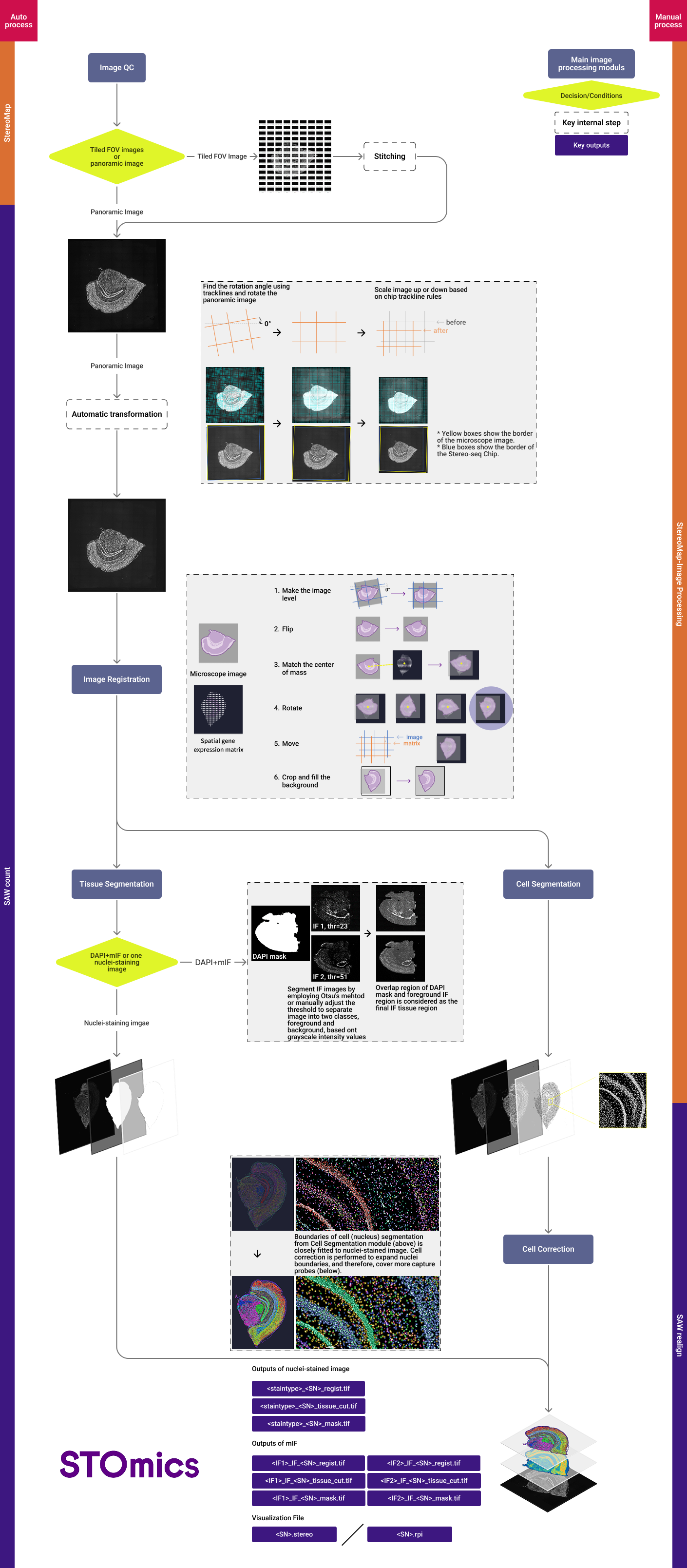
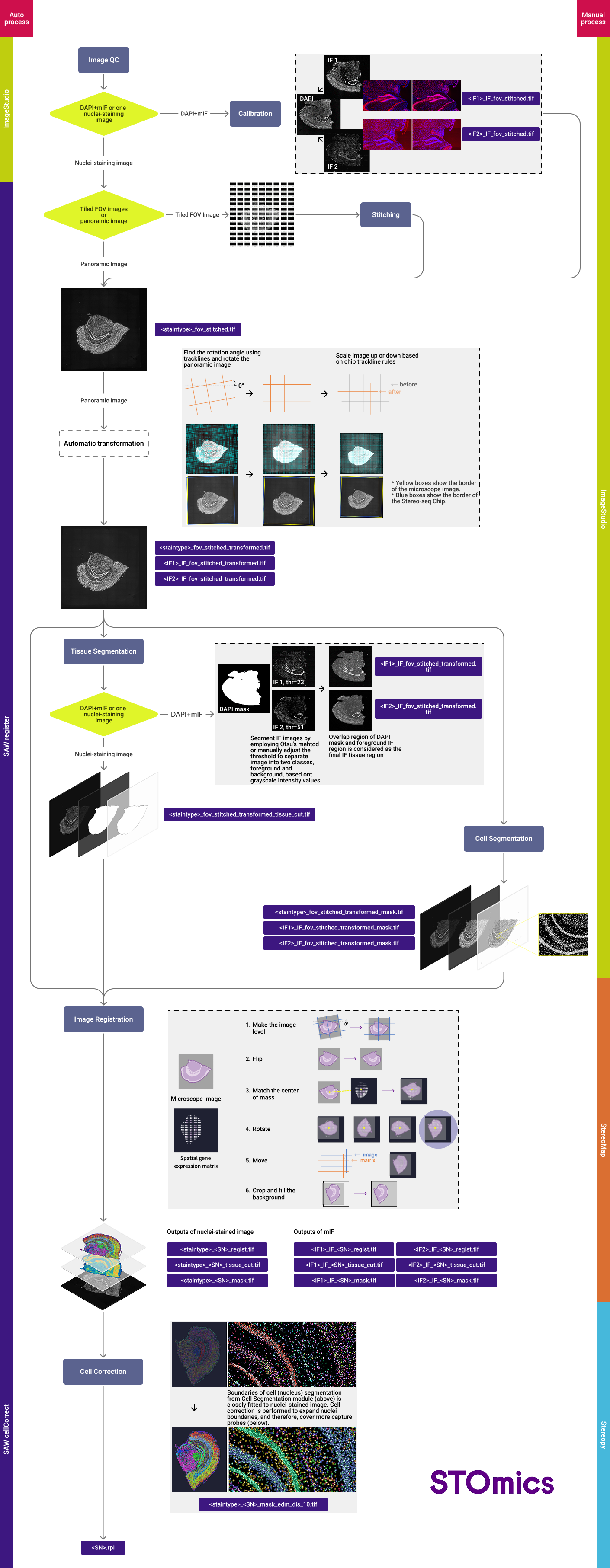
- 显微镜的物镜标识的放大倍率由于各种原因,可能和实际倍率有出入,导致显微镜成像的图不是真实的10x。而StereoMap 小工具模块的图像 QC 算法在倍率差异过大的时候会更容易出现误判或错误检测,因此出现类似问题的时候需要检查显微镜相机的实际放大倍率情况是否符合要求。
- StereoMap 小工具模块的图像 QC 算法可接受的实际放大倍率的范围为:[0.28 ~1 ] 微米/像素(µm/pixel)。通过以下步骤,获取必要信息。
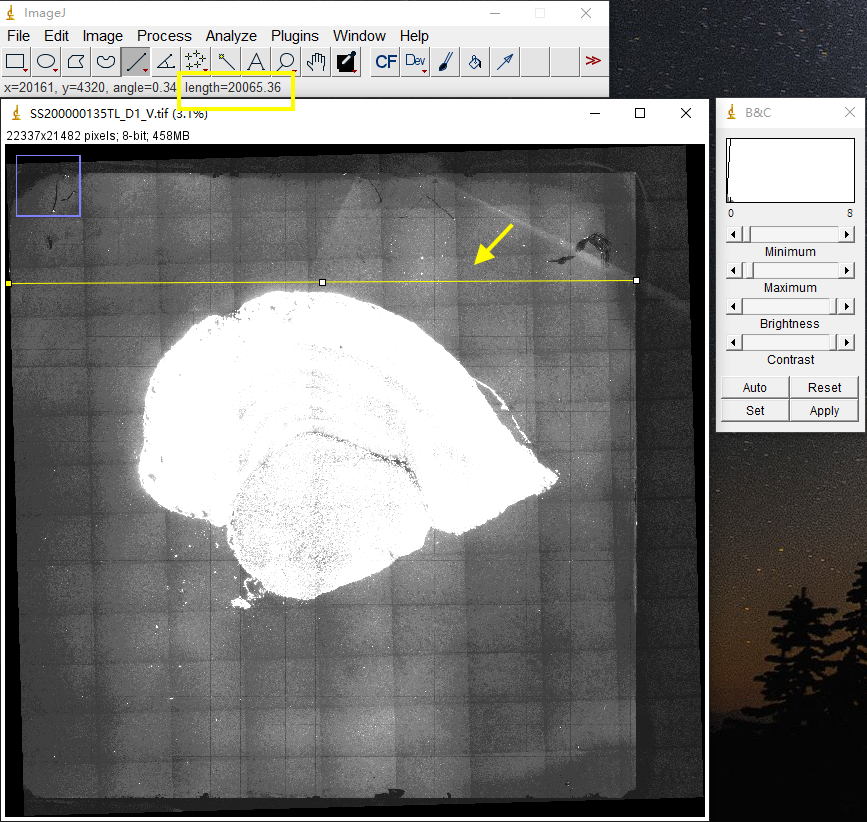
- 手动获取。拍摄一个芯片,已知芯片的规格为1 cm * 1 cm,使用imageJ选择芯片的宽度获得图像中的芯片pixel数,理论宽度 (µm) / 实际测量宽度 (pixel),得到放大倍率。
如果您的实际放大倍率不在算法可以接受的范围内:建议首先确认下选择的物镜放大倍率是否是 10X,其次可以跟显微镜工程师联系做调整。
- 理论上一个S1芯片的宽度为1 cm。如示意,实际测量芯片宽度为20065.36 pixel,实际放大倍率Scale = 1 cm / 20065.36 pixel = 10,000 µm / 20065.36 pixel = 0.498 µm/pixel。
- 0.498 µm/pixel在QC算法可接受的实际放大倍率Scale[0.28 ~1 ] 微米/像素(µm/pixel)范围内。
全部
搜产品
搜资源
搜新闻
搜问答
搜索


.png)