2024年6月21日,新加坡科学技术研究局(A*STAR)和杜克-新加坡国立大学医学院研究员陈金妙课题组在Nature Methods发表了一篇研究论文,题为“Deciphering spatial domains from spatial multi-omics with SpatialGlue”。该研究基于华大生命科学研究院研发的Stereo-CITE-seq技术,对空间多组学数据进行了深入分析。
研究团队开发了一种名为SpatialGlue的新方法,该方法基于图神经网络,通过双重注意力机制,首次成功整合了小鼠脾脏、胸腺和大脑的空间多组学数据。研究不仅揭示了组织内部复杂的细胞组成和功能结构,还通过分析健康和病理模型,揭示了多种组学数据之间的关键差异。这一突破性研究为空间多组学数据的精准整合和分析提供了新的思路和工具。

文章题目:Deciphering spatial domains from spatial multi-omics with SpatialGlue
发表时间:2024-06-21
发表期刊:Nature Methods
主要研究团队:新加坡科学技术研究局(A*STAR)、杜克-新加坡国立大学医学院等
影响因子:36.100
DOI:10.1038/s41592-024-02316-4
空间转录组学作为继单细胞转录组学之后的一项重大技术革新,在分析生物样本方面取得了显著进展。当前,这一技术正逐步向空间多组学领域拓展,实现了在同一个组织切片上同时分析不同组学的功能。空间技术主要可以划分为测序技术和成像技术两大类别。测序技术包括DBiT-seq、spatial-CITE-seq、spatial ATAC–RNA-seq、CUT&Tag-RNA-seq、SPOTS、SM-Omics、Stereo-CITE-seq、spatial RNA-TCR-seq和10x Genomics Xenium等;成像技术包括DNA seqFISH+、DNA-MERFISH-based DNA和RNA profiling、MERSCOPE和Nanostring CosMx等。通过这些技术,研究人员可以在空间背景下获取每个细胞的多种互补视图,为深入了解细胞和组织特性提供了前所未有的机遇。
为了充分利用空间多组学数据构建组织的完整图景,需要对空间感知的异质数据模态进行整合。然而整合来自不同组学的数据面临几个关键挑战。首先,不同组学(如蛋白质和转录本)之间的特征数量可能差异极大,而且它们的统计分布也各不相同。更复杂的是,当尝试将这些组学数据与空间信息相结合时,这些挑战变得更加棘手。当前市面上几乎没有专门用于整合从同一组织切片中获取的空间多组学数据的工具。现有的方法要么只关注单一组学,要么在整合过程中忽视了空间信息。虽然有一个名为MEFISTO的工具声称能整合空间多组学数据,但它之前主要被用于单细胞多组学或空间转录组学,尚未广泛适用于更复杂的场景。
对于非空间的多组学数据整合,虽然有一些算法如Seurat WNN、MOFA+、StabMap、totalVI、MultiVI和scMM等可供选择,但它们要么只适用于特定类型的组学数据,要么简单地假设所有组学的数据特征都同等重要,这在实际情况中并不总是成立。因此,需要更先进的方法来更准确地整合空间多组学数据,以便更深入地理解组织的生物学特性。鉴于上述原因,开发一套专门针对空间多组学数据整合的工具显得尤为重要,这将有助于我们应对空间多组学数据在下游分析中的需求与挑战。
SpatialGlue是一款专门用于空间多组学分析的方法。它能够将来自同一组织切片的多种空间组学数据模态进行有效整合,进而以更高的空间分辨率揭示组织样本的空间域。SpatialGlue运用图神经网络技术,为每个数据模态学习低维嵌入,随后实现跨模态数据的精准整合。为了更准确地整合空间信息与个体组学数据,并促进跨组学的综合分析,SpatialGlue特别引入了双重注意力聚合机制,该机制能够自适应地评估不同数据模态的重要性,从而极大地提升了整合的准确性和效率。
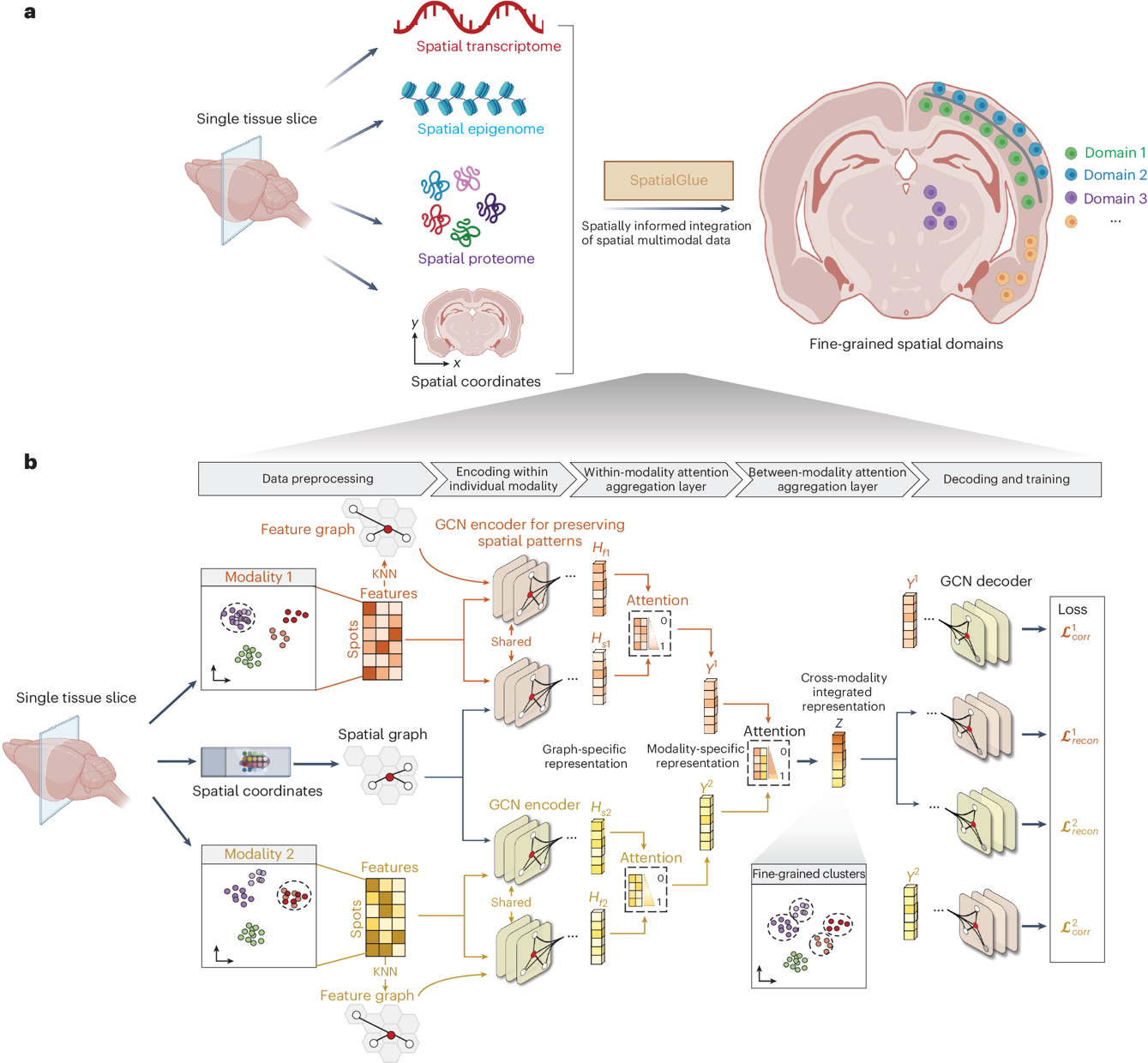
SpatialGlue -用于空间多组学数据分析的可解释深度双重注意模型
首先,研究人员在模拟数据和实验获取的真实数据上测试了SpatialGlue的性能,对于人类淋巴结数据,作者使用空间转录组技术和蛋白质共谱技术生成淋巴结数据集。结果显示,SpatialGlue在定量性能和捕捉解剖细节方面均优于现有方法。这表明SpatialGlue能够更准确地整合多种数据模态,并提供更精细的组织结构信息。
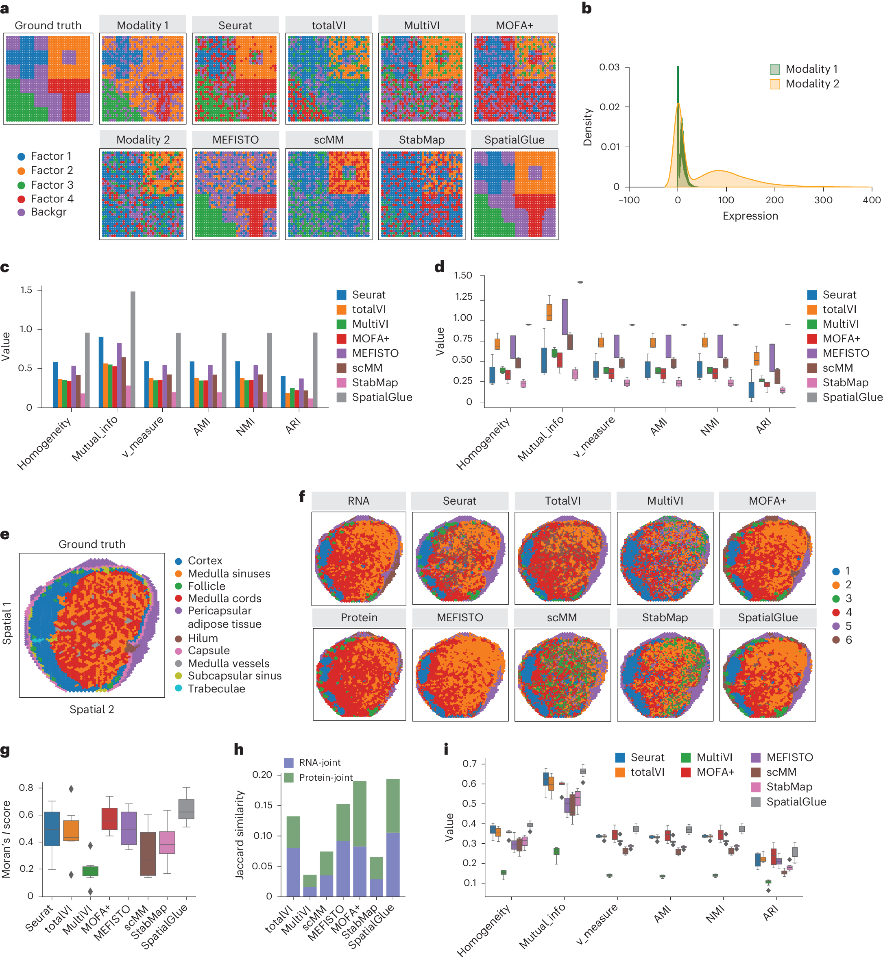
SpatialGlue在模拟和真实数据中准确识别空间域
其次,研究人员在小鼠大脑样本中应用了SpatialGlue,成功整合了空间表观基因组和转录组数据。鼠脑数据共分为两类,一类是ATAC–RNA-seq,目的是测量mRNA和开放染色质区域;另一个是RNA-seq与CUT&Tag模式。通过这一整合方法,研究人员能够识别和区分更多的皮层层次,比原始数据注释更为详细。这一发现揭示了大脑结构的复杂性,并展示了SpatialGlue在解析复杂组织样本方面的潜力。
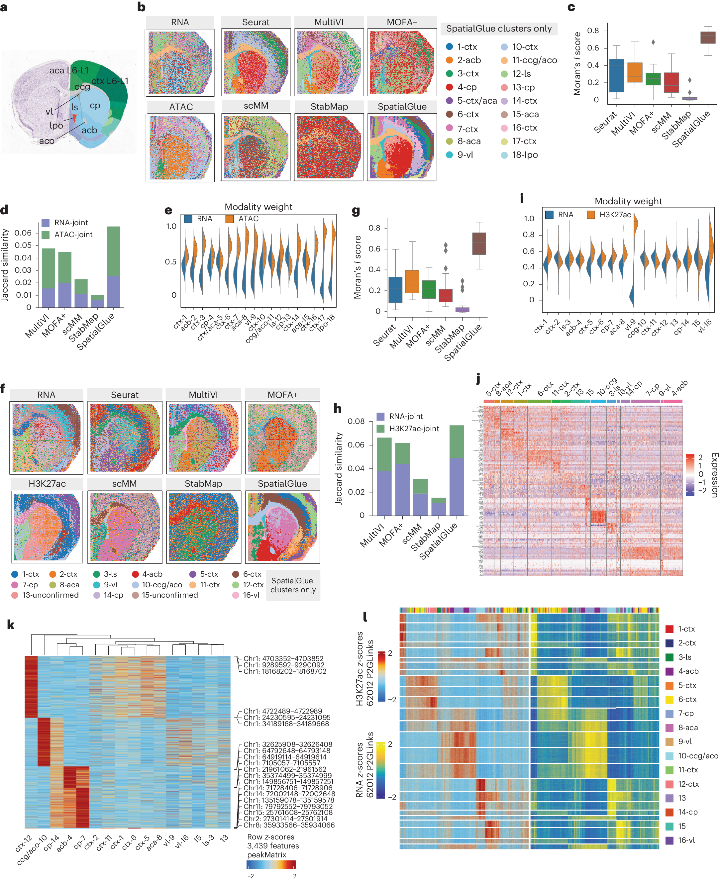
SpatialGlue 以更高的分辨率剖析了小鼠大脑样本的空间表观基因组-转录组数据
此外,研究团队展示了SpatialGlue在各技术平台上具有广泛适用性,并进一步将其应用于Stereo-CITE-seq和SPOTS获取的数据。Stereo-CITE-seq被用于分析小鼠胸腺切片,在亚细胞分辨率下捕获mRNA和蛋白质,并准确识别出髓质和皮质区域,其性能优于其他七种方法。研究还发现SpatialGlue能够区分脾脏中的不同巨噬细胞亚群,提供了关于免疫细胞功能和分布的新见解。这一发现展示了SpatialGlue在解读免疫系统复杂性方面的应用前景。
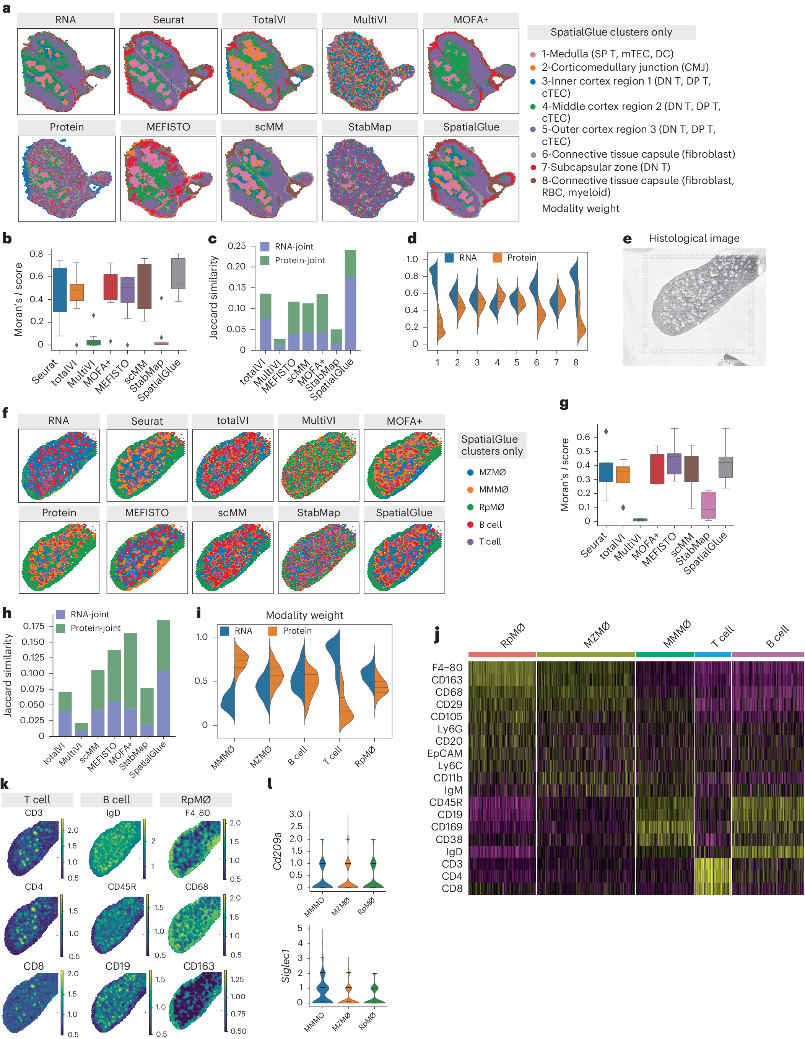
SpatialGlue 准确整合了小鼠胸腺和脾脏的多模态数据
通过上述实验,研究人员发现SpatialGlue具有跨模态数据整合的显著优势。相比于单一模态的数据分析,SpatialGlue能够结合不同组学的数据,更全面地理解组织样本的功能和结构。它不仅揭示了更多的生物学细节,还帮助研究人员识别了潜在的生物机制。
总体而言,SpatialGlue展示了在空间多组学数据整合中的强大能力,为深入研究生物样本的时空特性提供了新的工具和方法。这一研究不仅提升了研究人员对复杂组织样本的理解,也为未来的多组学数据整合和分析提供了重要的参考和借鉴。
陈金妙博士,新加坡科学技术研究局(A*STAR)和杜克-新加坡国立大学医学院研究员。对用于精准免疫学的人工智能驱动的单细胞和空间组学分析感兴趣。其研究团队主要从事人工智能算法和多组学的数据库开发。
内容 | 李睿思、Toh Qin Kane、陈慧
审校 | 翦敏、黎晓玲、赵芳





