可视化指南
可视化入口
从 StereoMap 启动页的 Visual Explore 进入可视化。
导入数据
打开 StereMap 软件的 Visual Explore 模块,选择 .stereo 文件。 (可以参考可视化输入文件获取更多信息).
可视化界面
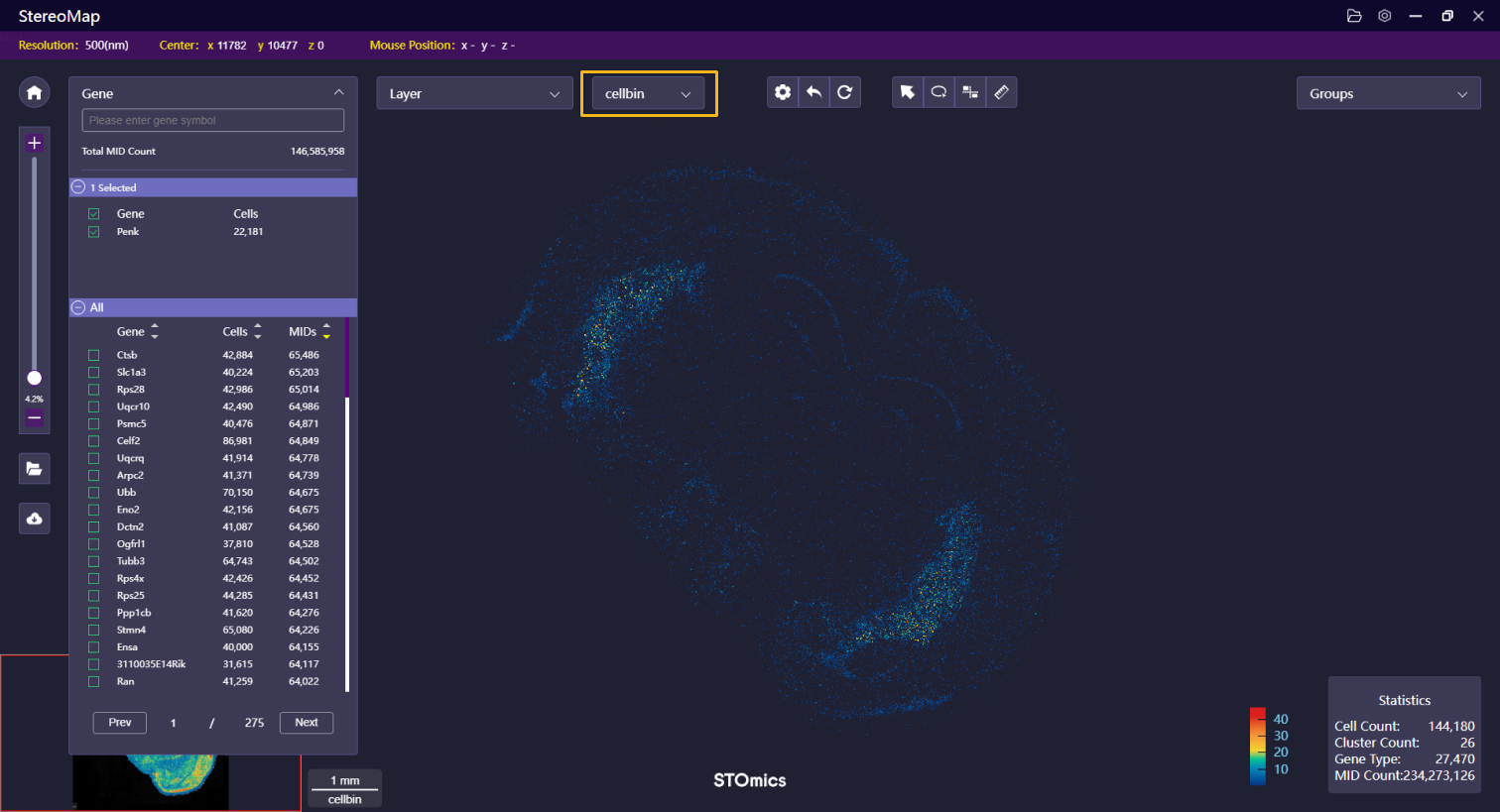
如下图所示,展示了可视化模块的界面布局。
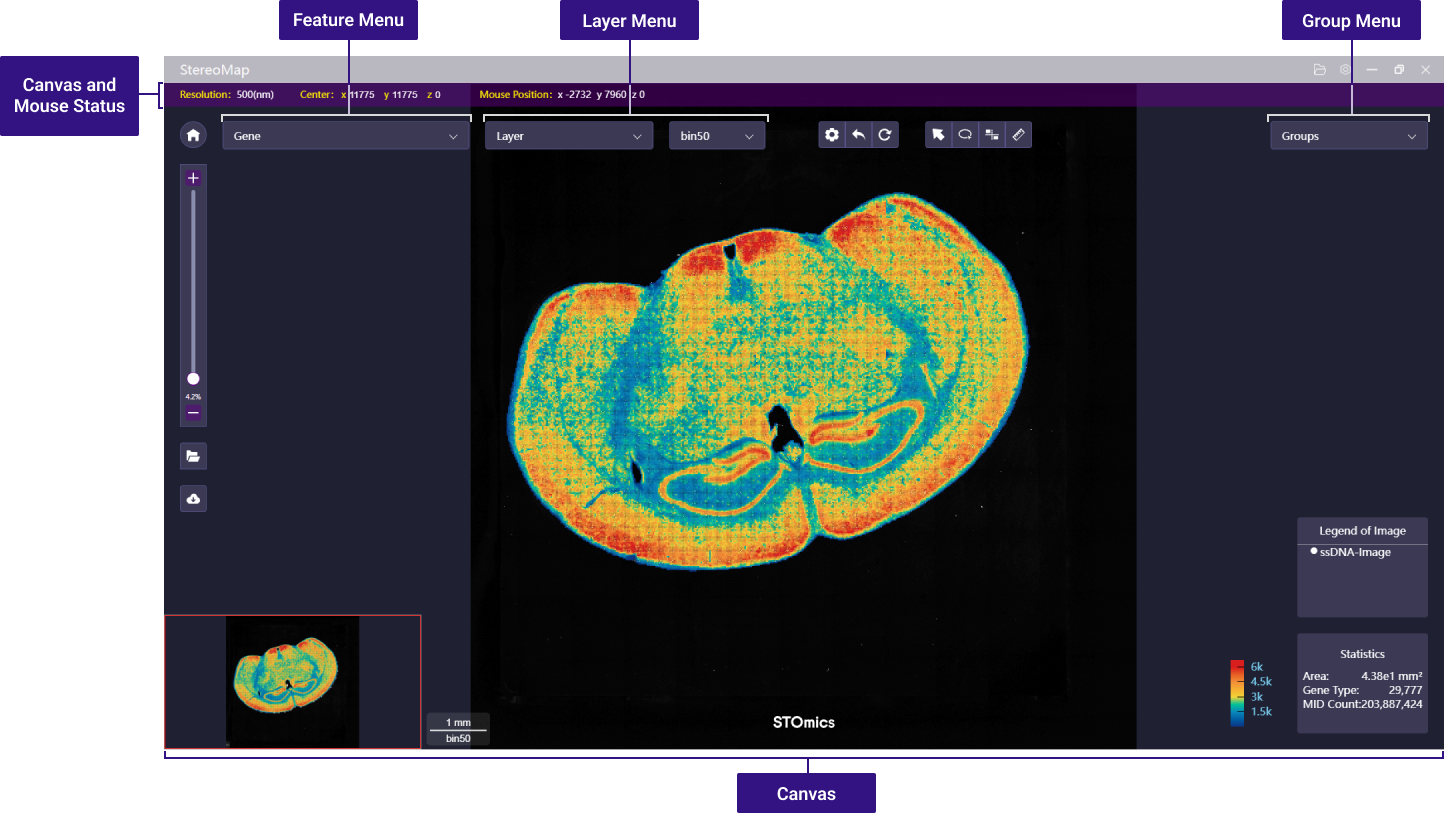
画布
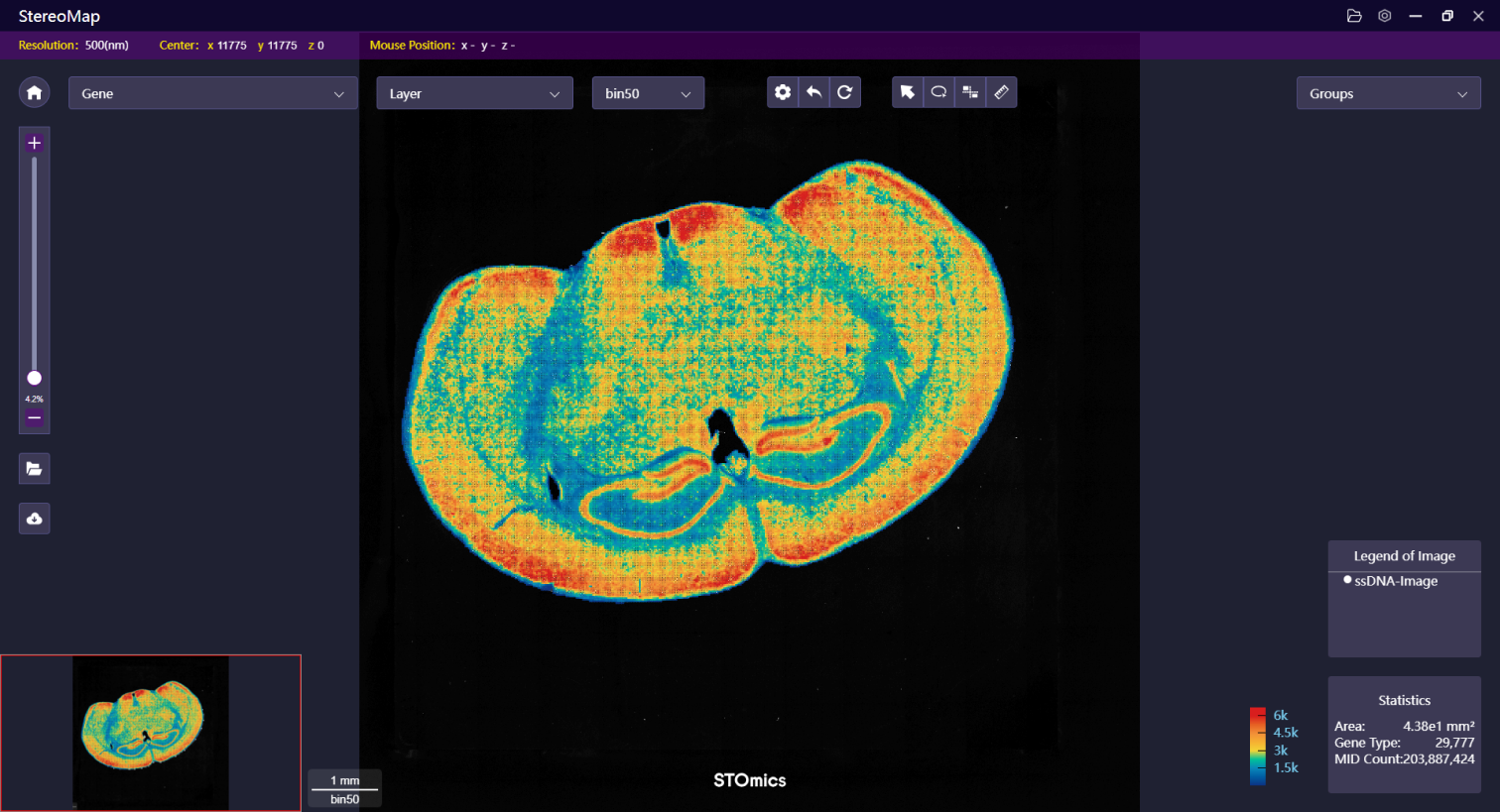
如下画布展示了空间表达矩阵的数据按照点的形式叠加在 ssDNA 图像的组织切片上,其它工具悬浮在画布上。
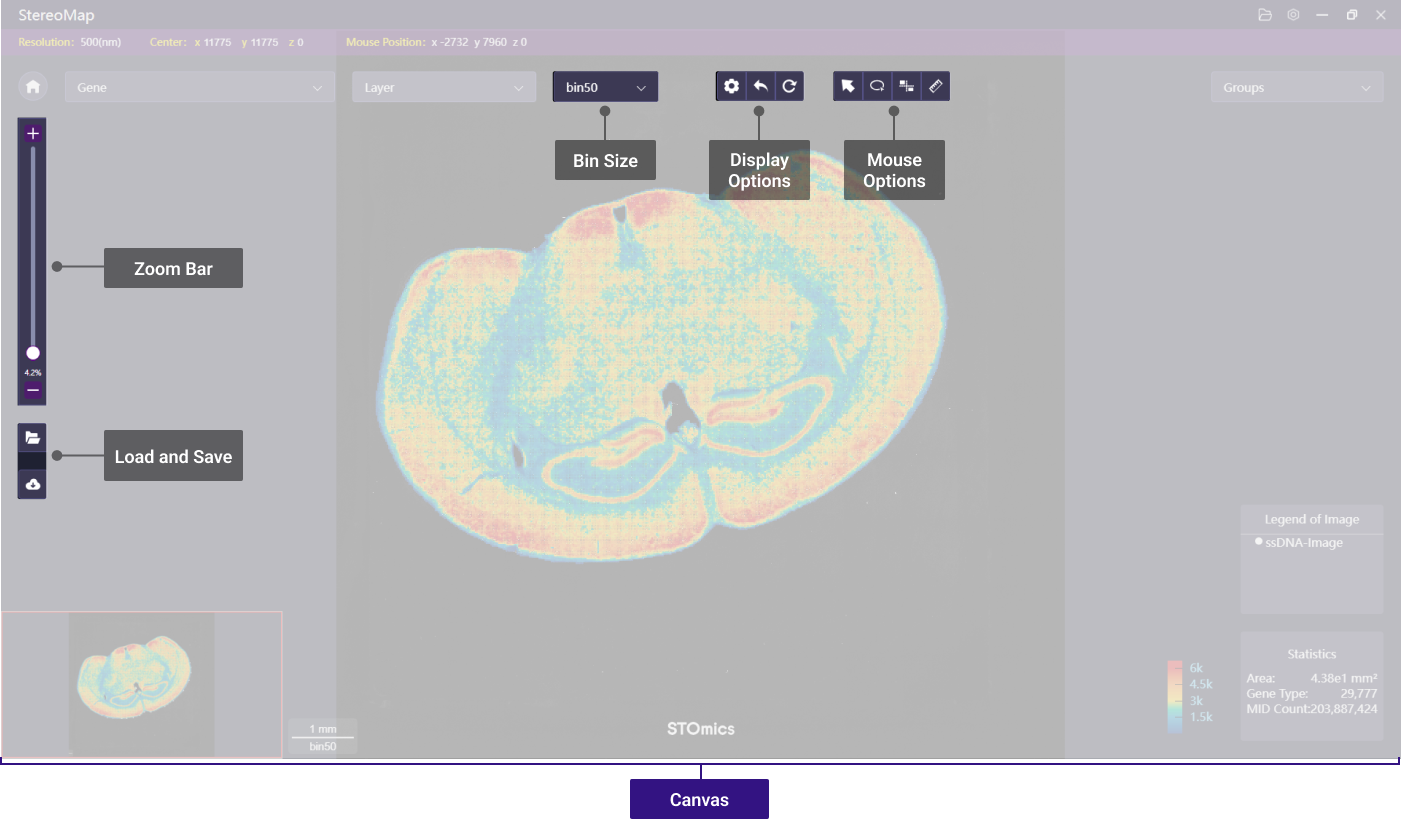
Binsize 选择
点击 Binsize 的下拉列表 为空间表达矩阵的热图选择合适的分辨率进行展示。
为空间表达矩阵的热图选择合适的分辨率进行展示。
显示设置
这三个按钮 主要调整画布的操作信息及组件的显示/隐藏,从左到右的依次说明如下:
主要调整画布的操作信息及组件的显示/隐藏,从左到右的依次说明如下:
操作工具
从左到右依次为:
- 游标:点击图标可退出手动操作的模式。
- 套索:使用套索工具手动绘制感兴趣的 ROI 区域。按住 Ctrl + 鼠标左键并移动鼠标开始圈选,点击 Enter 即可结束一次圈套。如果需要绘制多个区域,请松开鼠标左键,然后每次绘制时再次按住 Ctrl + 鼠标左键并移动鼠标开始圈选。套索功能可与新建 Group 组(参考 Group 菜单获取更多信息)结合使用。可参考标注区域并生成新的热图或基于 H&E 图像的区域标注获取更多信息。
- 配准模板:单击配准模板按钮展示矩阵的 track 线模板,可用于辅助观察图像图层是否和表达矩阵配准。可以参考检查图像的配准获取更多信息。
- 测量:主要是测量两点之间的距离,单位为 PX (像素)。
导入及导出
导入及导出的选项如下:
- 导入:单击导入按钮,可以加载 lasso 或者差异分析的文件。
.png>)
- 选择 Load a Lasso Record 加载
.lasso.geojson文件,可以继续进行套索区域的修改。 - 选择 Load CSV File 加载差异分析的结果文件,将打开一个新的窗口展示 CVS 文件的内容。
- 选择 Load a Lasso Record 加载
- 输入自定义图像的文件名称。
- 选择导出的图像类型:
- Screenshot Image :当前画布的图像,截图的清晰度取决于当前使用电脑的分辨率。
- HD Image:当前屏幕图像的高清图。若开启了图例,则图例会单独作为一张图片保存下来。
- 如果选择了 Export 您的资源管理器将会打开,需要您选择对应图像的路径。
.png)
缩放条
点击缩放条可以放大或者缩小画布的大小。
Feature 菜单
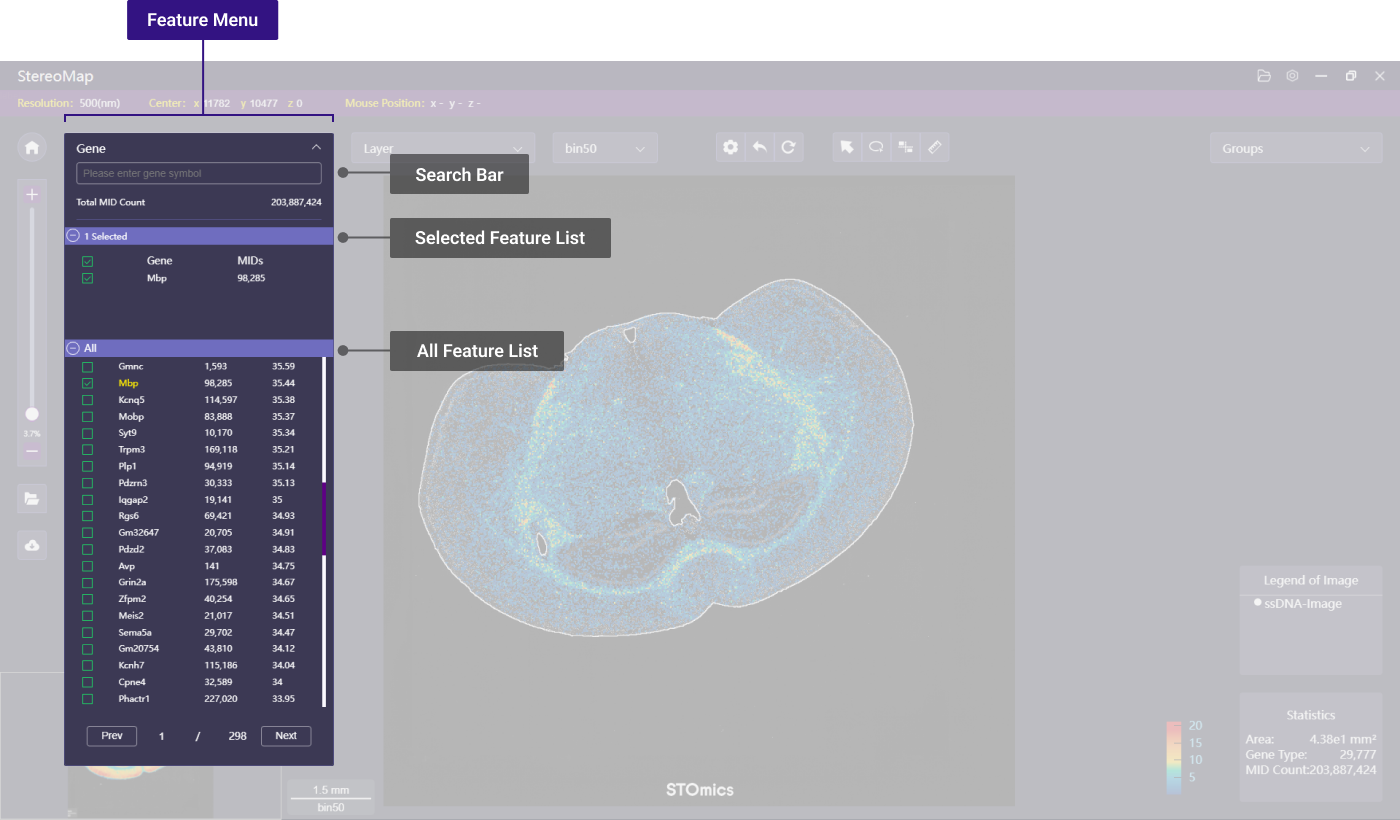
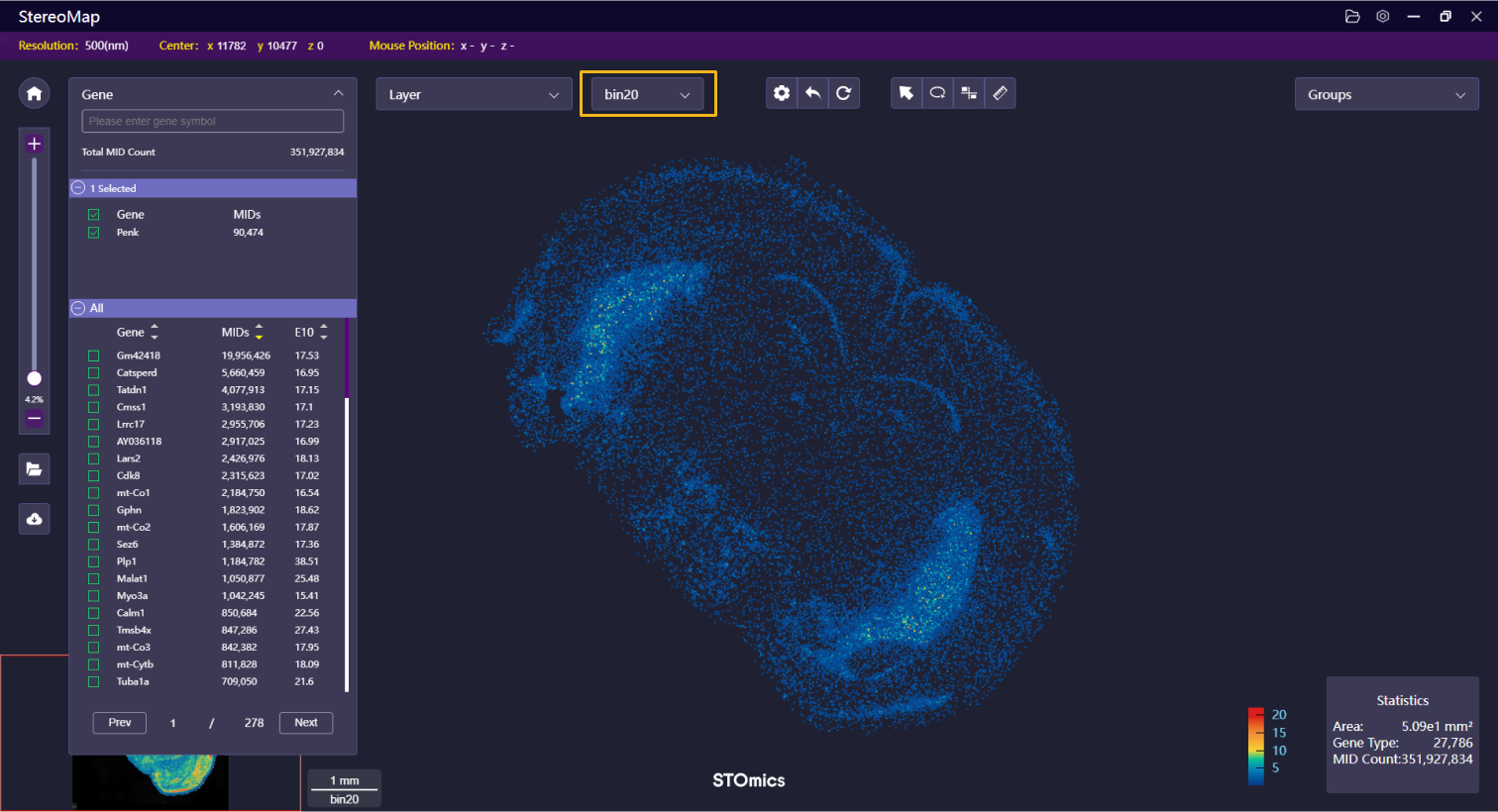
Feature 菜单显示了当前主分析图层基因/蛋白汇总的特征结果,点击菜单栏可以展开/折叠面板。
如果您选择的是 bin5 、bin20 之类的 square bin: 面板中会展示特征(基因/蛋白)名称、MID 总数和 E10 三列信息。面板默认按照 MID 计数降序排列,但您可以点击每个表头右侧的小箭头,按所选列升序或者降序排列。E10 为该特征(基因/蛋白)的空间富集程度打分,高E10表示虽然特征可能在整个组织区域都有表达,但表达显著的部分比较聚集,有特别的表达模式。
而如果您选择的是 cell bin,面板中会展示特征(基因/蛋白)名称、该基因分布的细胞个数和 MID 总数三列信息。
您可以选择感兴趣的特征名称(基因/蛋白)来探索其表达情况。
- 搜索:您可以在搜索栏中输入基因/蛋白的名称,对基因/蛋白进行搜索(不区分大小写,支持模糊或者多名称搜索,多名称搜索需要使用英文逗号隔开)。
- 选择单基因/蛋白:点击某个基因/蛋白,该基因/蛋白对应的热图将会展示在画布中。
- 选择多基因/蛋白:勾选基因/蛋白名称前面的
复选框,可以查看多个蛋白/基因的热图,当然您也可以通过不同的颜色特征探索它们之间的共表达(可参阅基因共表达分析获取更多详情)。
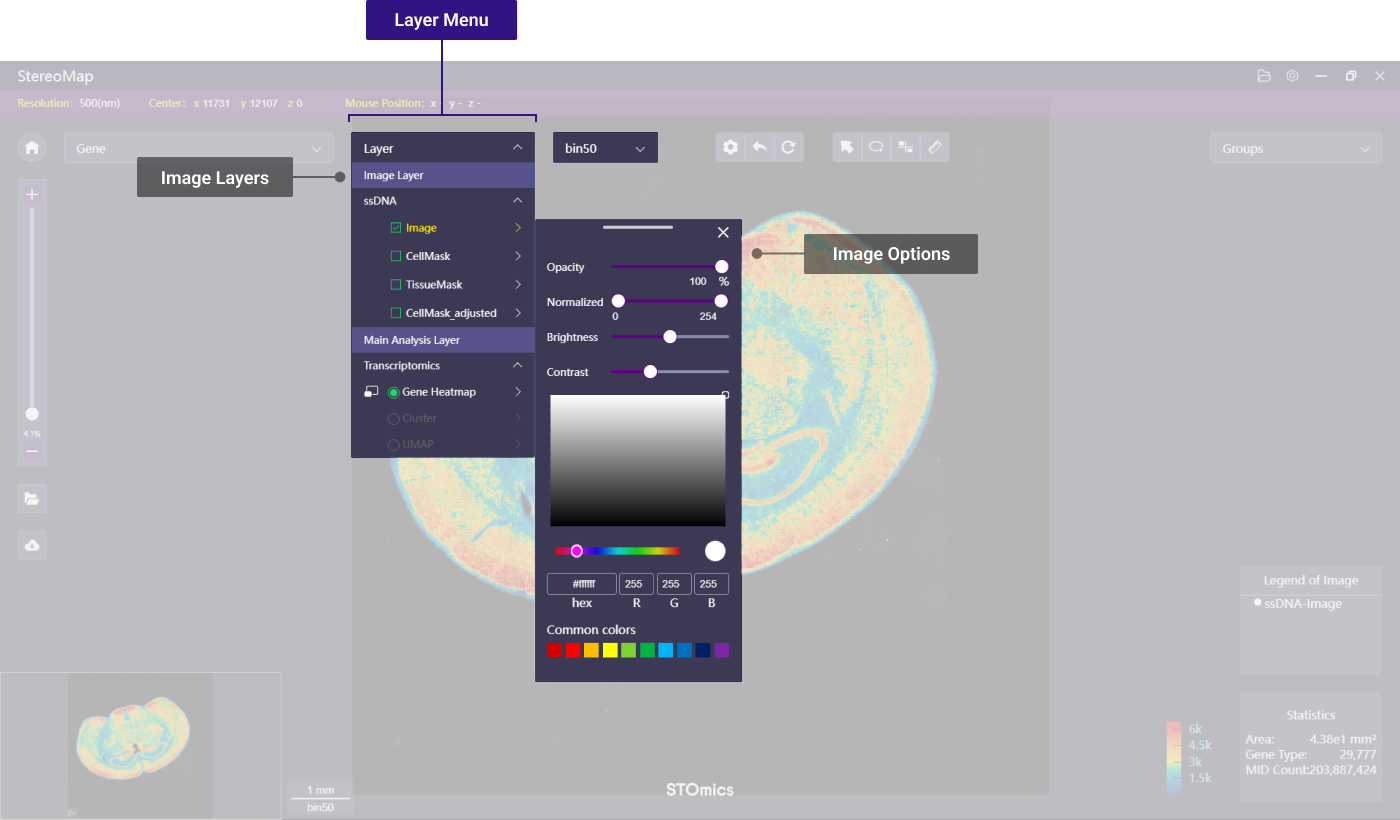
图层菜单及 Binsize 选择
图层菜单主要是控制数据在画布中的展示方式。
图层分为图像图层和主分析图层,binsize 选择提供当前数据的不同分辨率的展示。
图像图层展示的是跟表达矩阵配准后的影像图和分割后的 Mask(包括组织 Mask 和细胞 Mask )。图层参数的调节会依据图像类型的不同存在差异,一般包括透明度、归一化、亮度、对比度和颜色等。归一化调整图像的最大和最小值,有助于可视化展示 track 线。计算公式如下:
- 主分析图层包括表达矩阵的热图、聚类图或者 UMAP 图。聚类图和 UMAP 图只适用于有限的 binsize 图层,如想获取更多信息,请参考主分析图层展示选项。用户单击图层名称前面的图标
 可以打开一个新的链接子窗口,窗口间主要通过坐标联动,如想获取更多信息,可参阅微生物宿主分析获取更多详情 。多窗口联动也可以基于Stereo-CITE数据中预定义的蛋白或者基因分析,可参阅蛋白与其对应标记基因的空间分布获取更多详情。
可以打开一个新的链接子窗口,窗口间主要通过坐标联动,如想获取更多信息,可参阅微生物宿主分析获取更多详情 。多窗口联动也可以基于Stereo-CITE数据中预定义的蛋白或者基因分析,可参阅蛋白与其对应标记基因的空间分布获取更多详情。 - 当前数据的分辨率包括 bin1、bin5、bin10、bin20、bin50、bin100、bin150、bin200 以及cellbin(若该芯片有 cellbin 数据则包含,否则不包含)。Bin1 是每个 bin 中含有 1 个 DNB,cellbin 是基于细胞的维度对 DNB 进行分组,而 Bin5 则表示 5X5 个 DNB 作为一个分组的单元。自动-binsize 开关,如果您开启后,系统会根据画布放大系数自动切换 binsize。
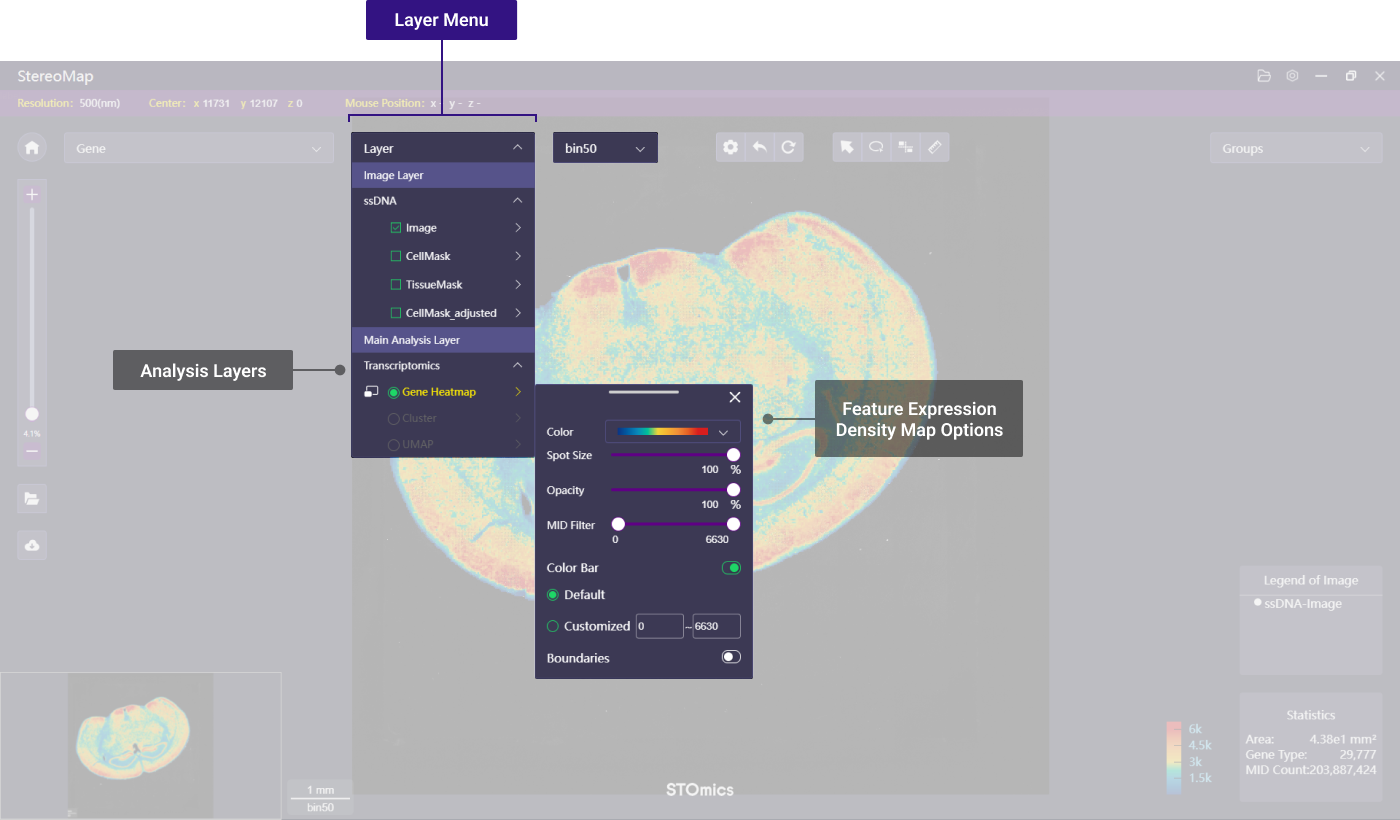
主分析图层展示选项
主分析图层可调整的选项会依据不同的数据或者 bin 类型而不同。
热图图层可调整的选项包括:
| 热图选项 | 说明 | Bin N | Cell Bin |
|---|---|---|---|
| 颜色 | 选择热图的配色方案 | 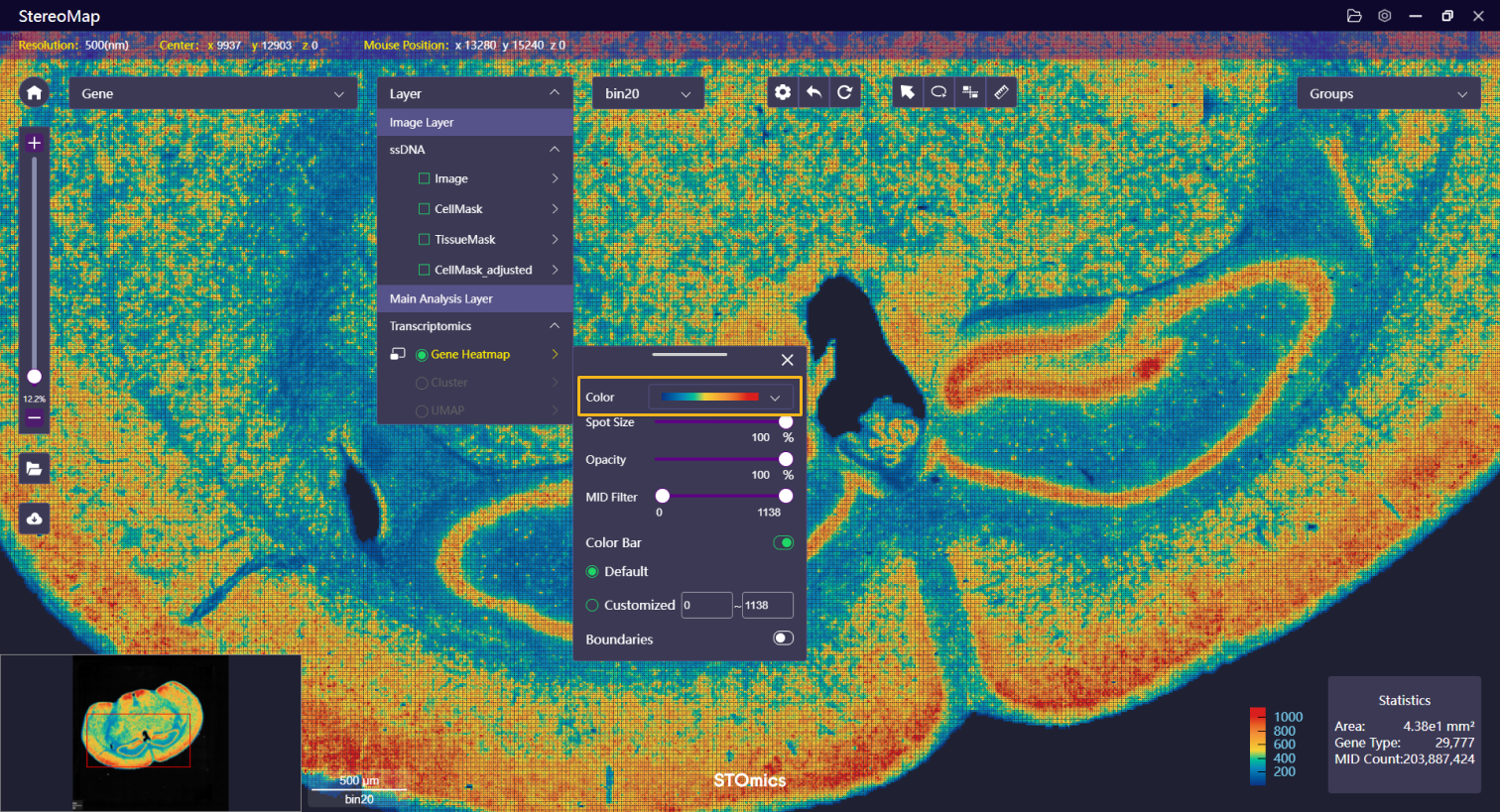 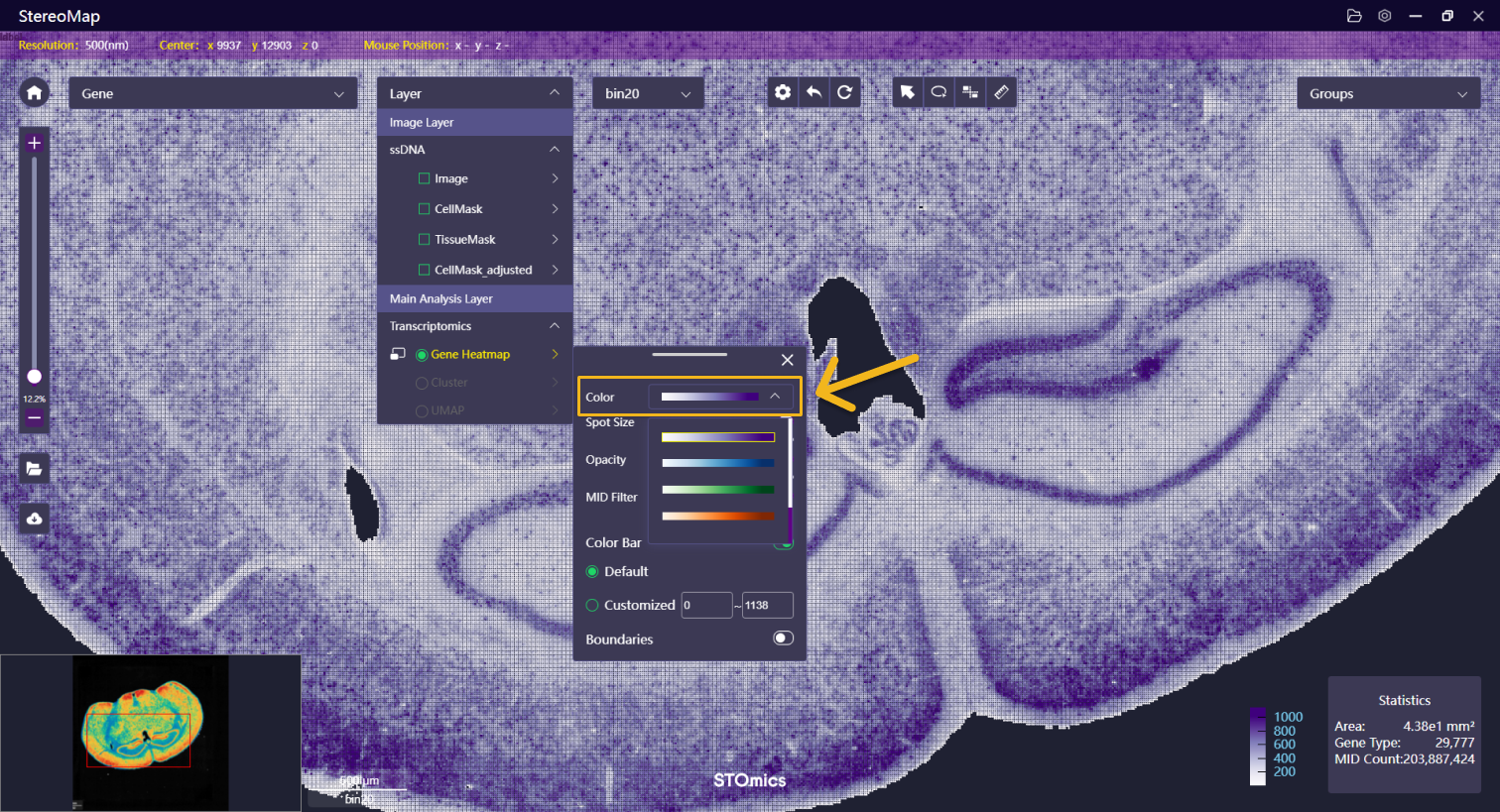 | 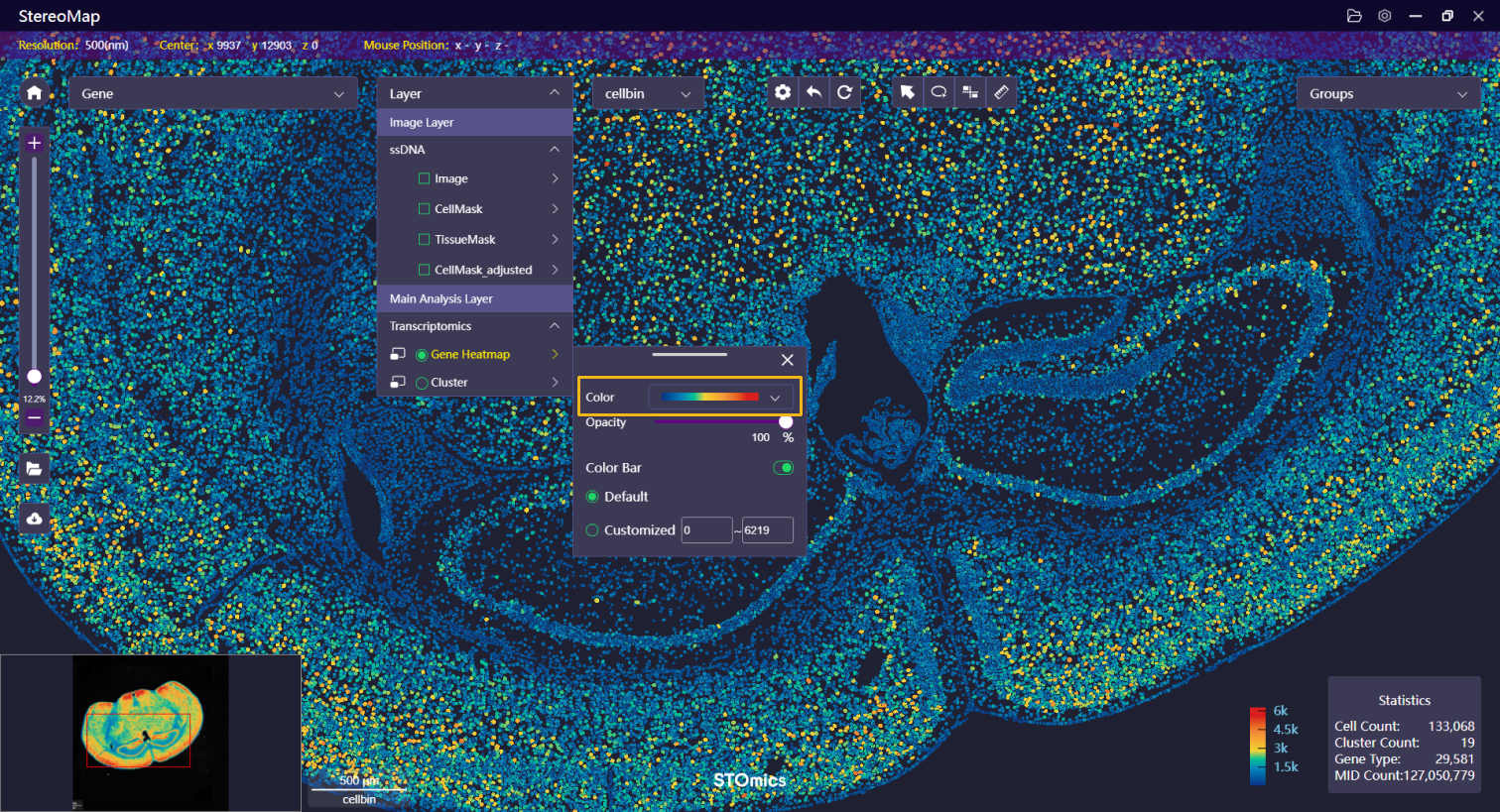 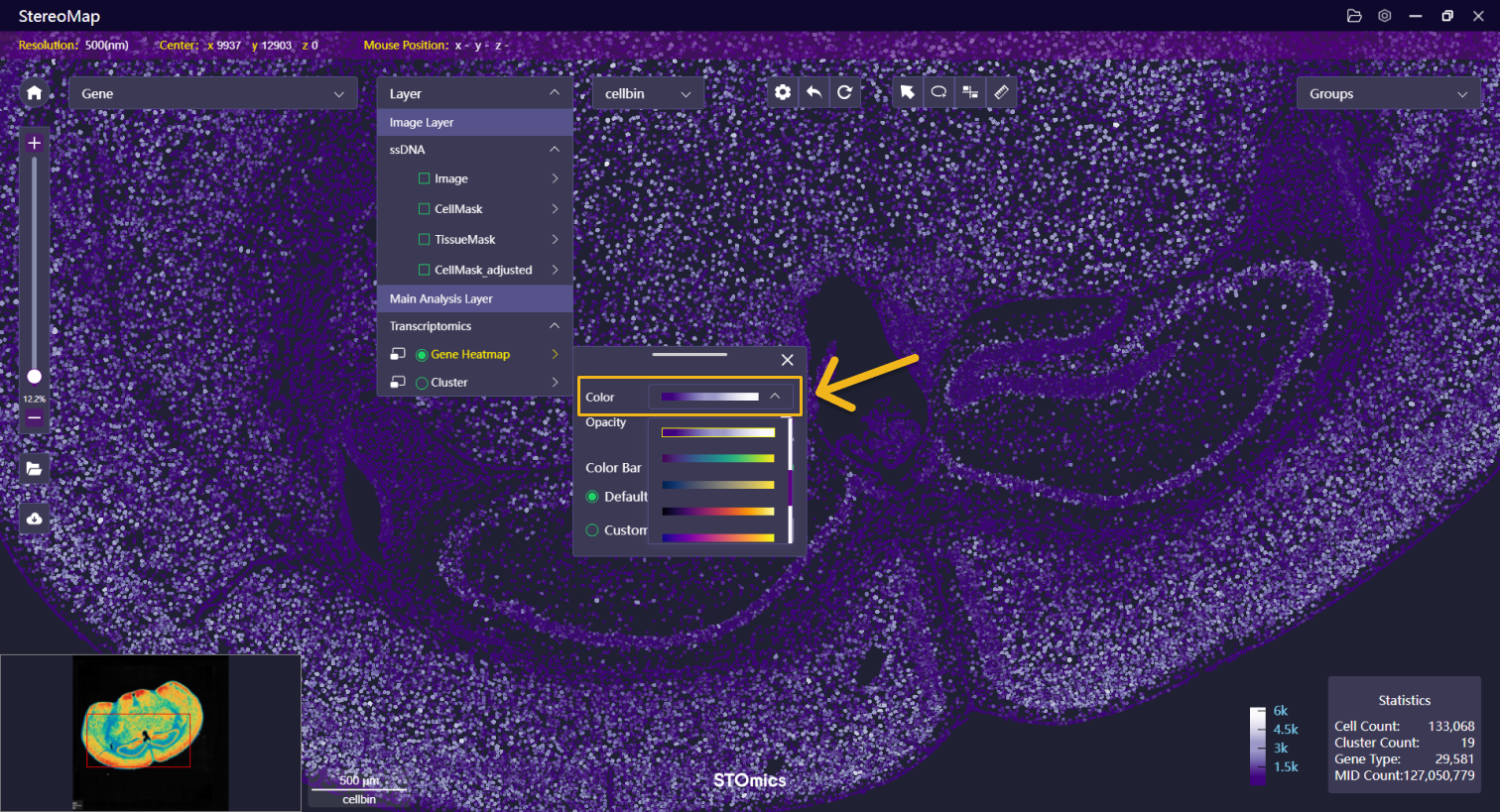 |
| 散点大小 | 调整热图的散点大小。 | 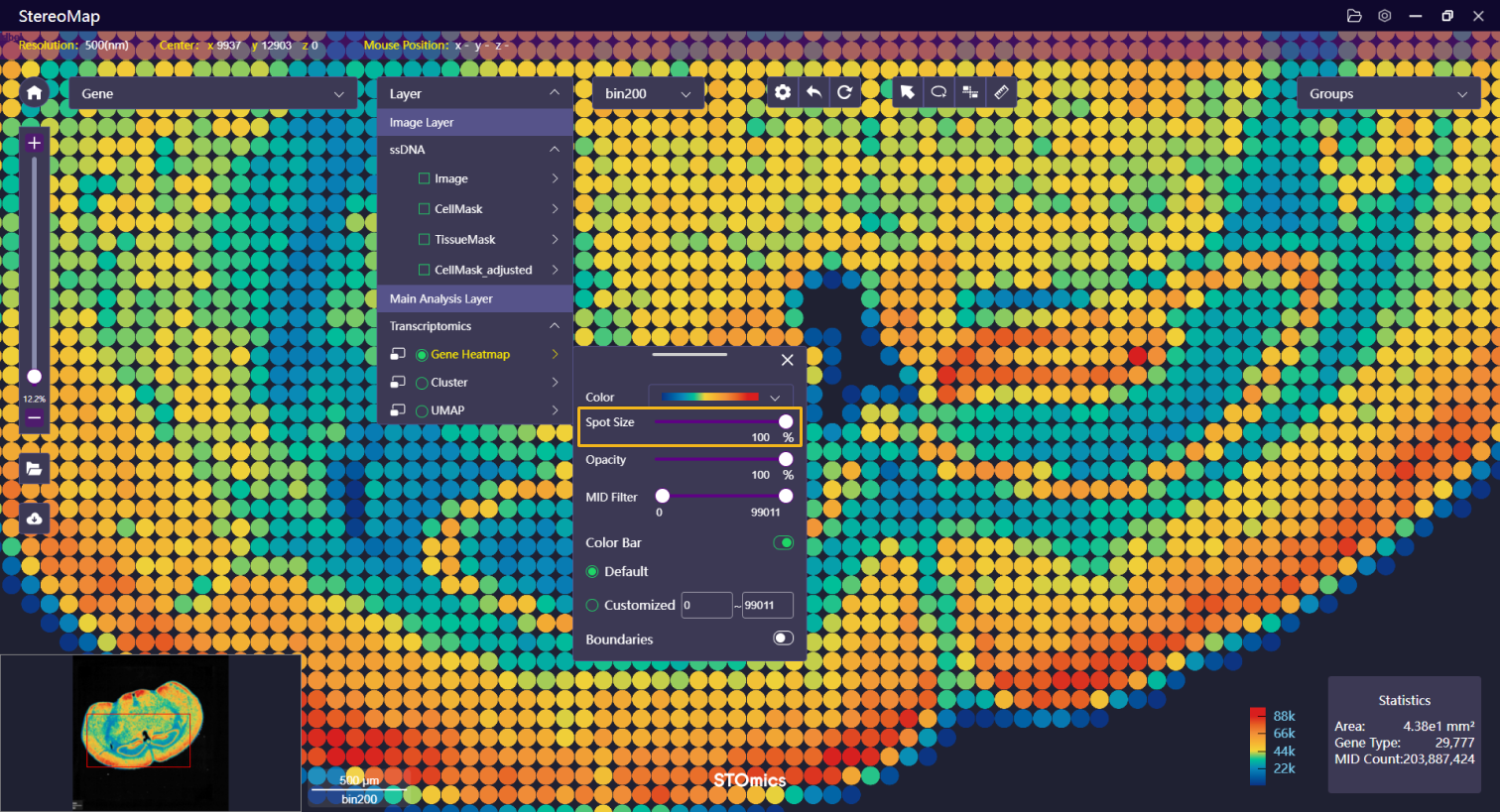 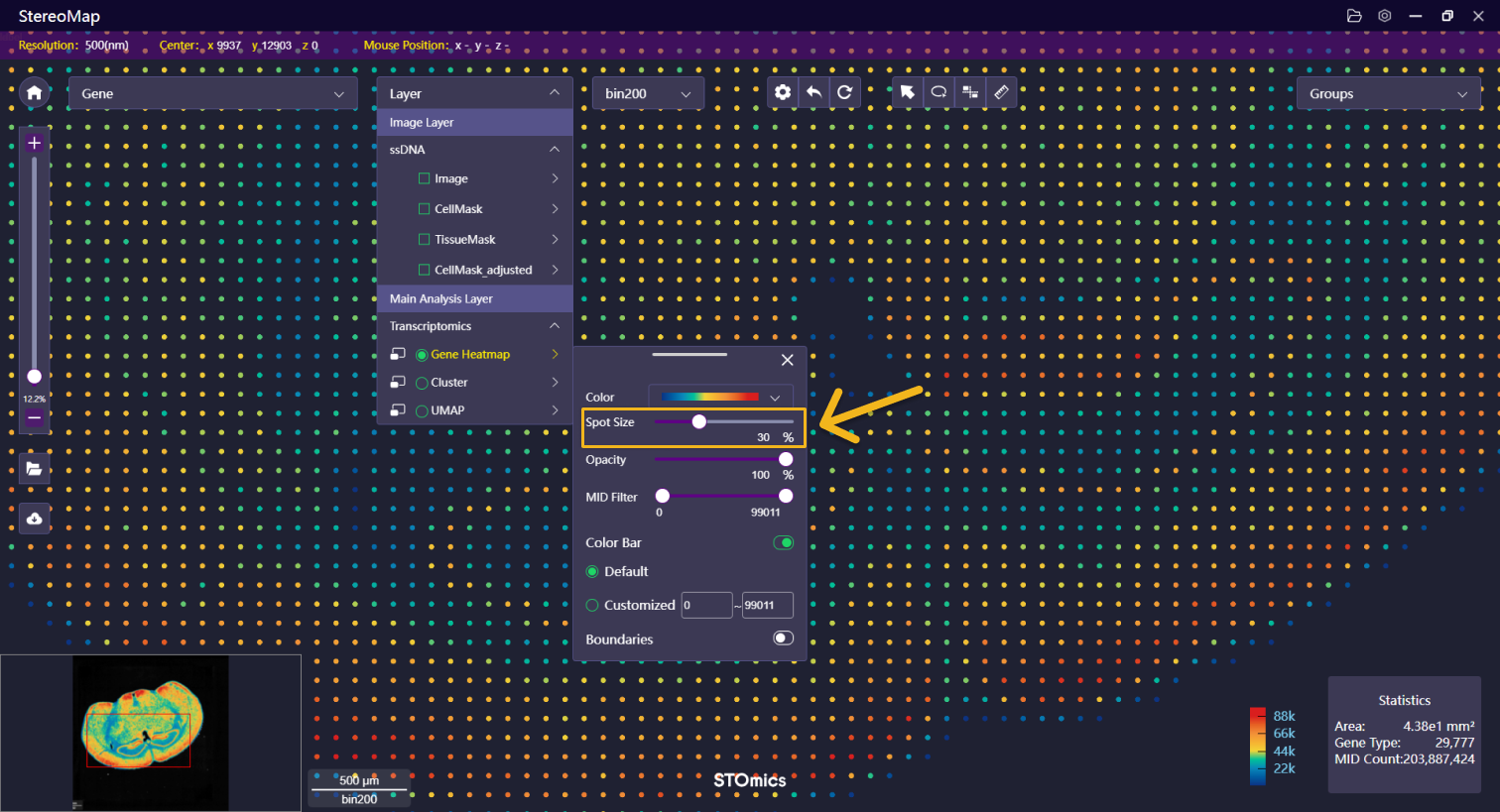 | NA |
| 透明度 | 调整热图的透明度以便更好的可视化展示。 | 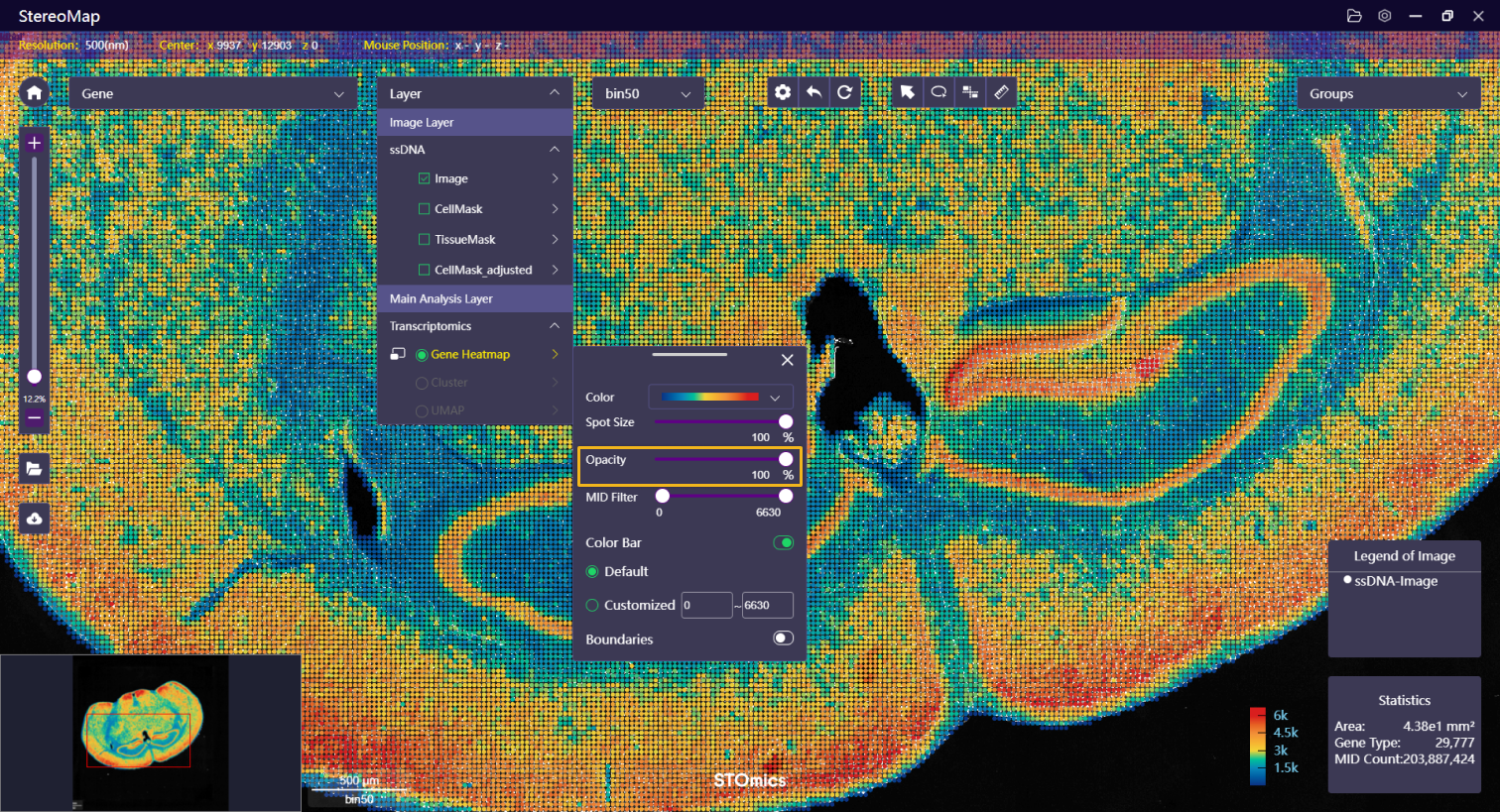 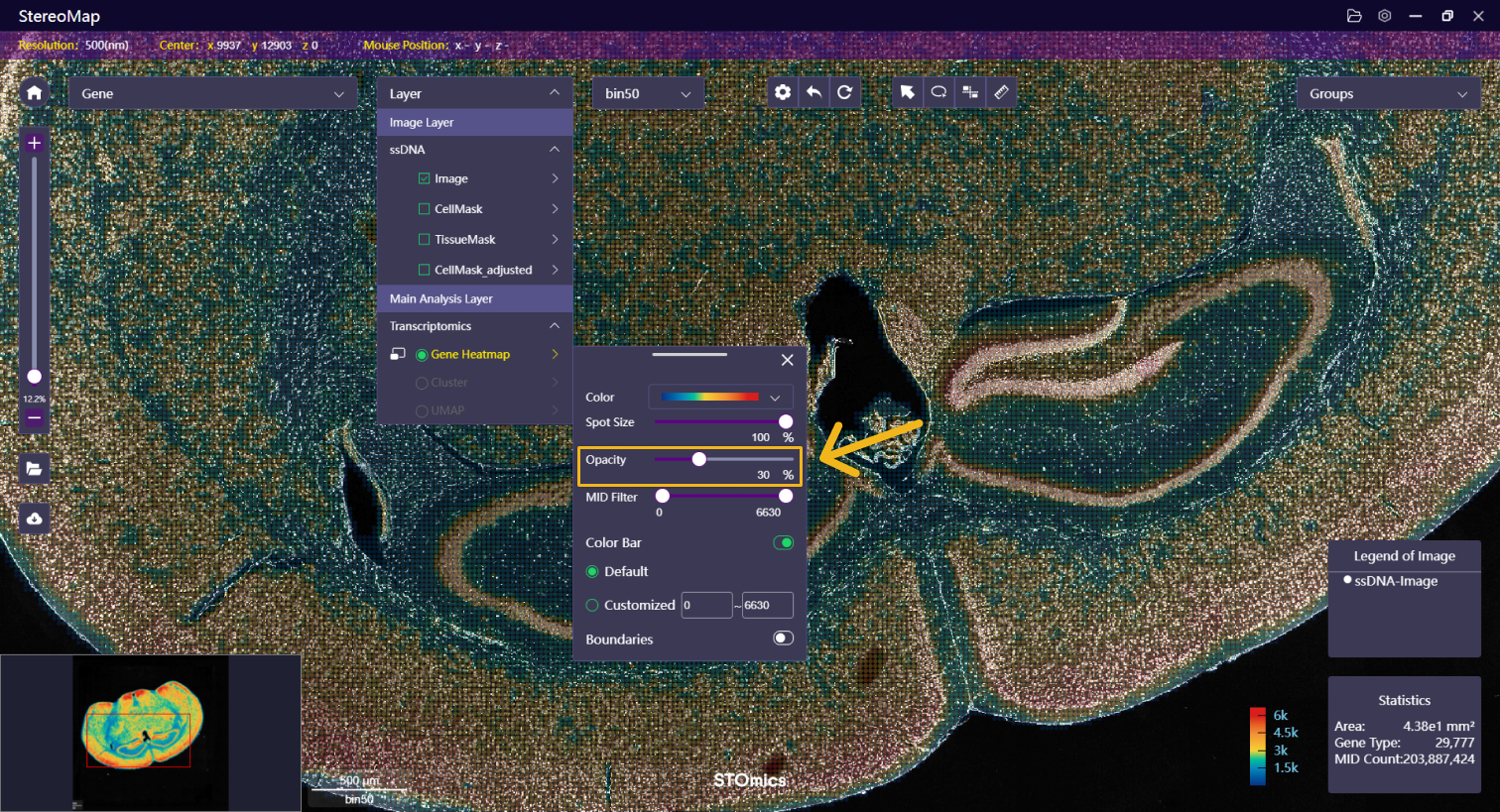 | 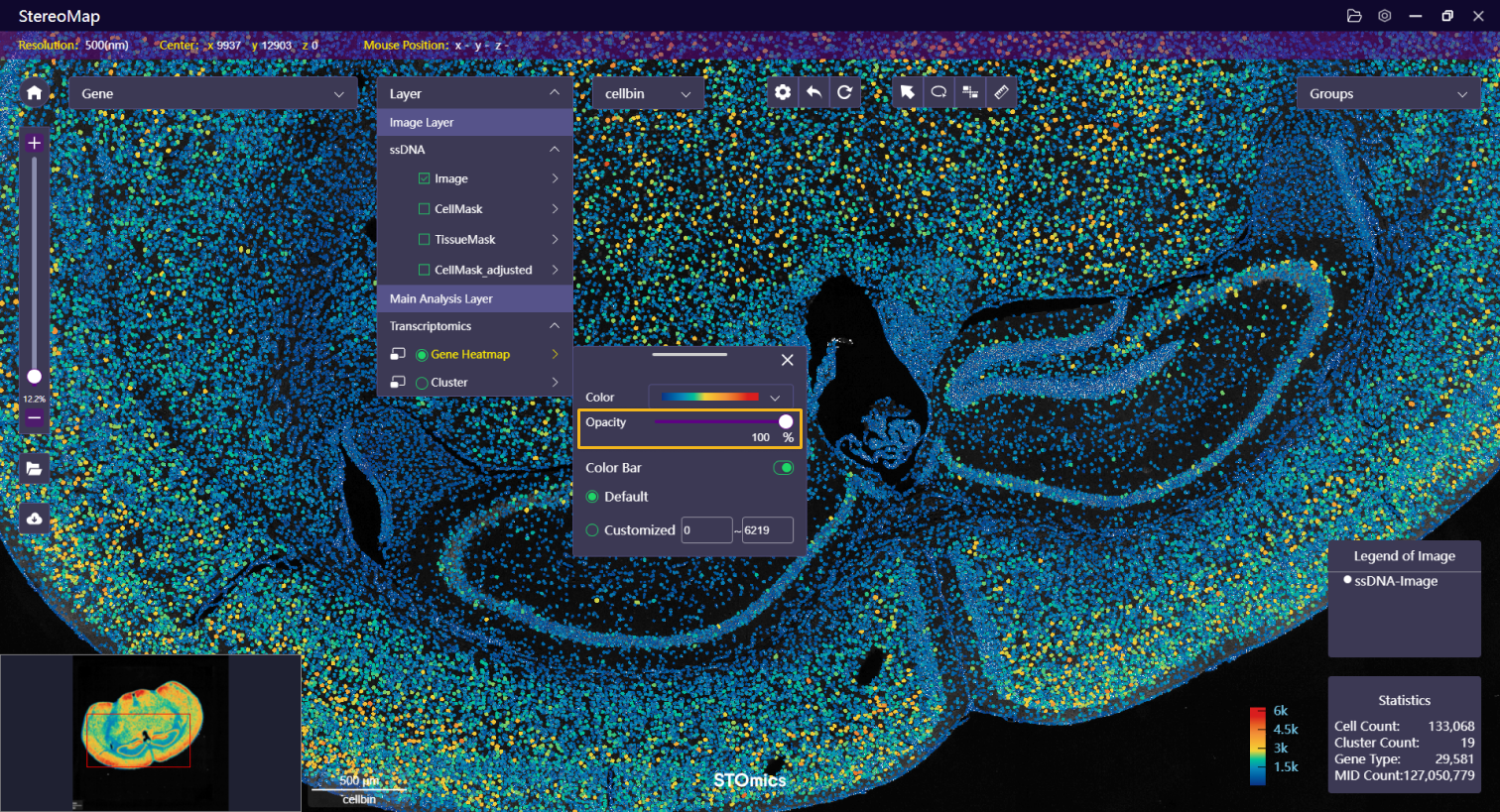  |
| MID 过滤 | 基于 MID 的计数过滤 spot点,有关更多信息请参考 MID过滤。
| 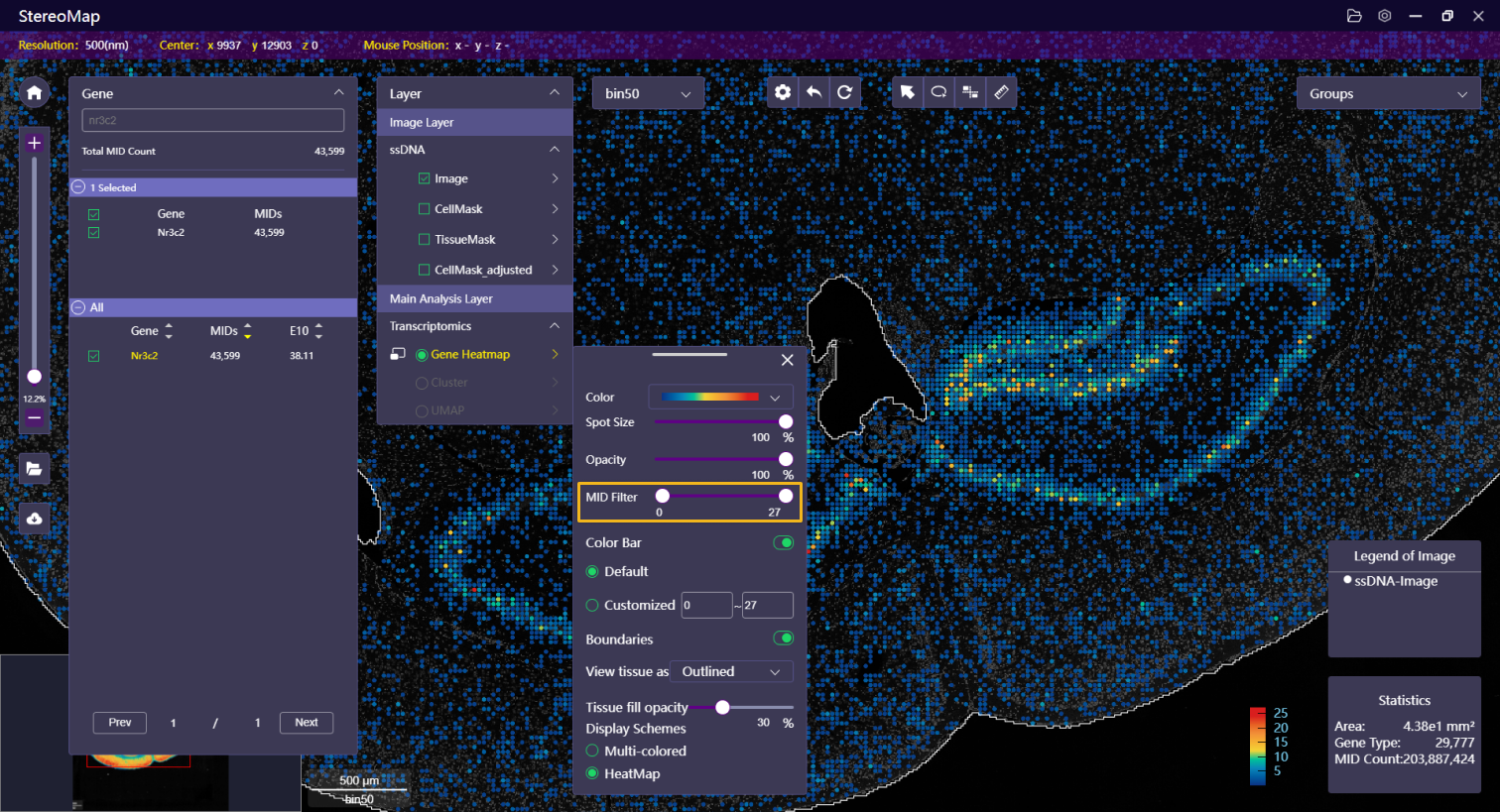 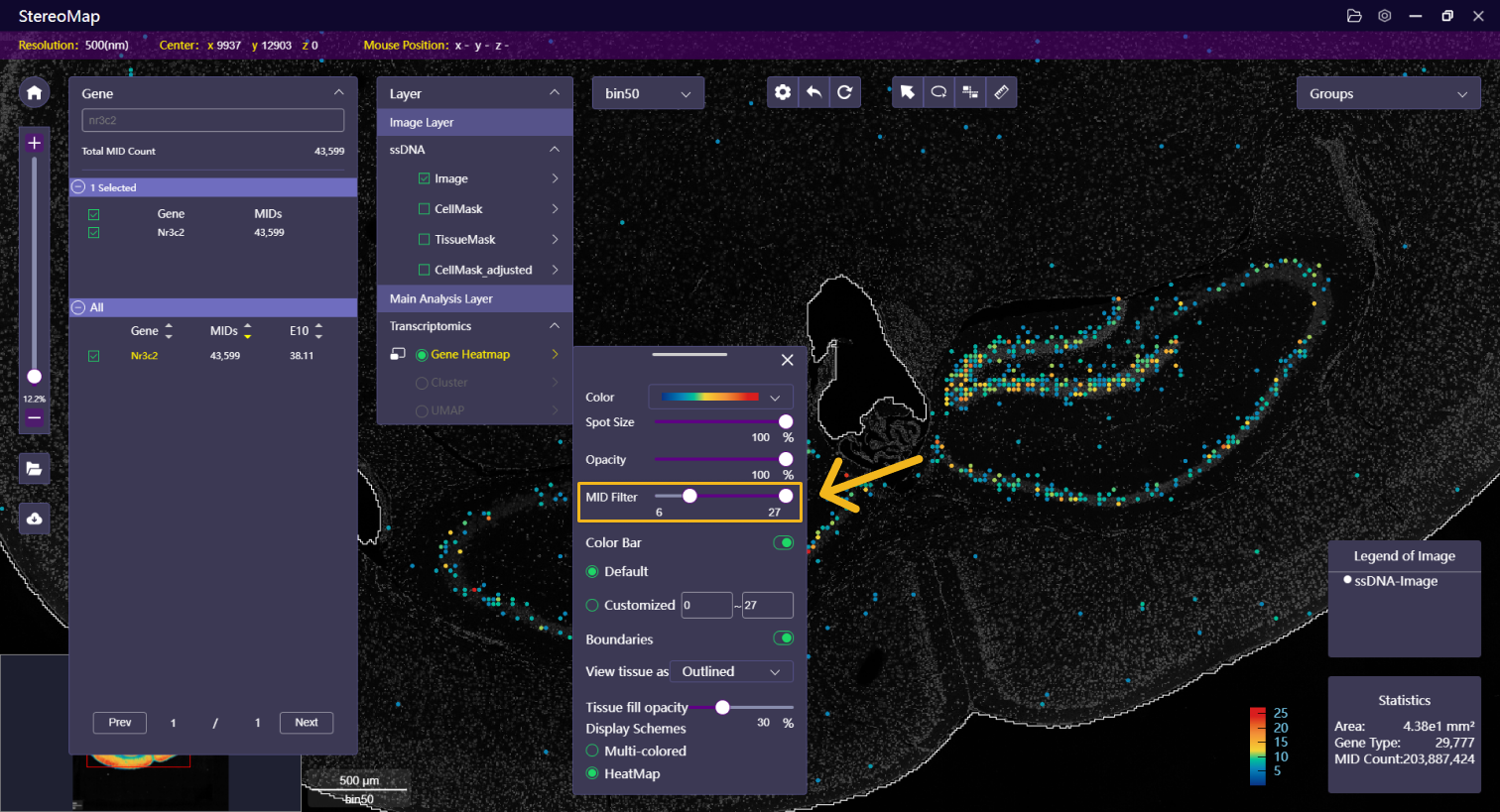 | NA |
| 色阶尺 | 默认情况下,色阶尺的范围为 0~MID 的最大值,用户可以自定义色阶范围,以便更好的可视化。 | 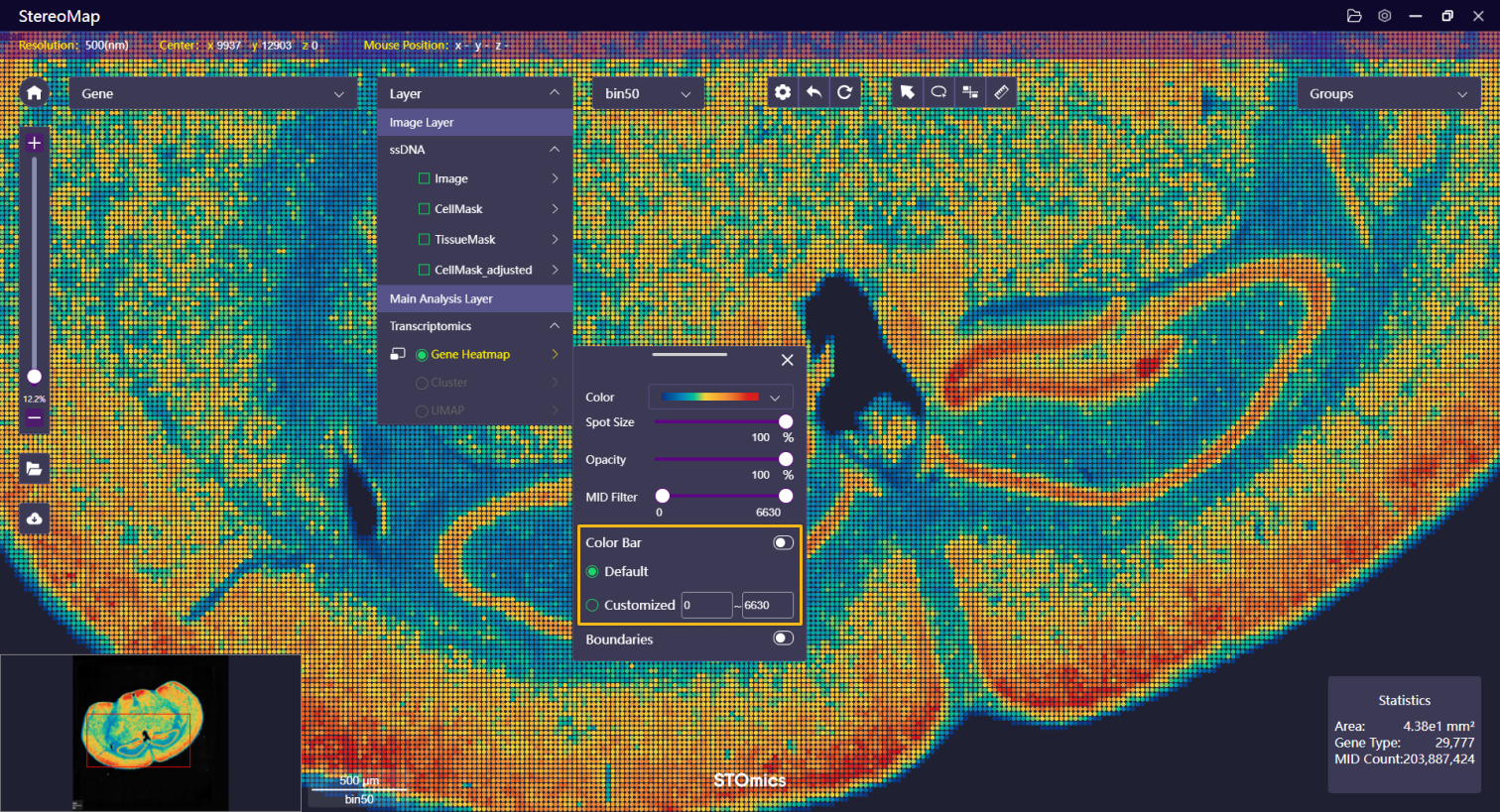 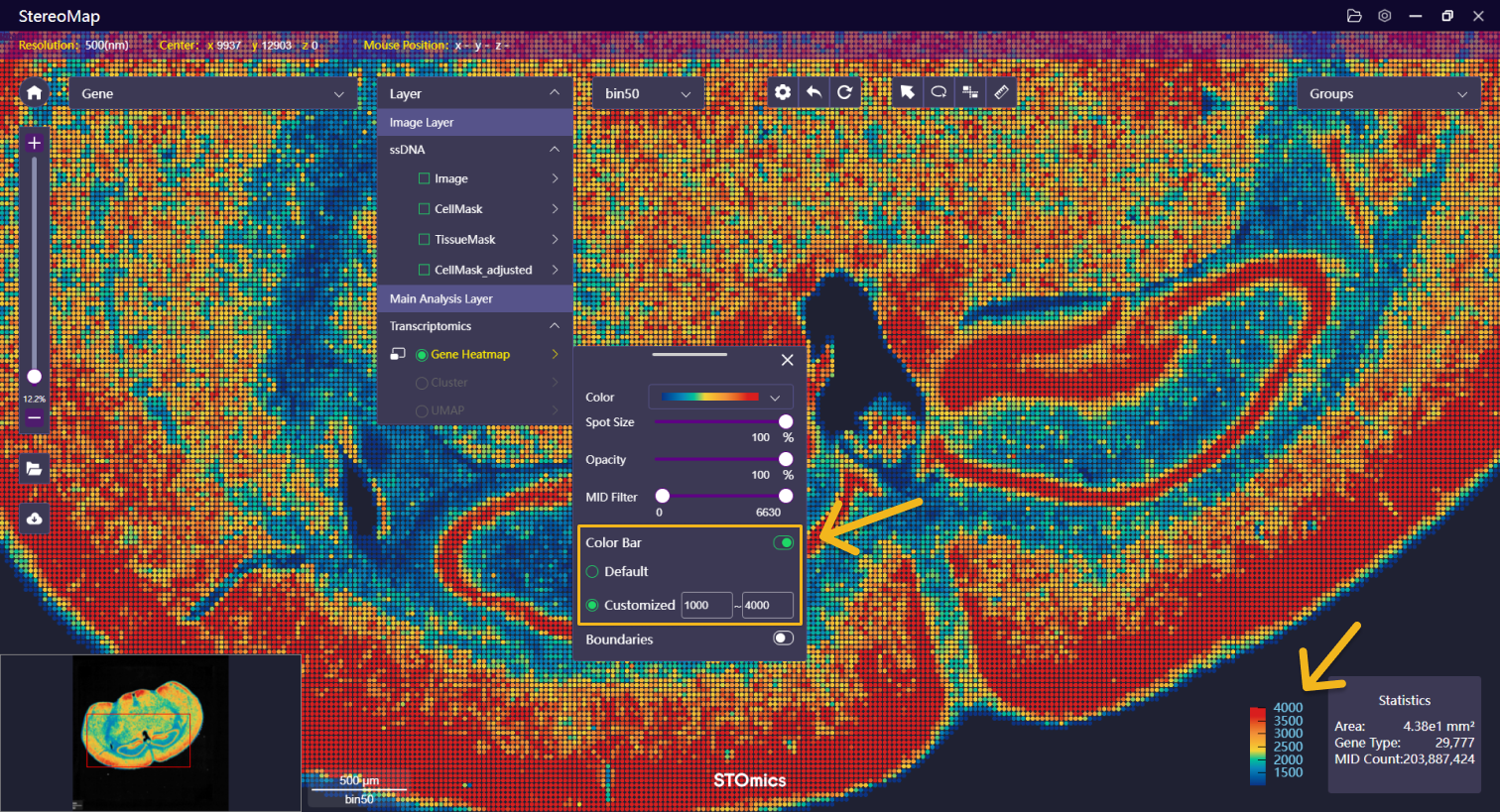 | 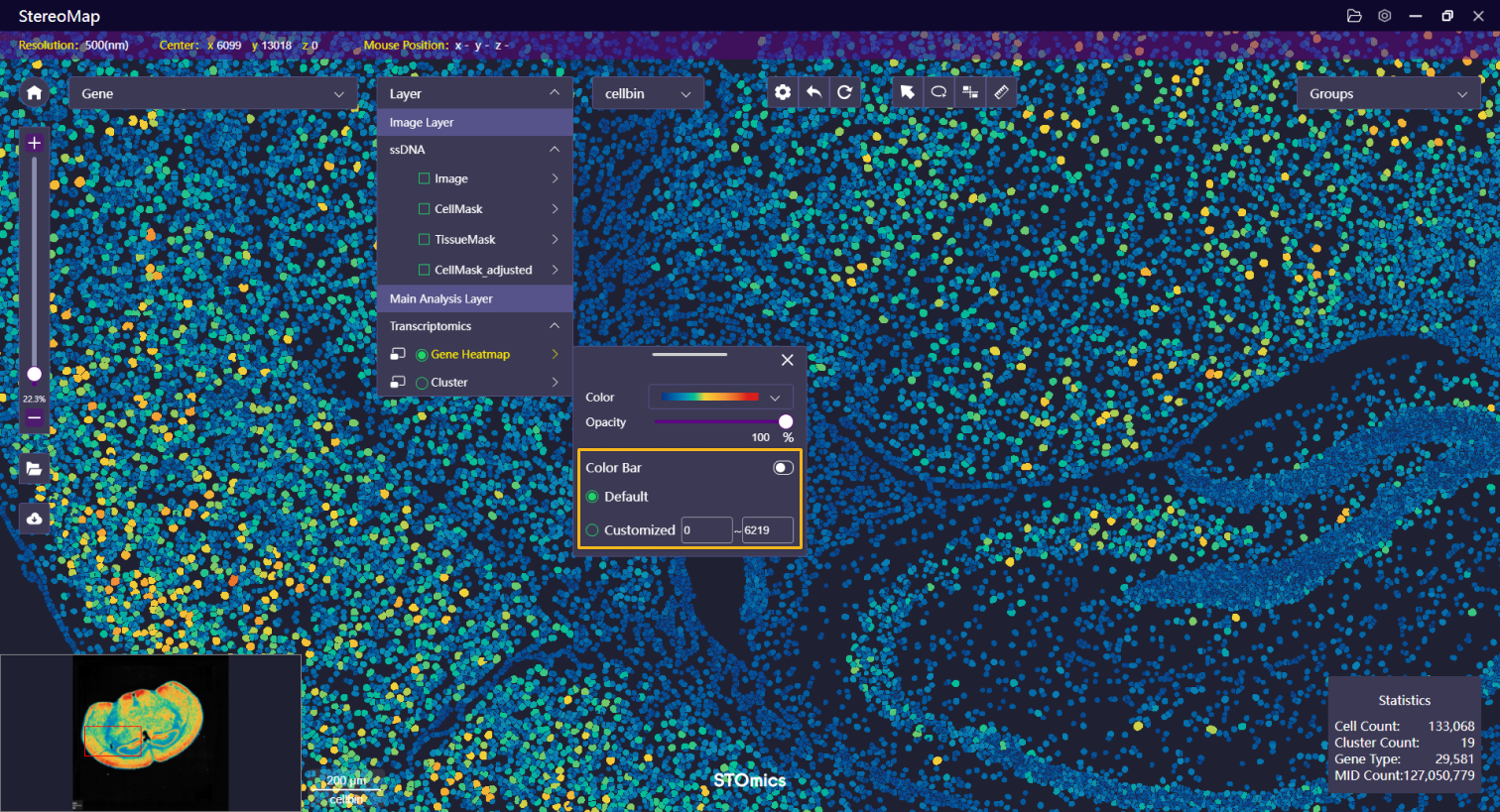 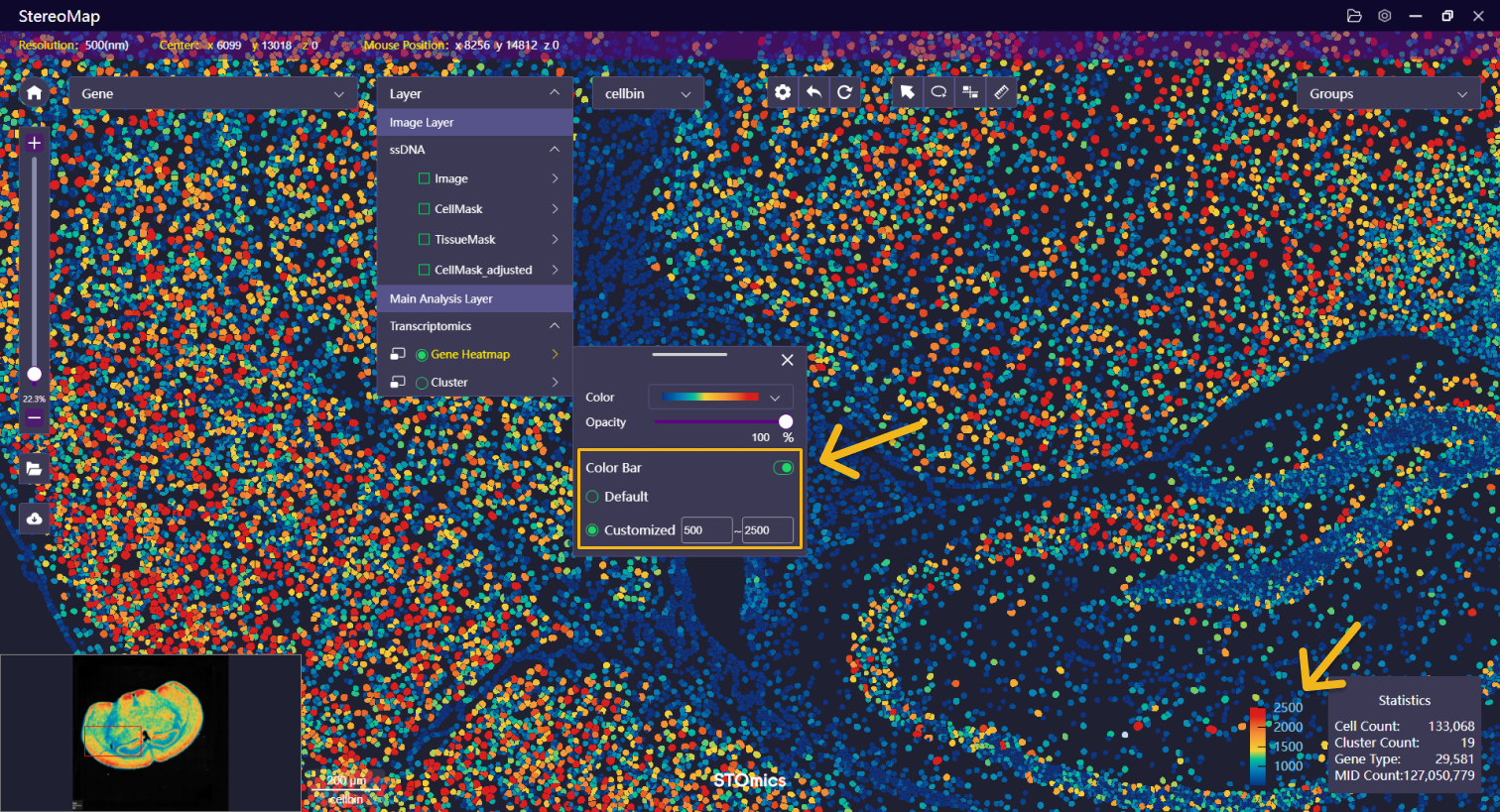 |
| 边界 | 可以选择显示或者隐藏由表达矩阵生成的组织边界,边界分为:轮廓、填充和两者兼顾,其中填充的透明度是可以调整的。 | 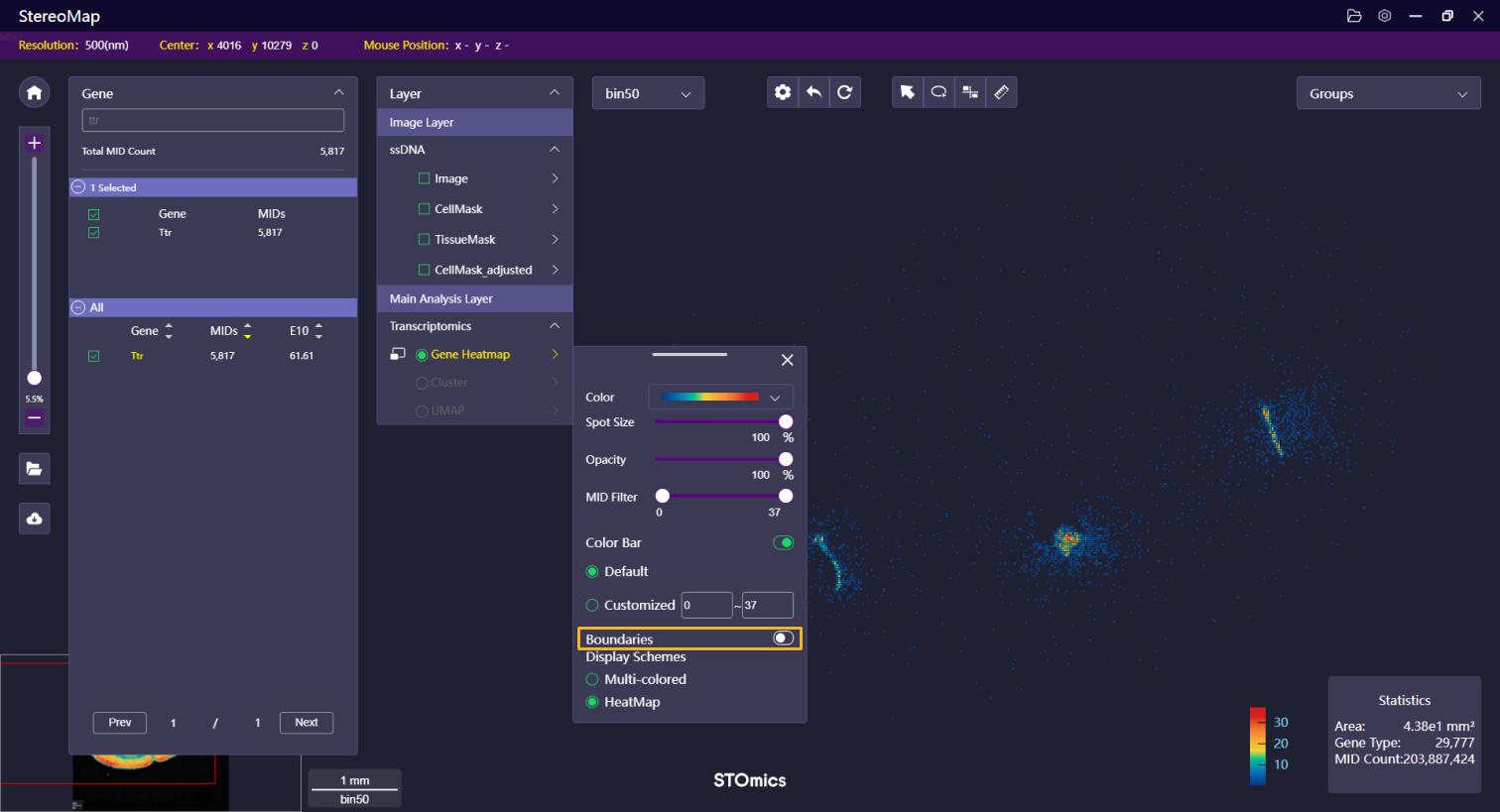 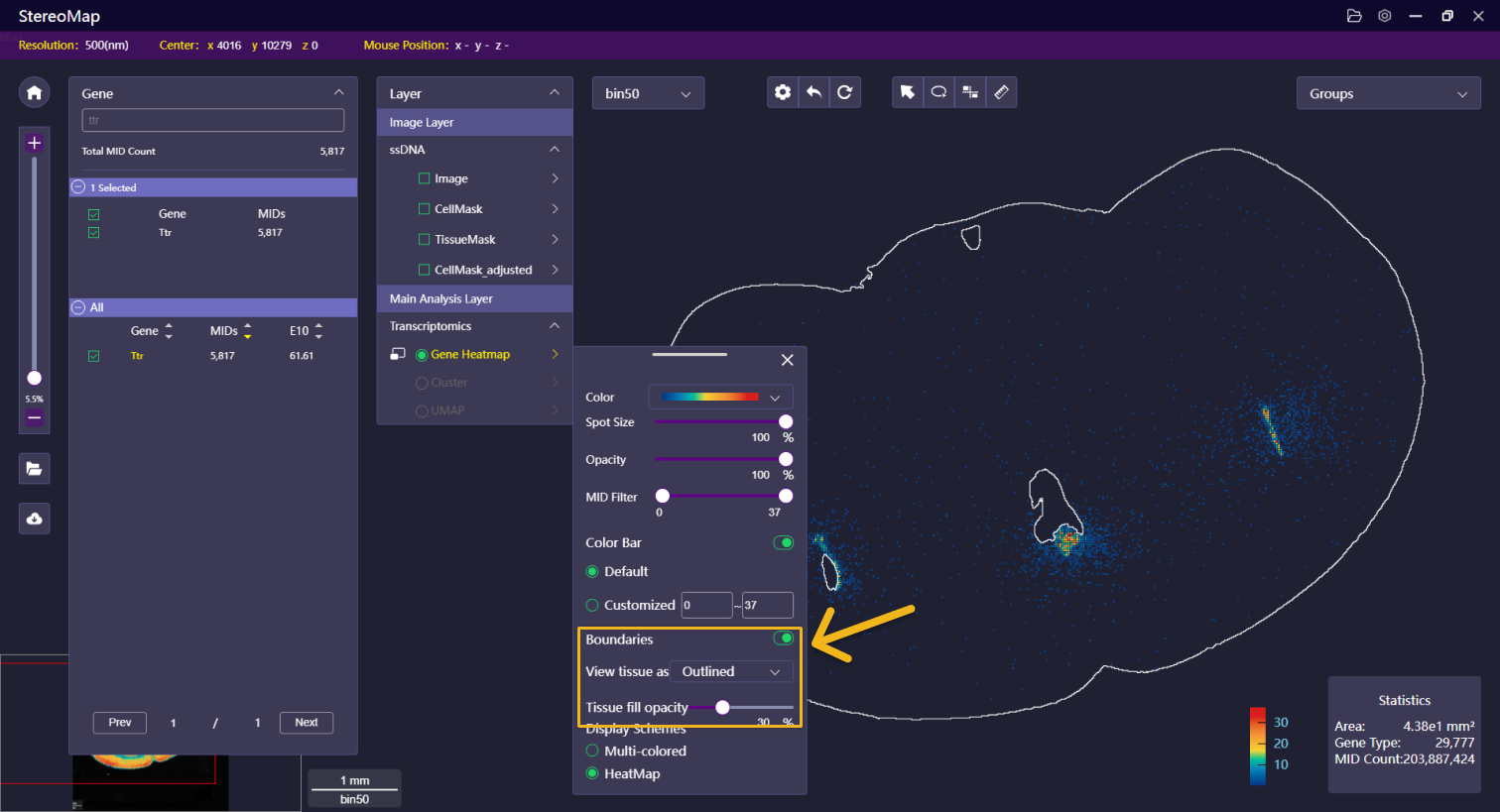 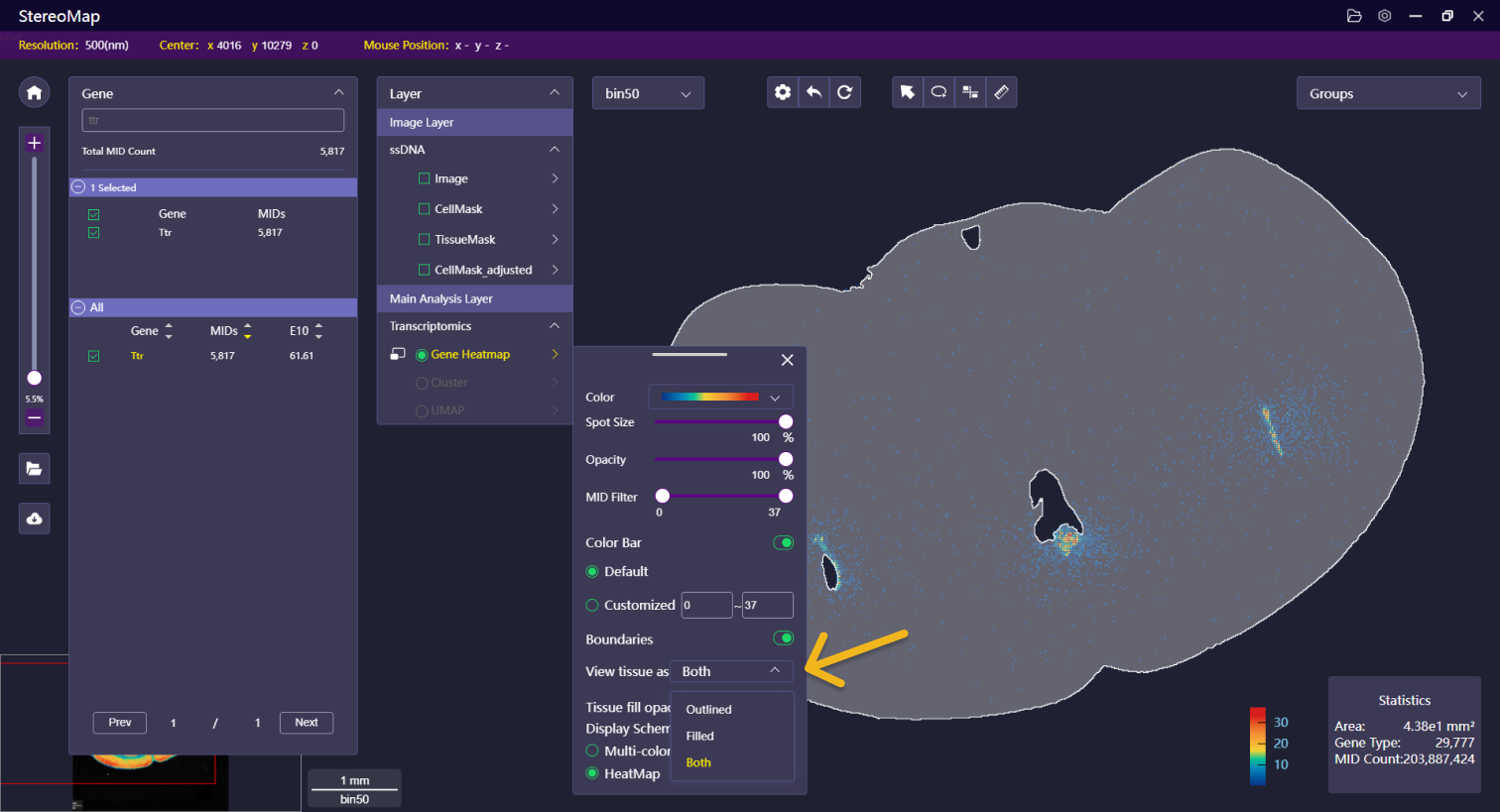 | NA |
| 展示形式 | 仅在勾选某个特征(基因/蛋白)后显示此选项,即以热图和伪彩图形式展示。有关更多信息请参阅基因共表达分析。 | 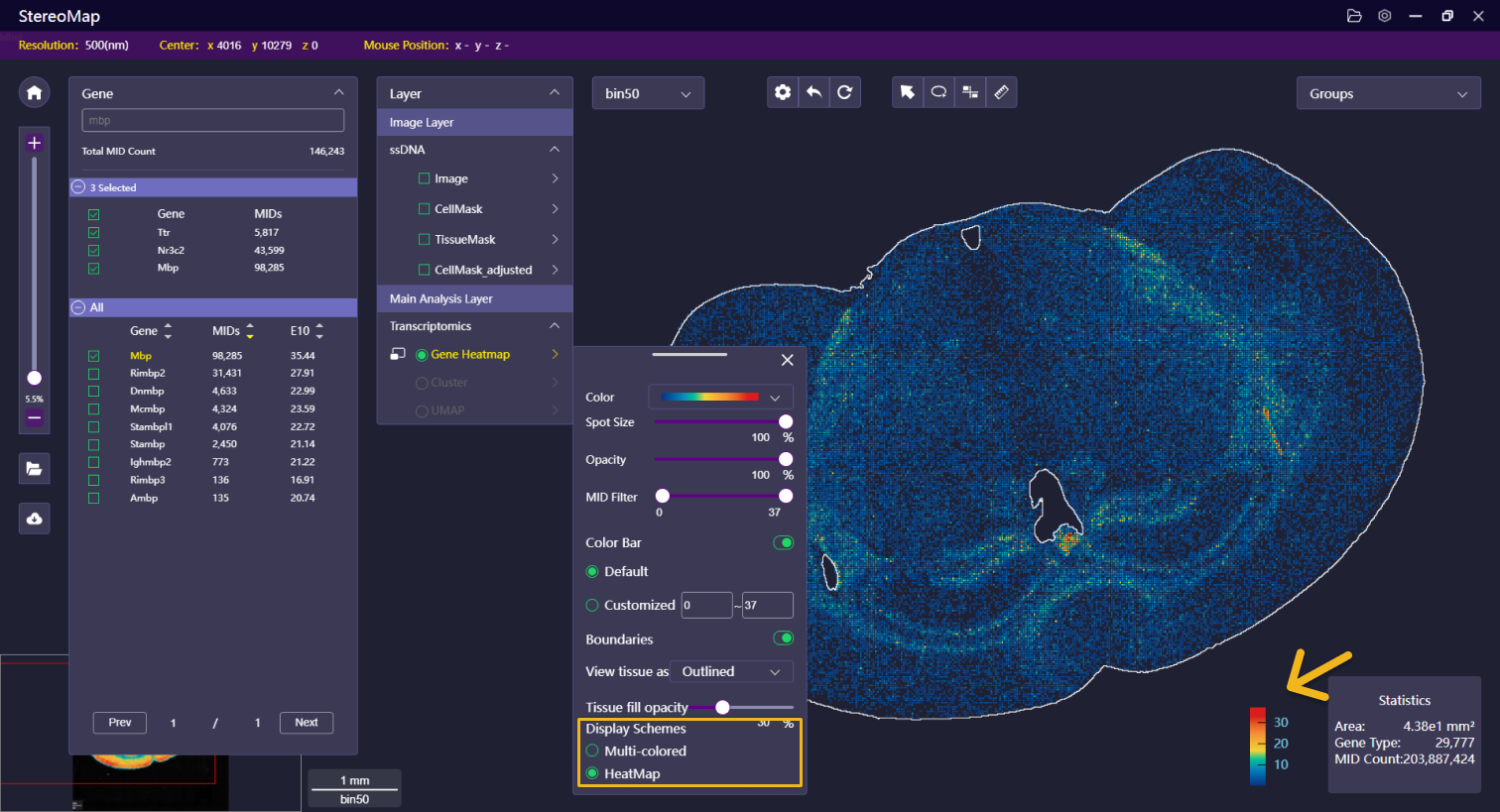 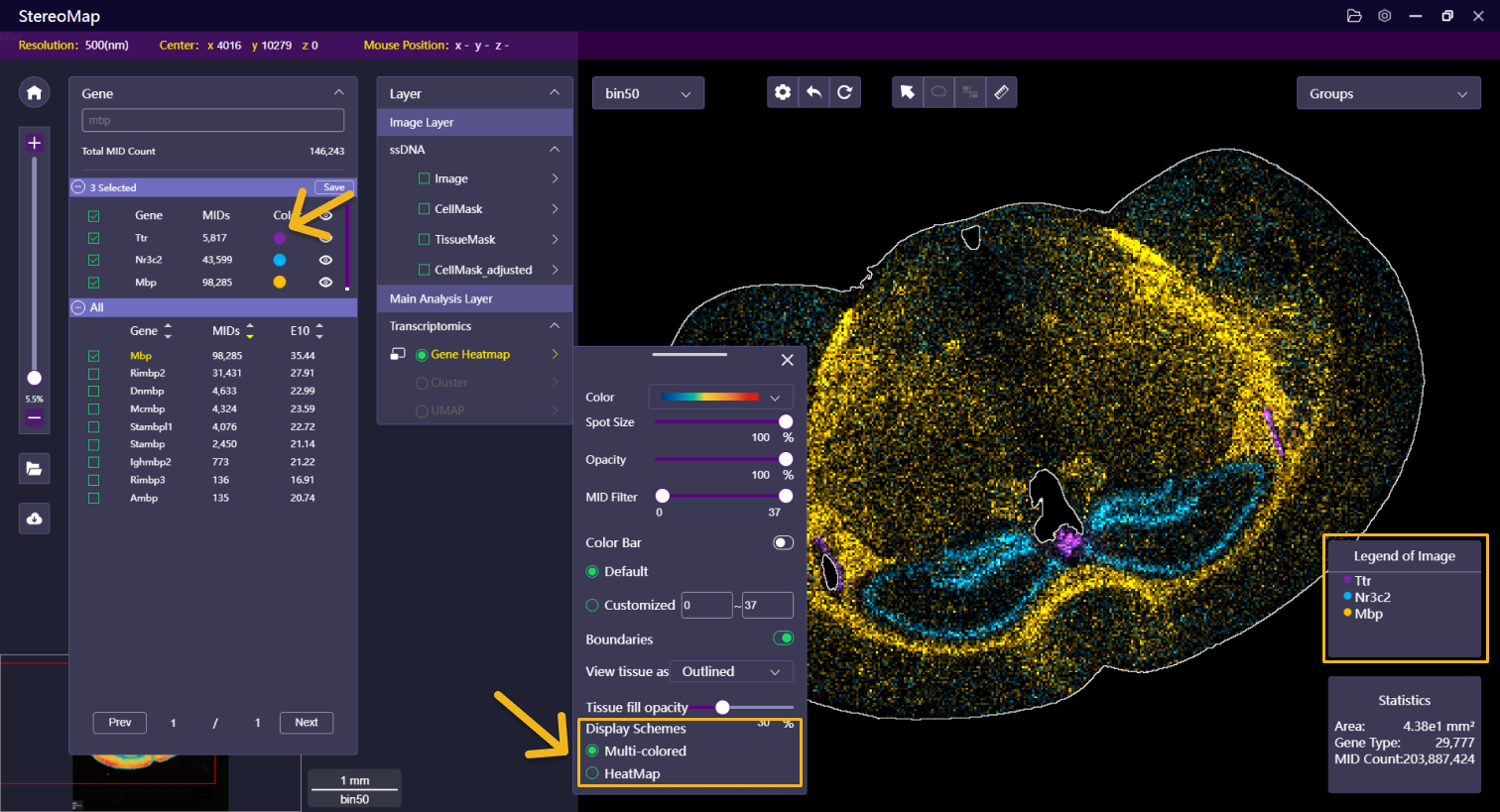 | NA |
聚类图层可以调整的选项包括:
| 聚类&UMAP | 说明 | BinN | Cell Bin |
|---|---|---|---|
| 透明度 | 调整聚类图层的透明度以便更好的可视化展示 | 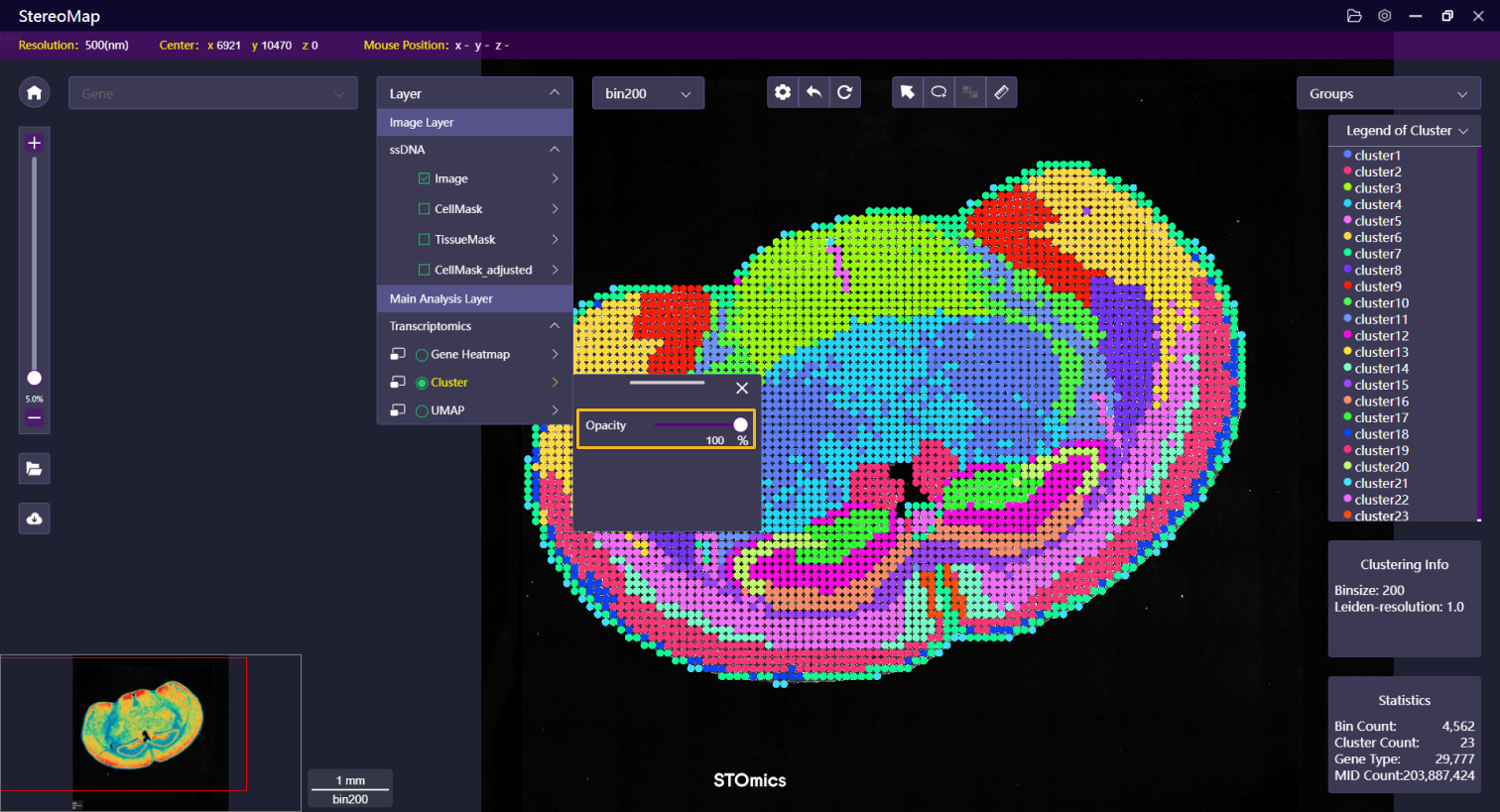 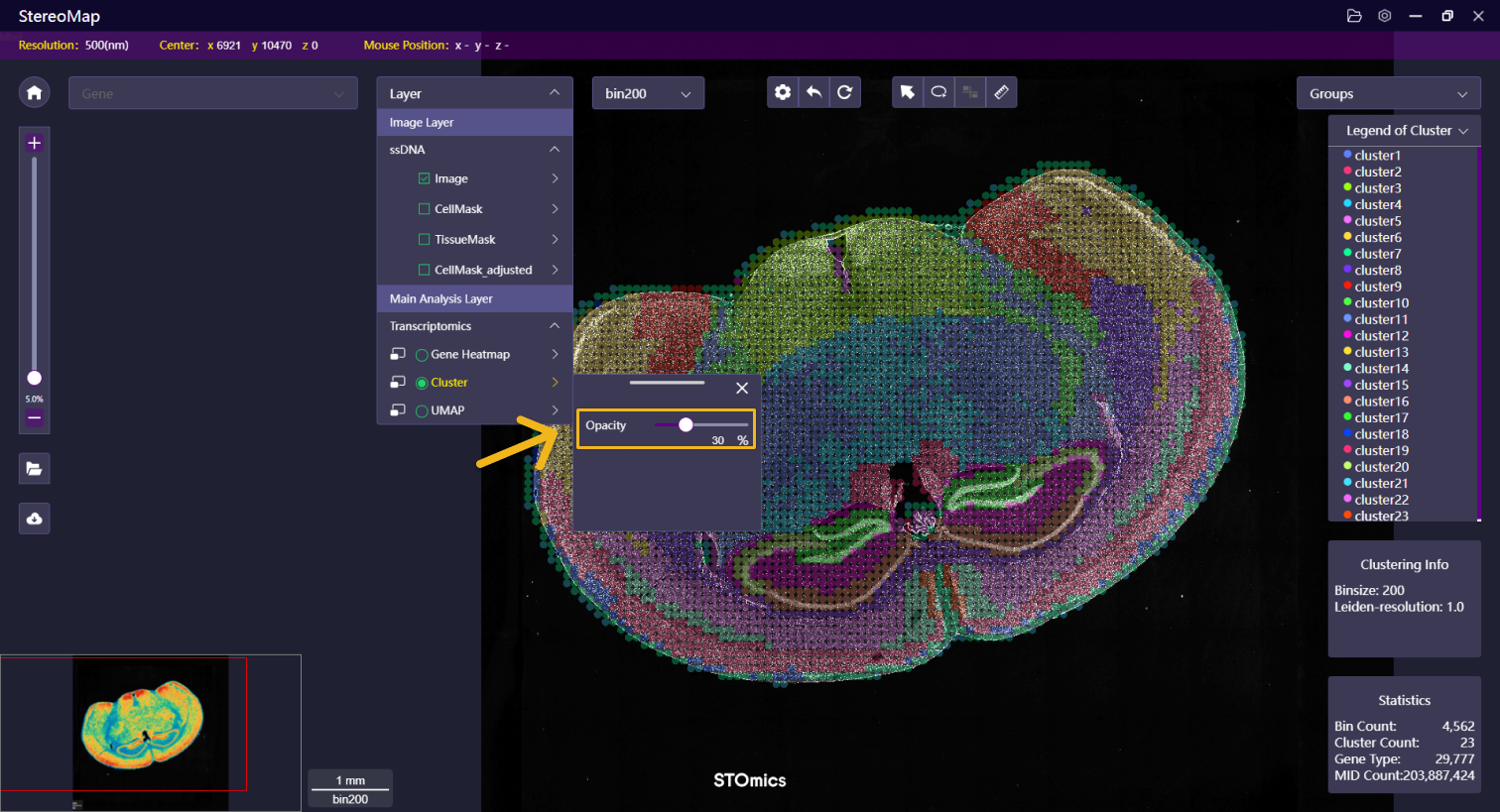 | 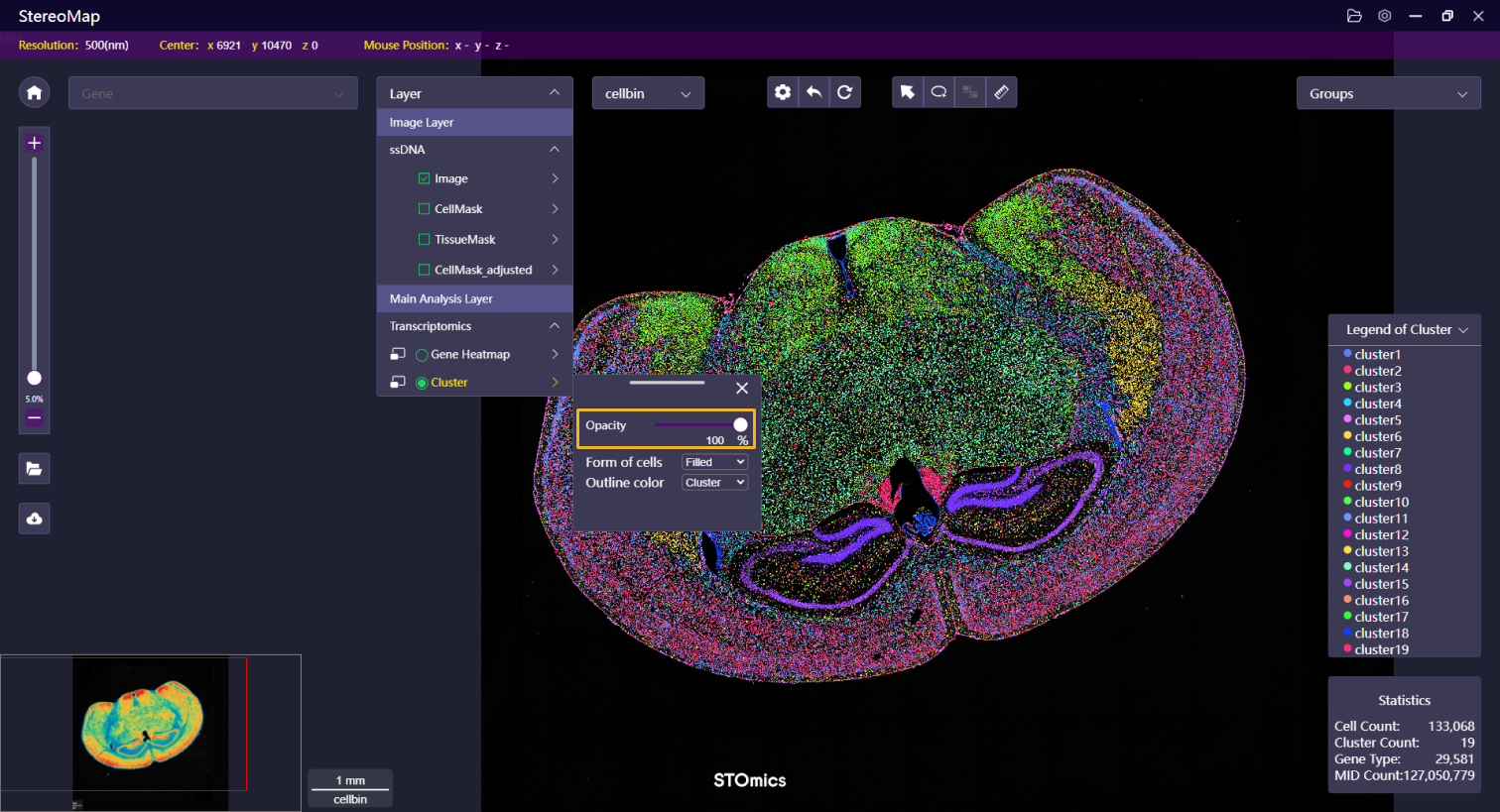 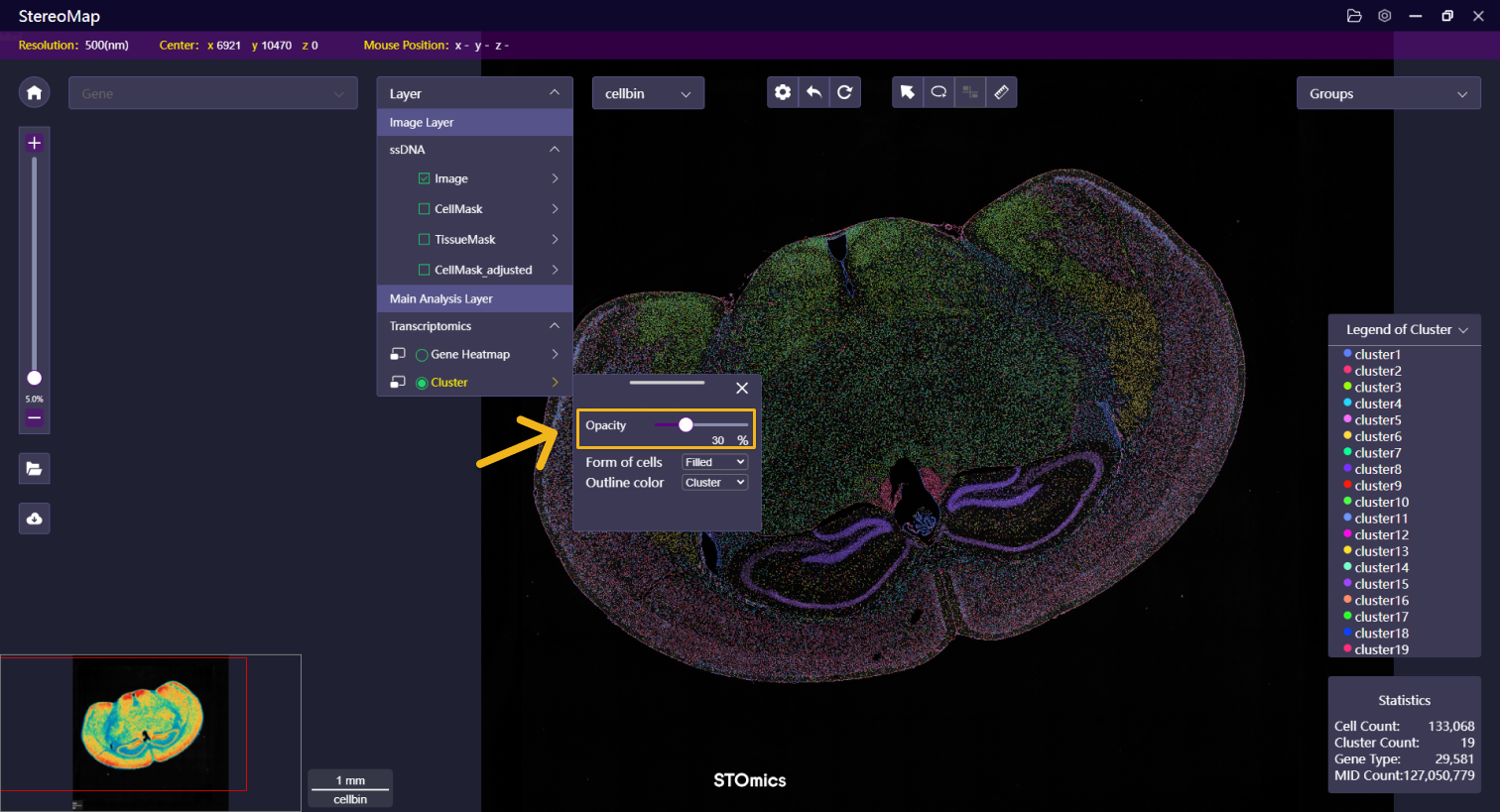 |
| 细胞展示形式和轮廓颜色 | 细胞的展示形式可以设置为:填充、轮廓或两者兼顾。细胞轮廓颜设置为聚类的颜色、白色、黑色和绿色。 | NA |    |
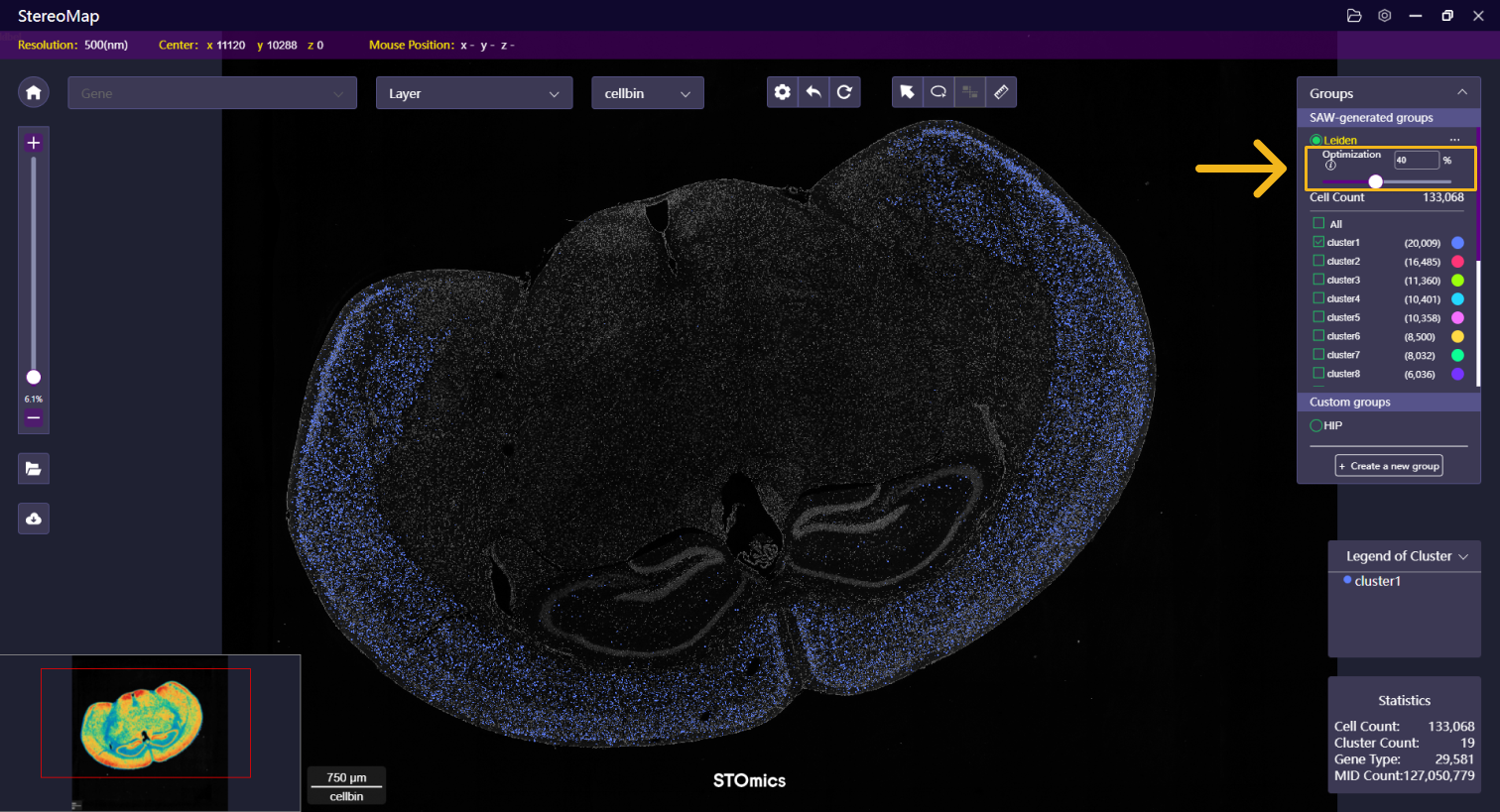
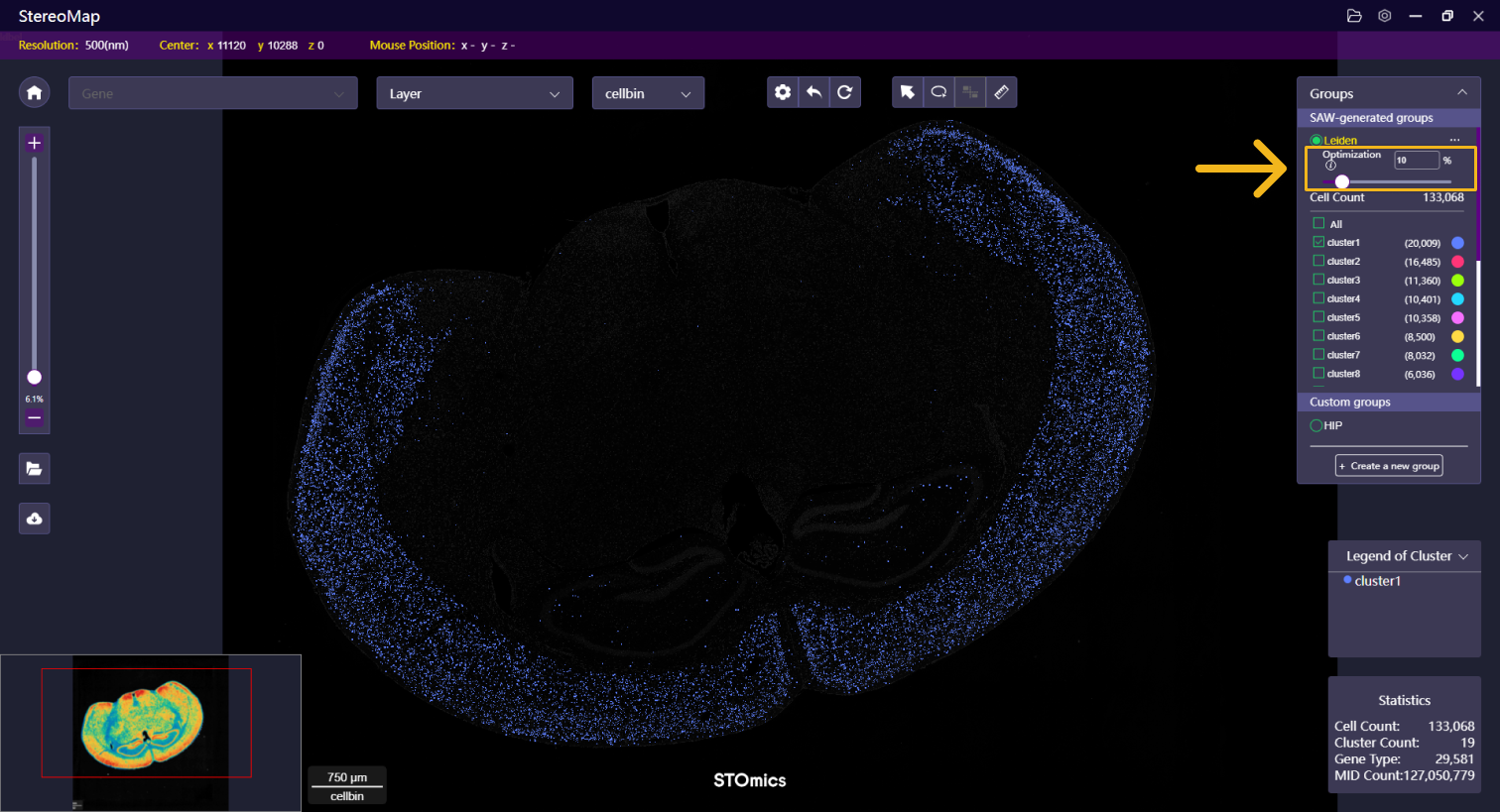
Group 菜单
Group 包括两种类型: SAW 生成的组和用户自定义的组,其中 SAW 生成的组主要展示 SAW 流程生成的聚类文件,流程包括 SAW count, SAW realign及SAW reanalyze。
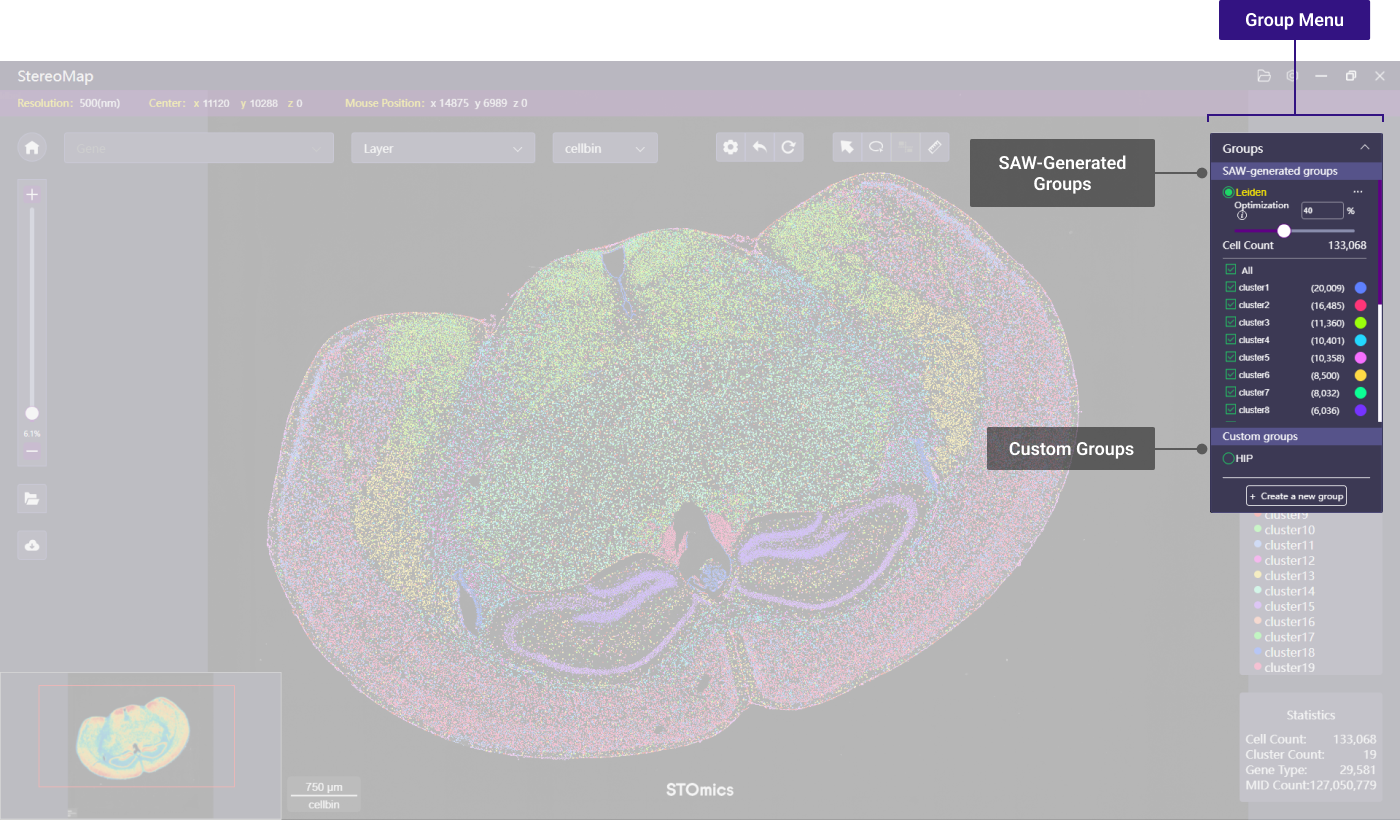
您可以在聚类图层下展示 SAW 生成的组,默认情况下,展示的是分群颗粒度为 1.0,binsize 大小为 200 或 cellbin(如果已经根据组织图像数据做了细胞分割)下的聚类数据。
- 效果增强:滑动滑块或者输入数值可以调整图像图层的透明度,以突出展示聚类图层。
- 复选框:点击聚类名称前面的复选框
,可以展示/隐藏某个聚类。
- 颜色:点击右侧打开调色板,拖动拾色器上的小圆圈/彩虹带即可修改选中的聚类颜色。
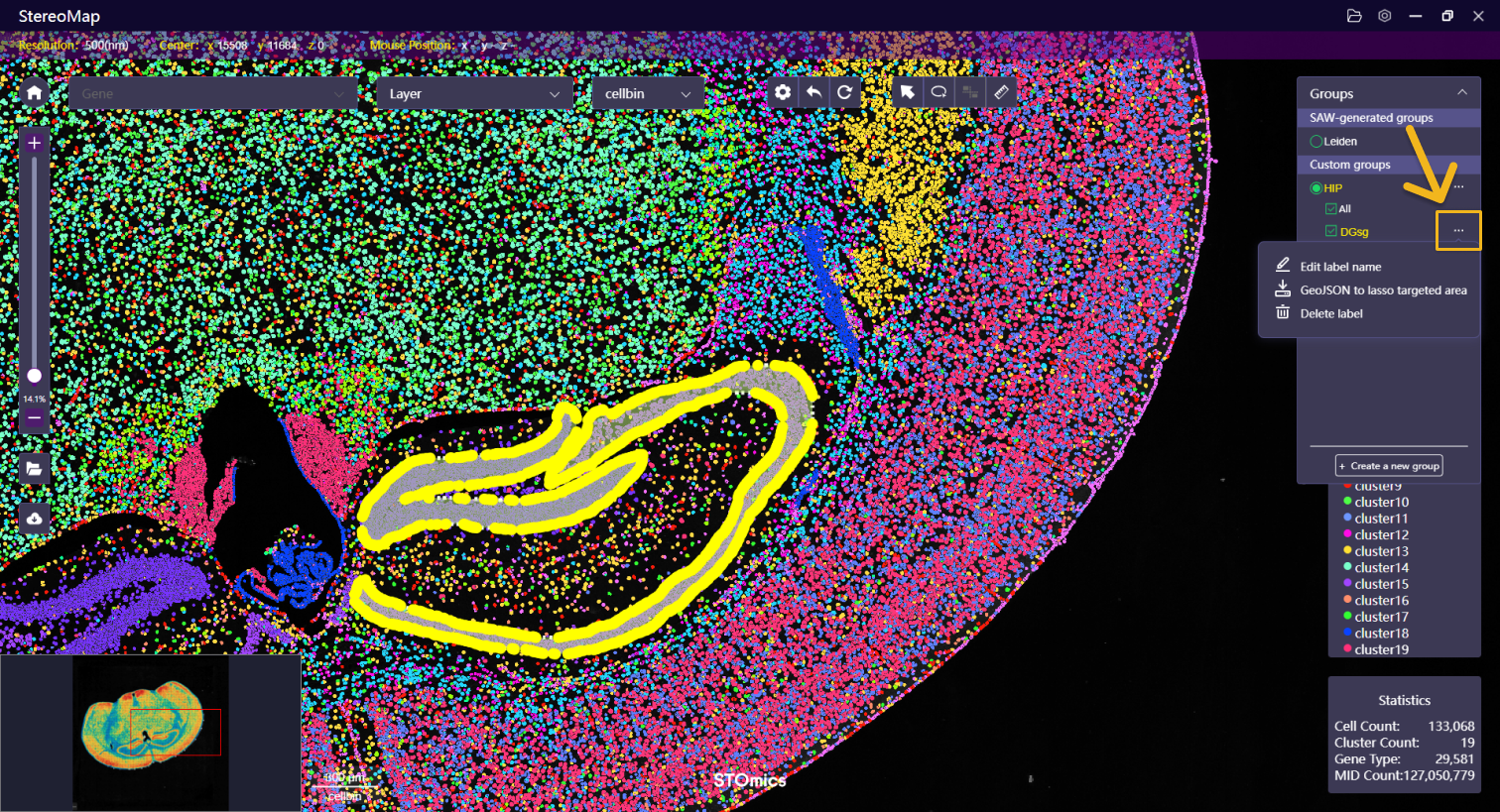
您可以使用套索功能选择一个区域,为该区域命名,并将该区域分配给某个组(如果没有可选择的组名,可以直接创建新的组)。或者,您可以通过单击 + Create group 来创建新的组,并在保存套索标签时将标签分配给新创建的组。
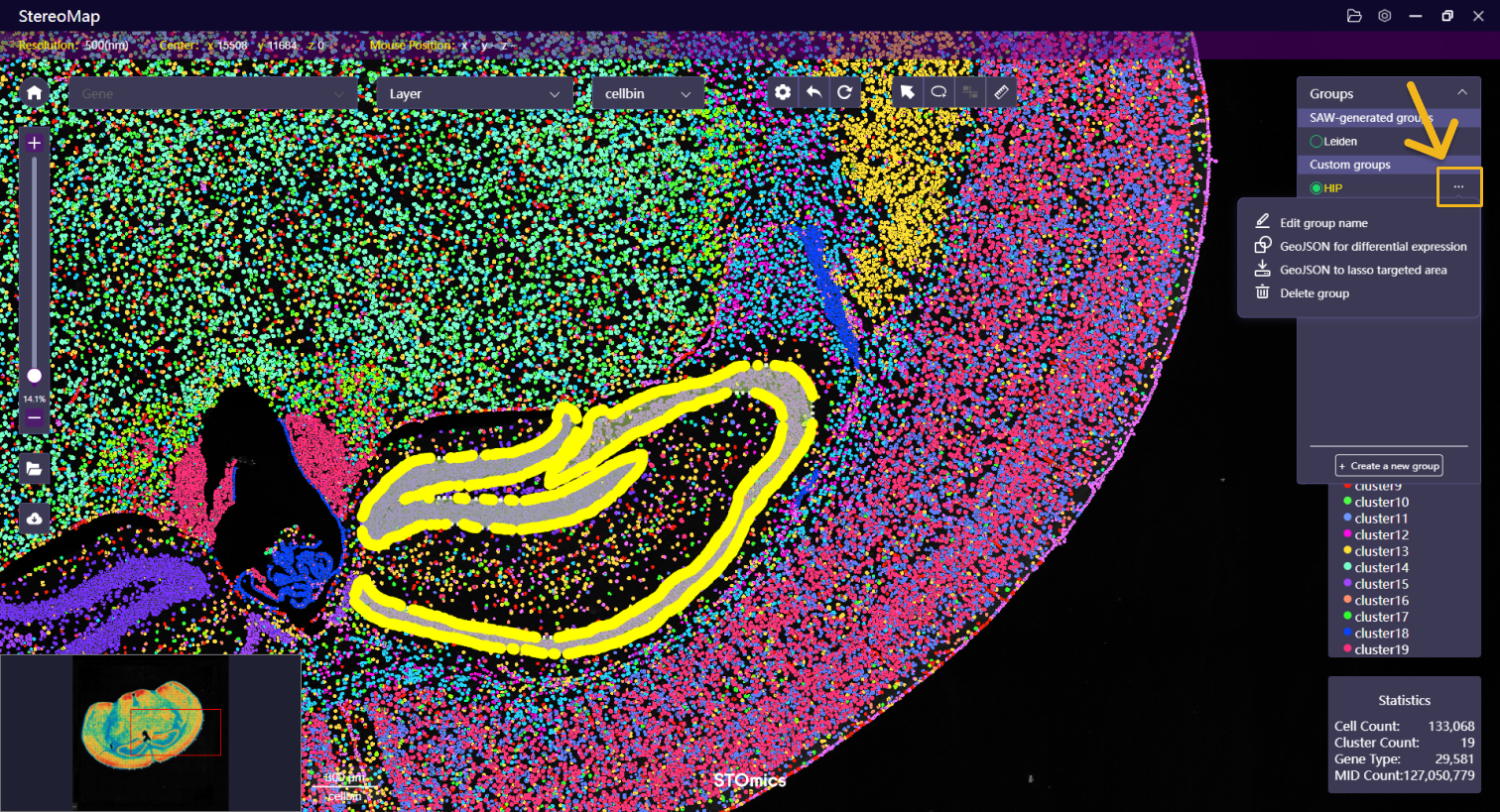
- 点击更多图标
 可以编辑组的名称或者按照 GeoJSON 的格式导出套索的区域。您还可以点击GeoJSON for differential expression 导出差异分析的参数,请参考差异分析获取更多详情。
可以编辑组的名称或者按照 GeoJSON 的格式导出套索的区域。您还可以点击GeoJSON for differential expression 导出差异分析的参数,请参考差异分析获取更多详情。
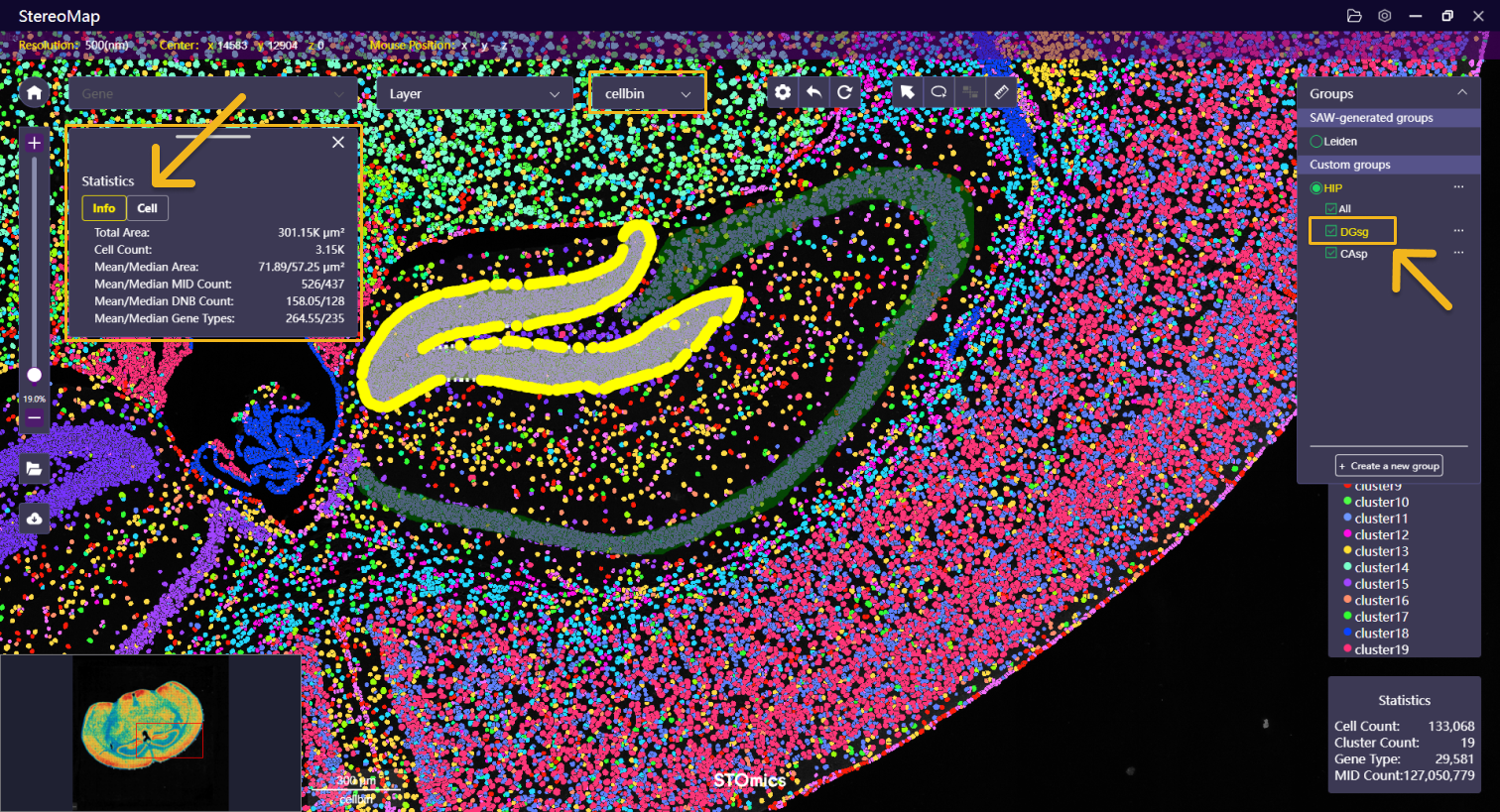
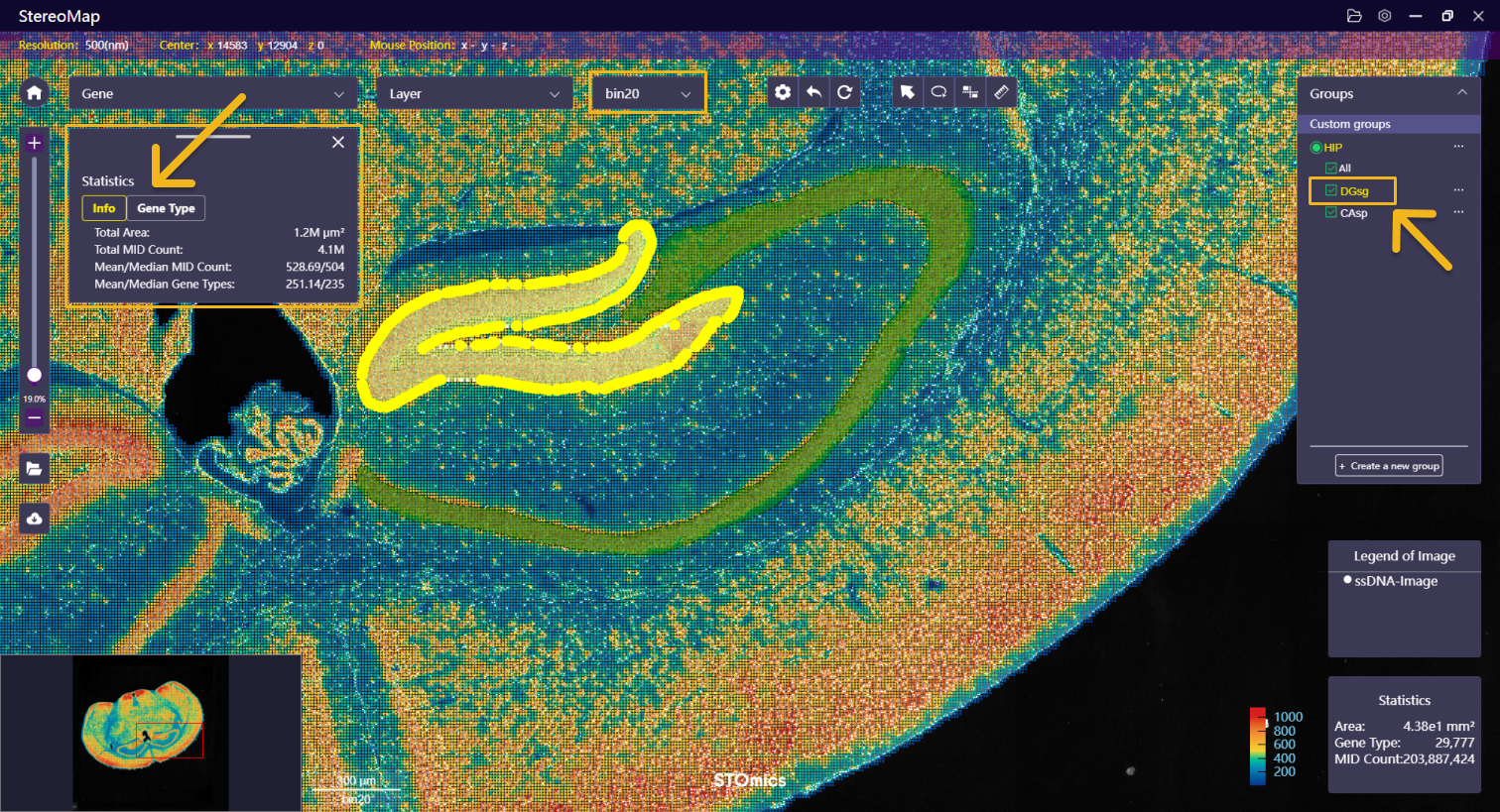
- 点击套索区域的名称,该名称及对应的套索区域会用黄色高亮展示,并在左侧浮动的面板中显示相应的统计信息。







.png)