Stereo-CITE T FF
通过空间多组学数据探索组织环境
研究人员利用空间多组学的数据可以探索不同组学层面细胞的形成过程。通过 RNA 的表达预测蛋白质水平虽说存在局限性,但将空间转录组学和蛋白质组学的数据整合分析,可以为后续研究提供宝贵的互补见解,彻底改变我们对复杂生物的理解。
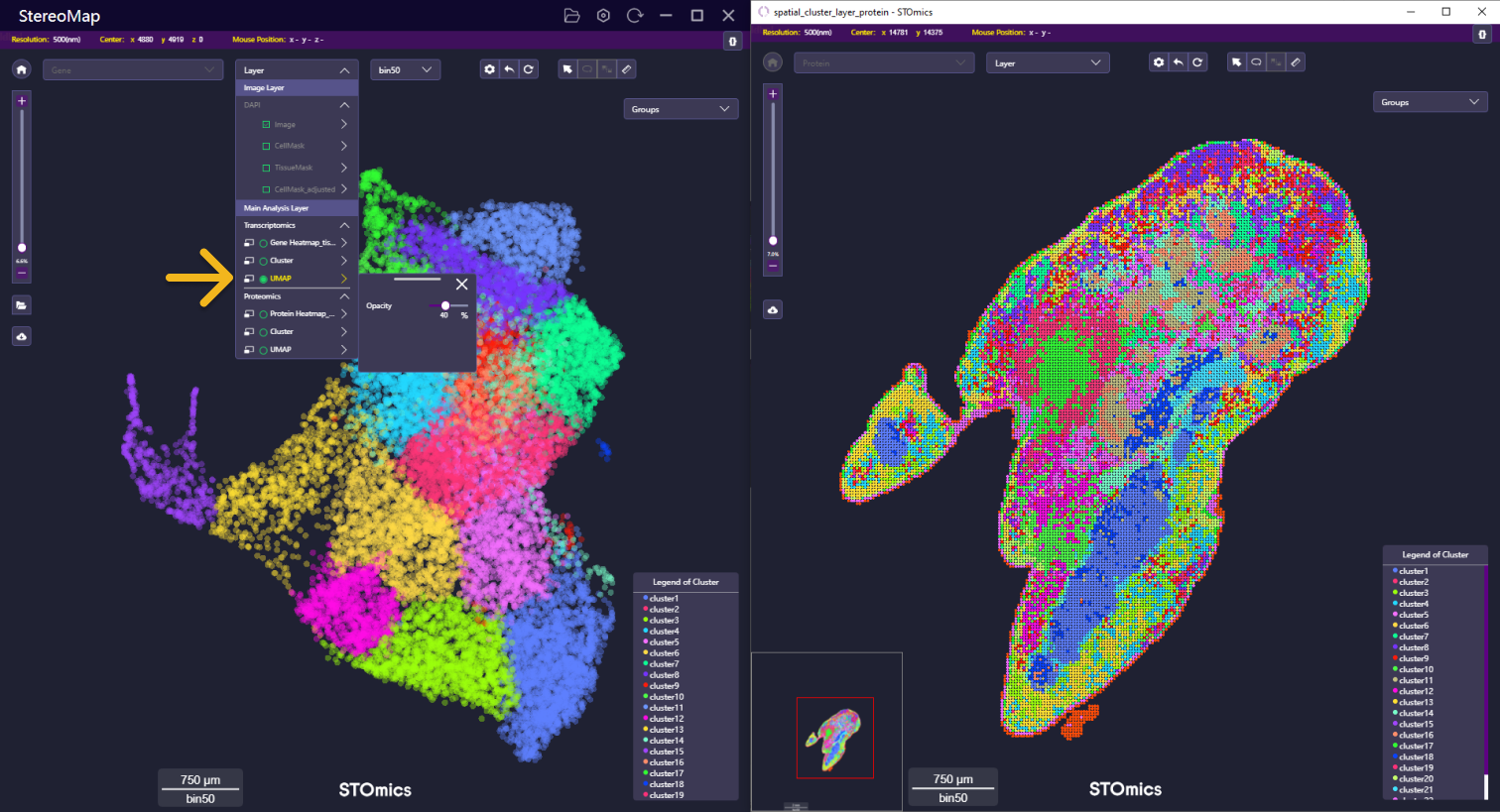
多窗口的联动可以根据相同的坐标点信息,同时操作同一数据集的多个窗口。
基于坐标联动操作的点很大程度上依赖动作的发起者,因此,只有主窗口可以作为发起方,联动的信息将反应到已打开的子窗口中。
选择某个 cluster 联动
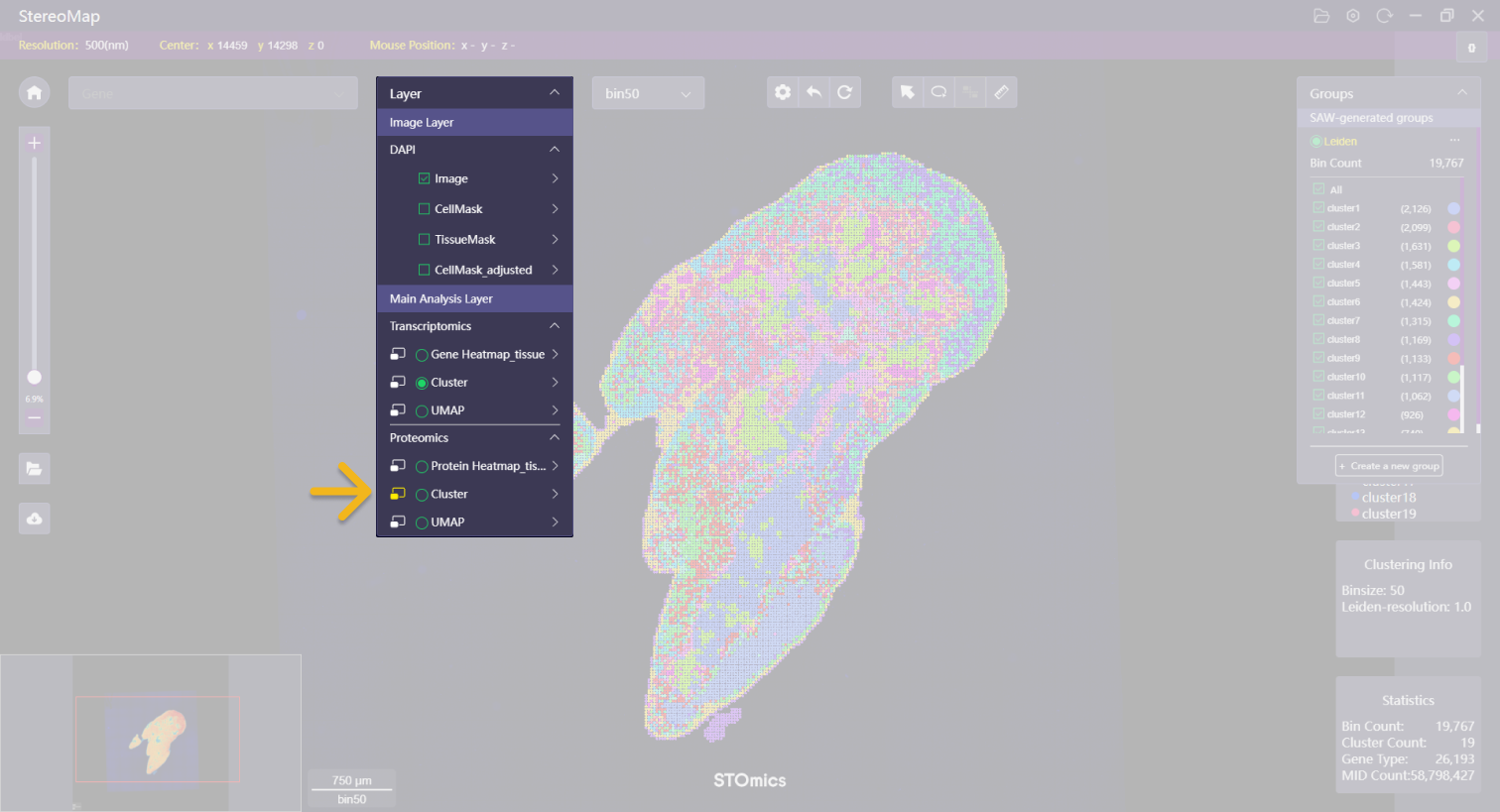
打开 Feature 菜单可以并排查看蛋白和基因的 cluster 图。点击蛋白组学 cluster 图层前面的  图标,可以打开一个新的 cluster 蛋白子窗口,您可以在主窗口中切换动作的发起方。
图标,可以打开一个新的 cluster 蛋白子窗口,您可以在主窗口中切换动作的发起方。
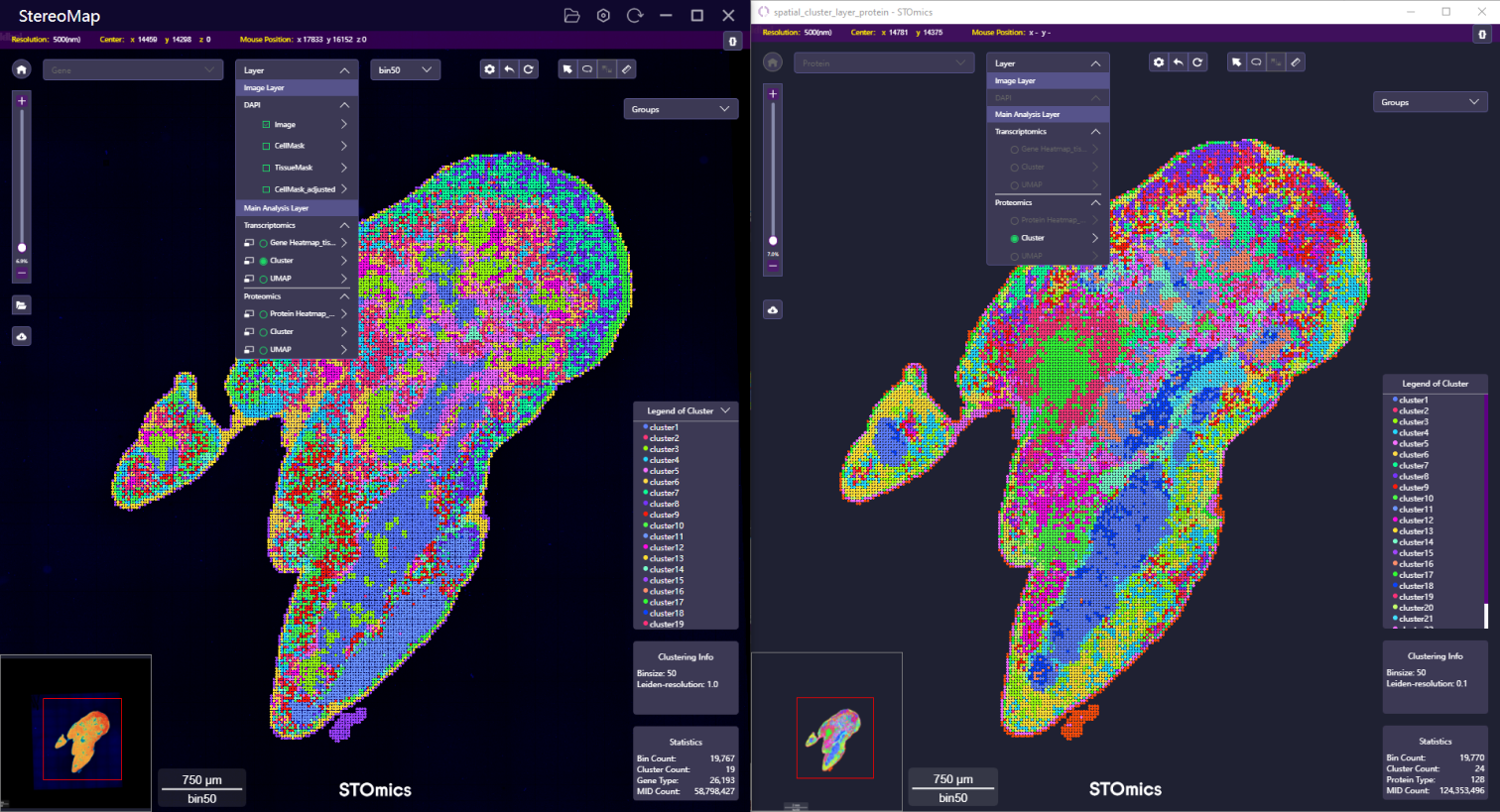
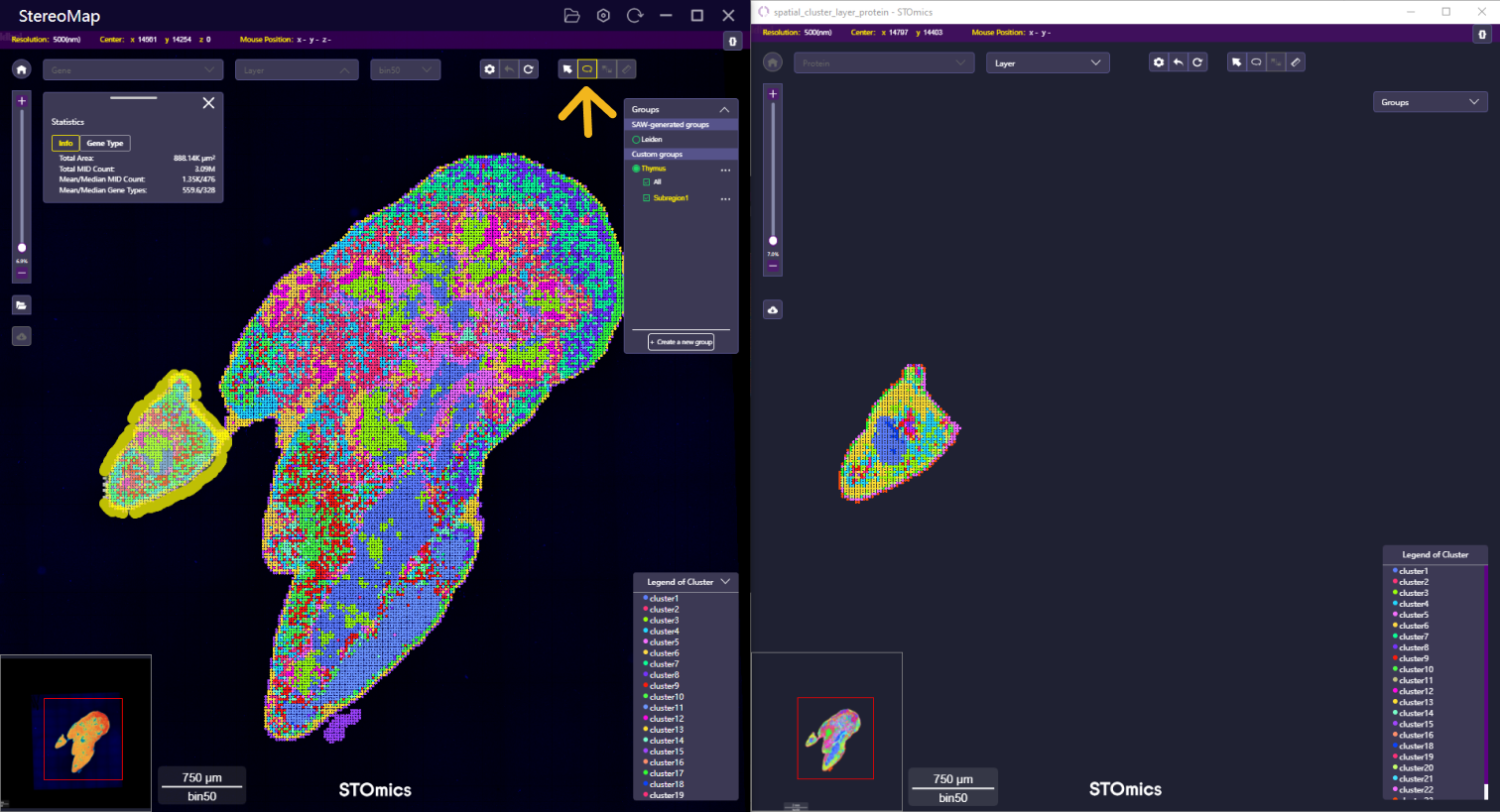
根据空间基因或蛋白质的表达水平做聚类分析,可能会存在相同坐标的点被归为不同的类别。如下所示:主窗口(左)展示的是基于空间基因表达矩阵的聚类结果,选择某个 cluster 后,画布中高亮展示该聚类的点。在子窗口(右)中,画布上仅显示跟字窗联动的坐标点,并根据蛋白的聚类结果进行着色。
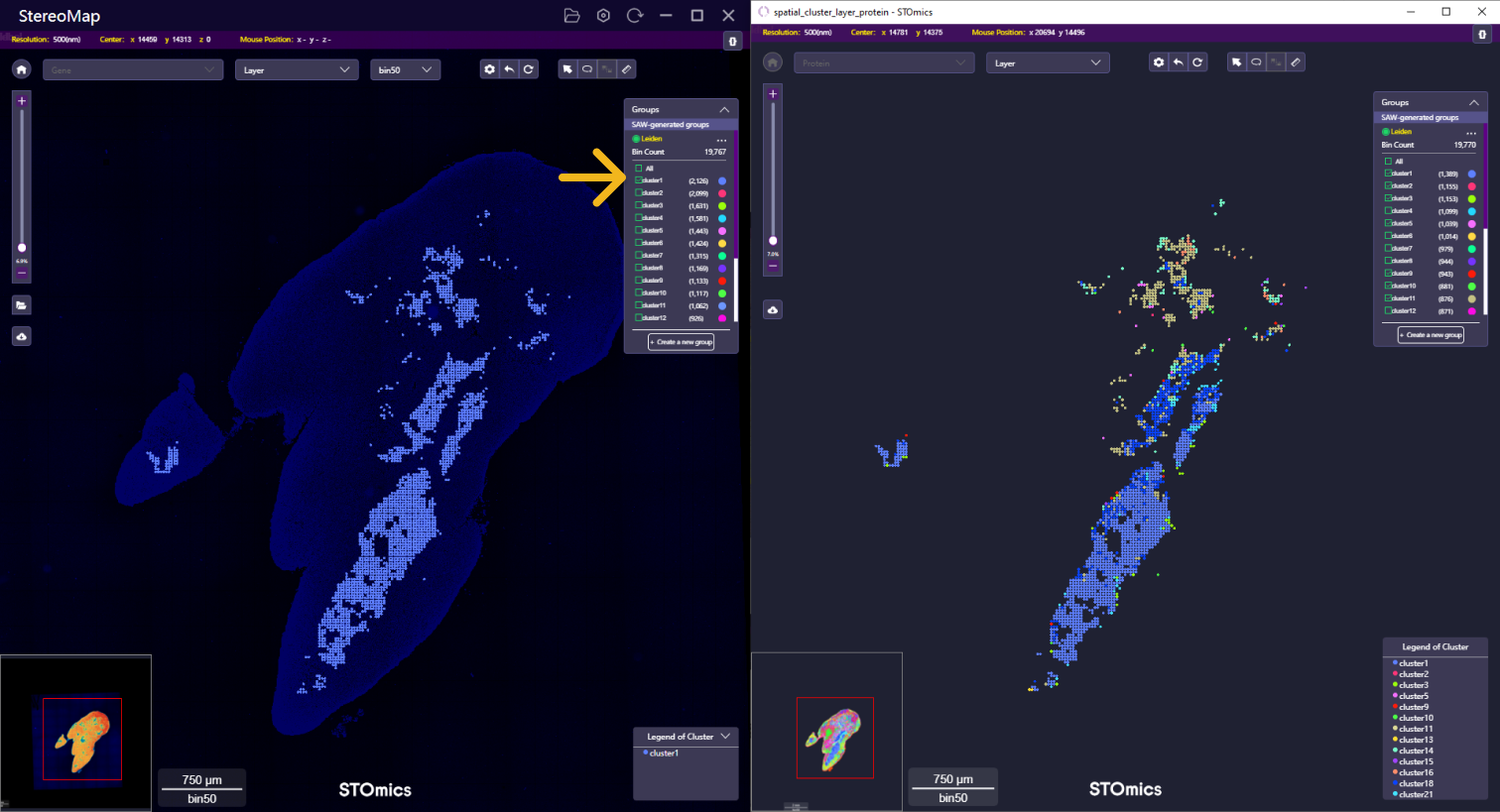
空间基因聚类结果 cluster 1 中的点坐标(左)在空间蛋白质聚类中具有不同的分类(右)
多窗口的联动同样也适用于UMAP图和空间聚类图。
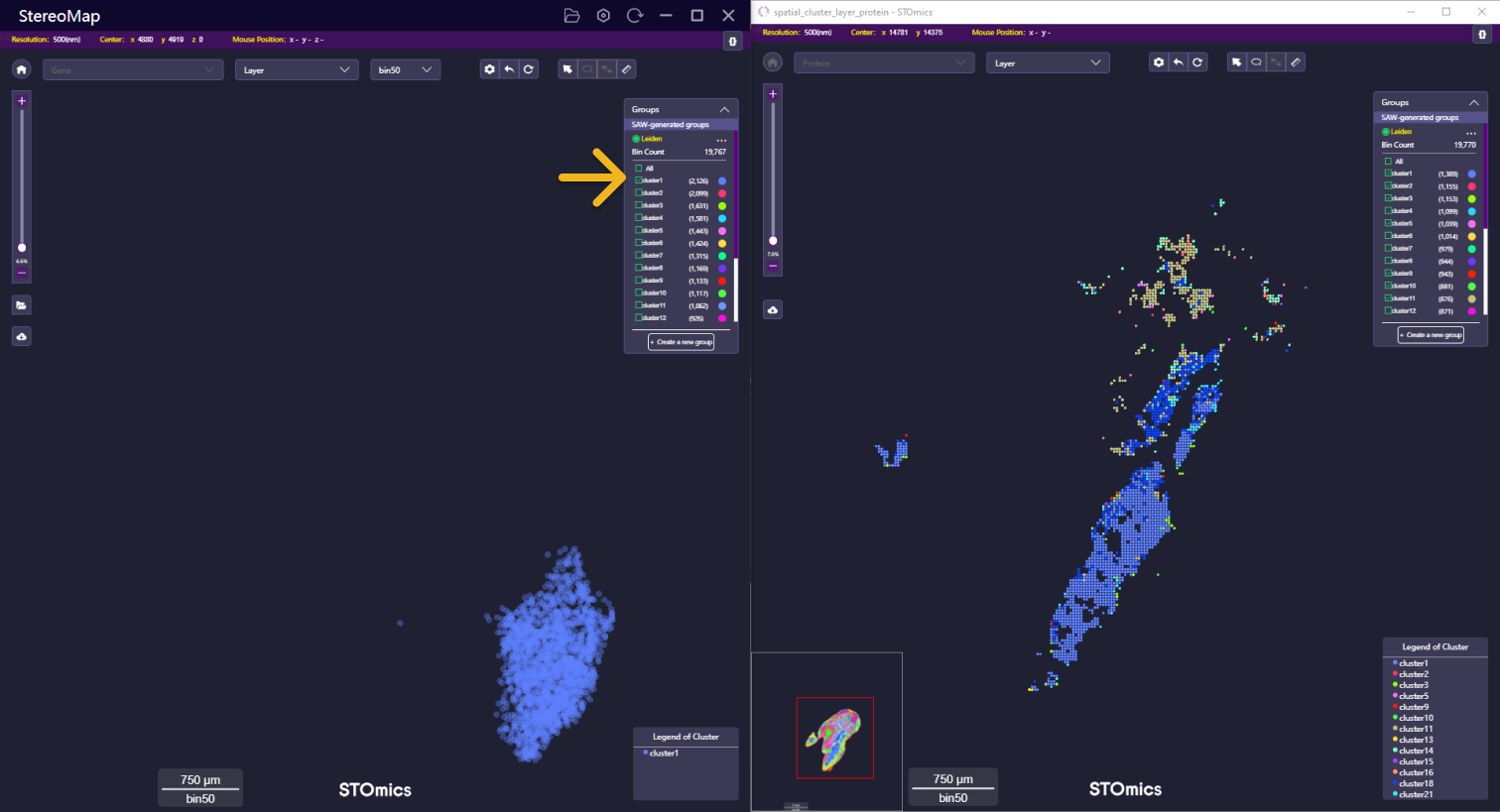
Lasso 某个区域联动
Lasso 功能的联动目前仅适用于空间分布的图像中,UMAP 或任何其他空间视图中的点无法联动。
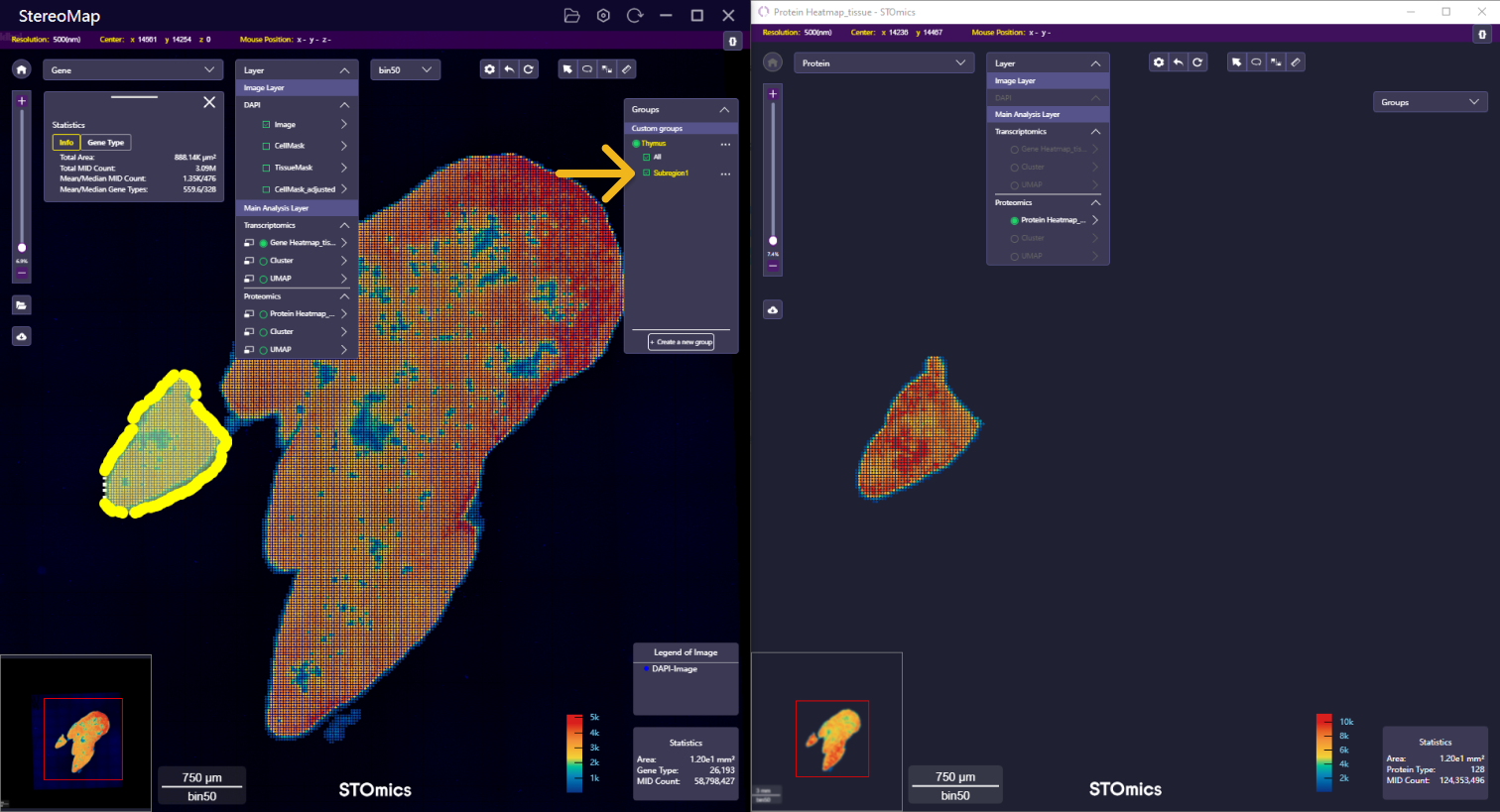
除了可以选择某个 cluster 可以联动外,您还可以使用 lasso 工具选择感兴趣的特定区域。保存套索区域后,您可以切换到任意的空间图层做联动,包括基因热图、蛋白质热图、基因的 cluster 和蛋白的 cluster。
蛋白与其对应标记基因的空间分布
此功能仅适用于square bin,例如:bin20, 50, 100等
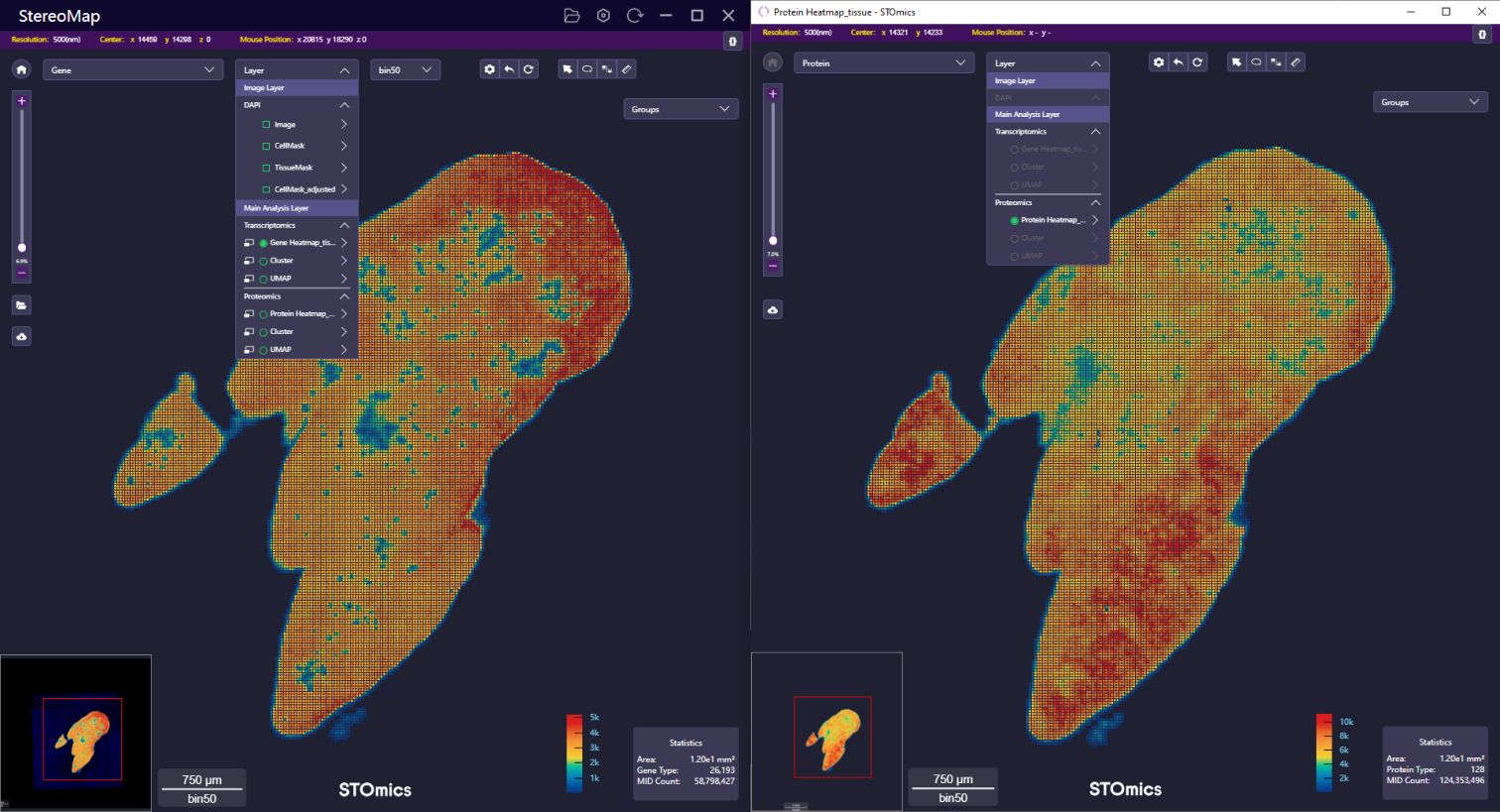
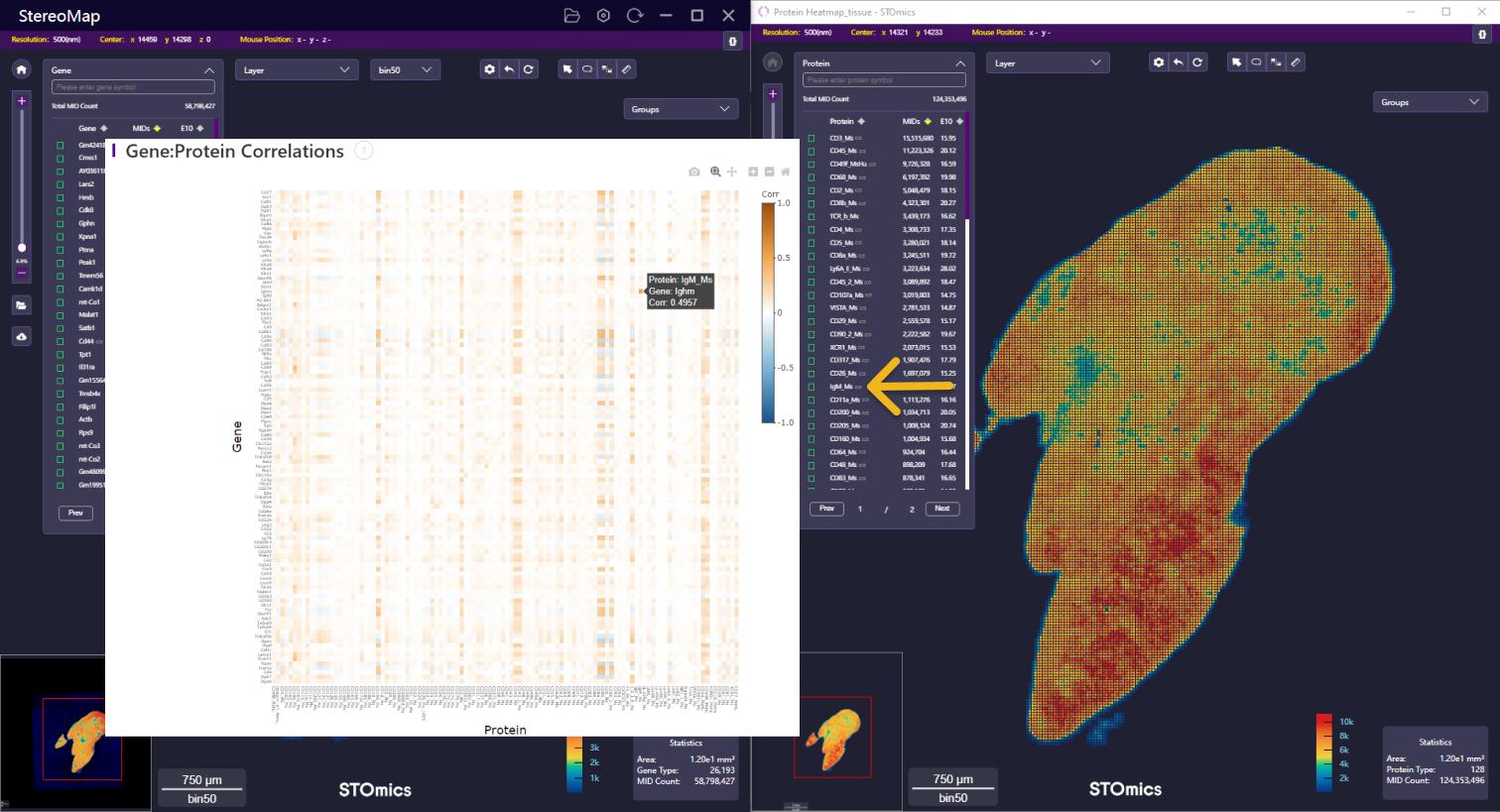
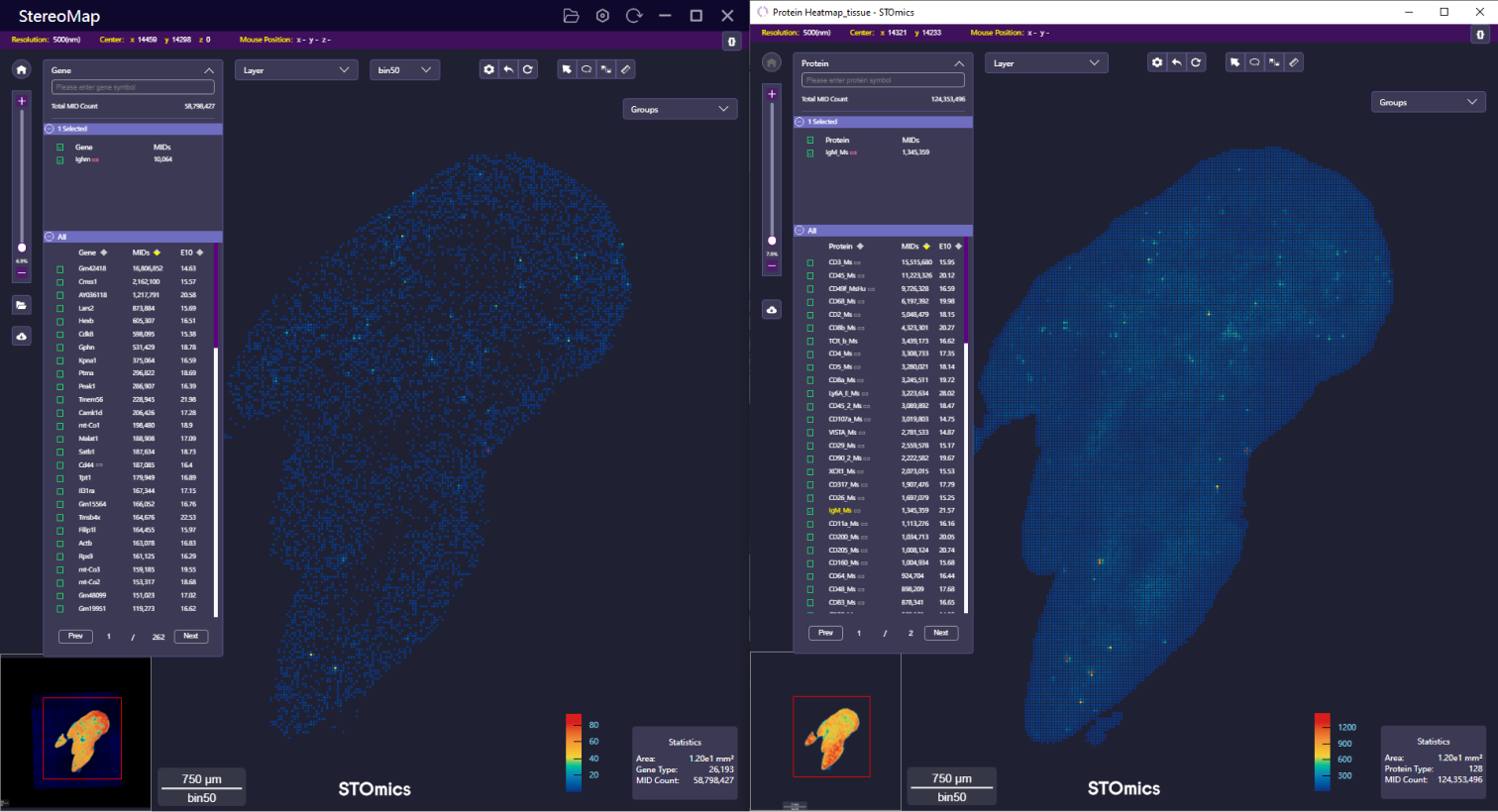
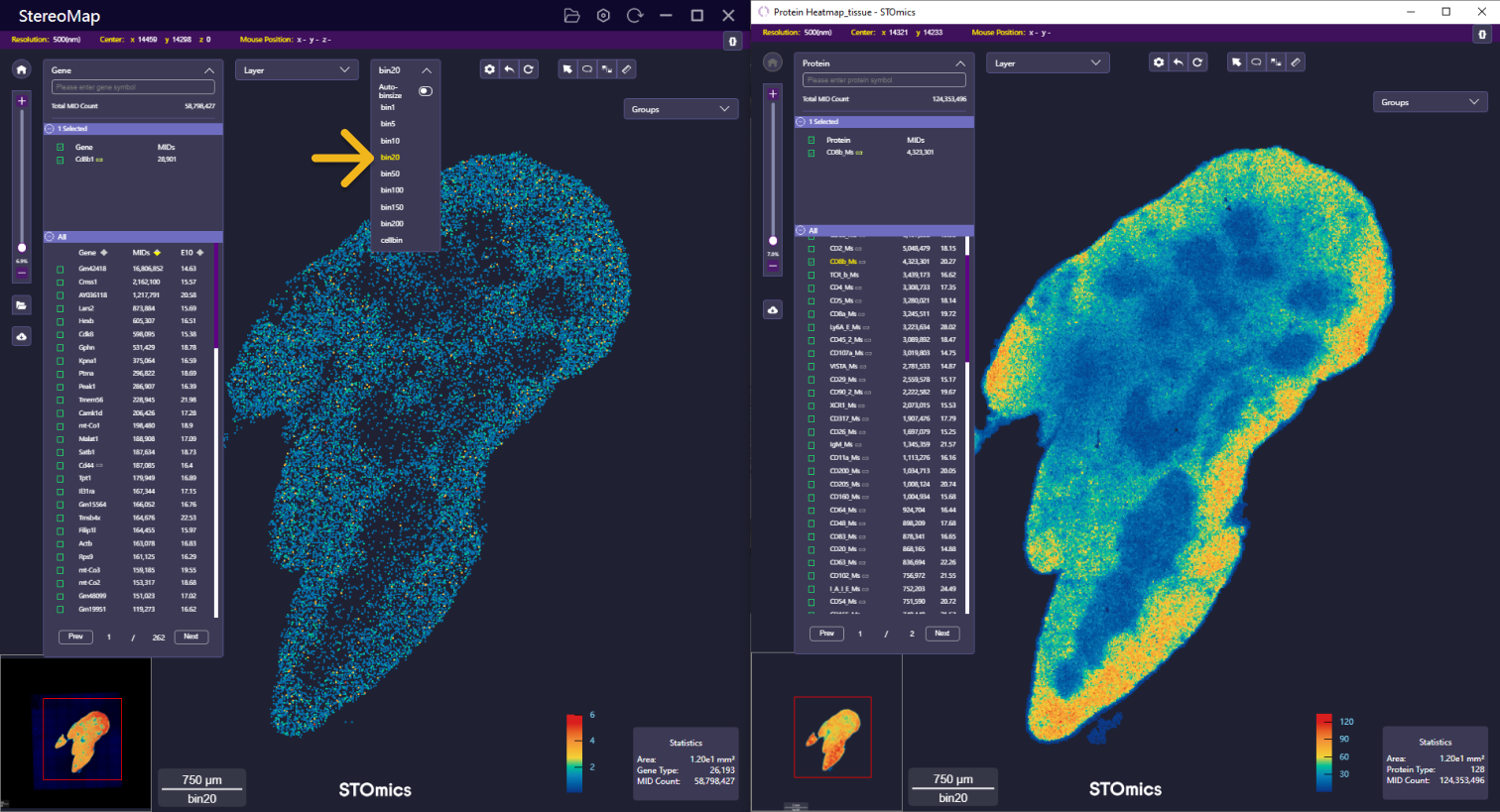
对于 Stereo-CITE 数据分析,SAW (>= 8.1)的 HTML 分析报告中展示了基因和蛋白的相关性,可以参考 SAW 用户手册的运行分析-输出结果-HTML Report - Gene : protein correlations 章节获取更多信息。在 StereoMap (>= 4.1) 中,您可以查看蛋白与其标记基因对的空间分布以便更好地了解相关性。
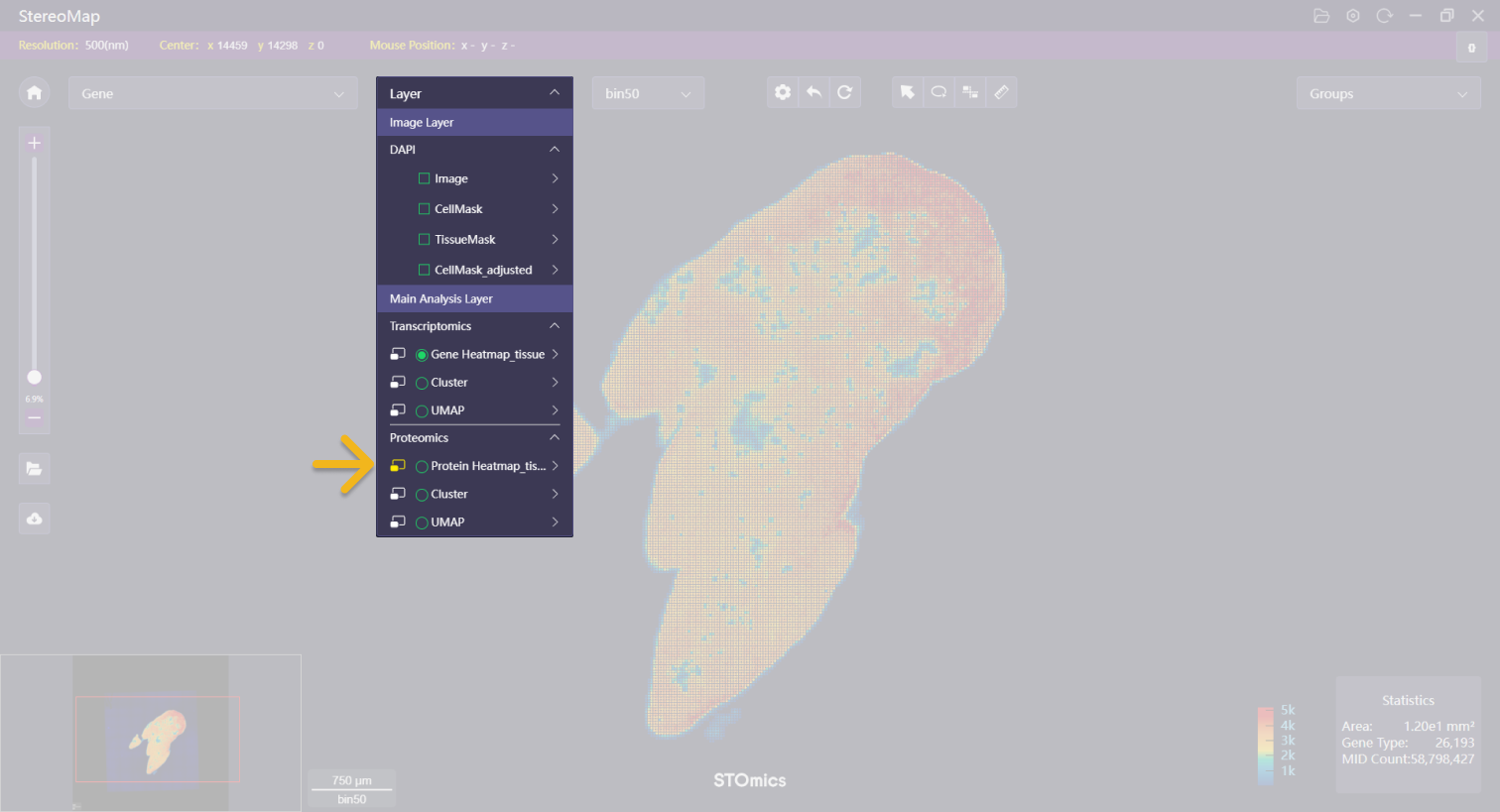
您需要打开 Feature 菜单同时查看蛋白热图和基因热图。单击Protein_Heatmap_tissue 前面的 ,在新的子窗口中打开蛋白的热图。这样您就可以并排查看两个不同组学的热图。同时您也可以先查看蛋白热图,然后在新的子窗口中打开基因热图。
,在新的子窗口中打开蛋白的热图。这样您就可以并排查看两个不同组学的热图。同时您也可以先查看蛋白热图,然后在新的子窗口中打开基因热图。
从 SAW HTML 报告中可以看出,蛋白质 IgM_Ms 和其标记基因 Ighm 呈现正相关性(Pearson's r = 0.4957),与先验知识一致 [1]。在 StereoMap 中,您可以点击基因/蛋白名称后面的

如果您想查看多个基因与蛋白的联动,您可以在面板中单击基因或者蛋白名称前面的
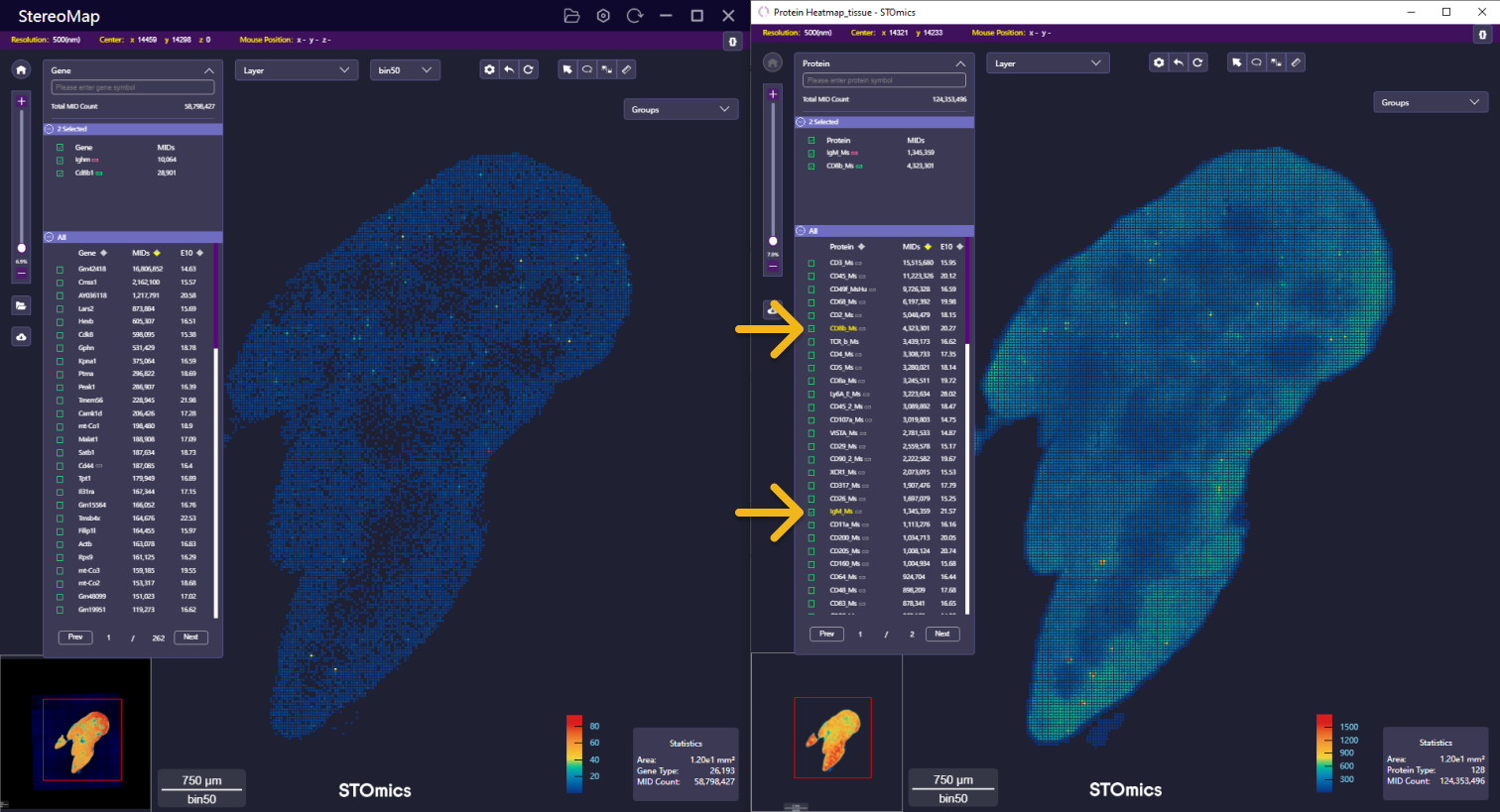
多个基因与蛋白的联动
如果您选择的 binszie 过大可能无法看清楚表达的 pattern,您可以在主窗口切换 binsize,对应的子窗口会同步切换。
参考附录
- Niu, X., Swett, A. D., Sotelo, J., Jiao, M. S., Stewart, C. M., Potenski, C., Mielinis, P., Roelli, P., Stoeckius, M., & Landau, D. A. (2023). Integration of whole transcriptome spatial profiling with protein markers. Nature Biotechnology, 41(6), 788-793. https://doi.org/10.1038/s41587-022-01536-3