Stereo-seq T FF
检查图像的配准
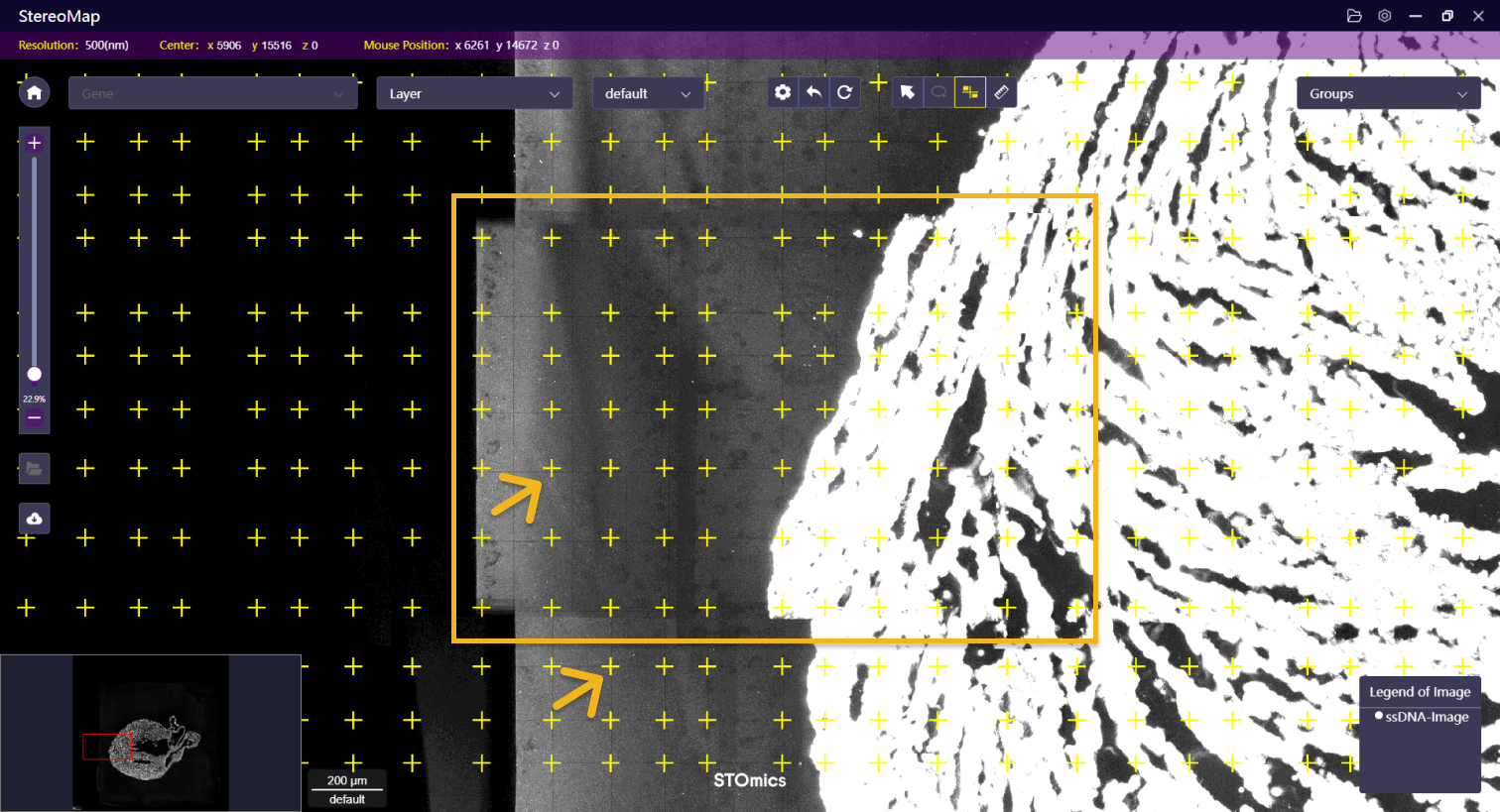
芯片表面的轨迹线在空间表达矩阵热图上展示为较窄的线条,可将其作为参考辅助图像配准。强烈建议用户放大图像的组织边缘,通过查看轨迹线是否与图像上的暗线重叠,确定图像的配准效果。
检查是否对齐的一个小窍门,首先检查两个对角的区域,如果这两个区域的轨迹线跟图像的暗线完全重合,那么整体情况很可能是相对较好的。
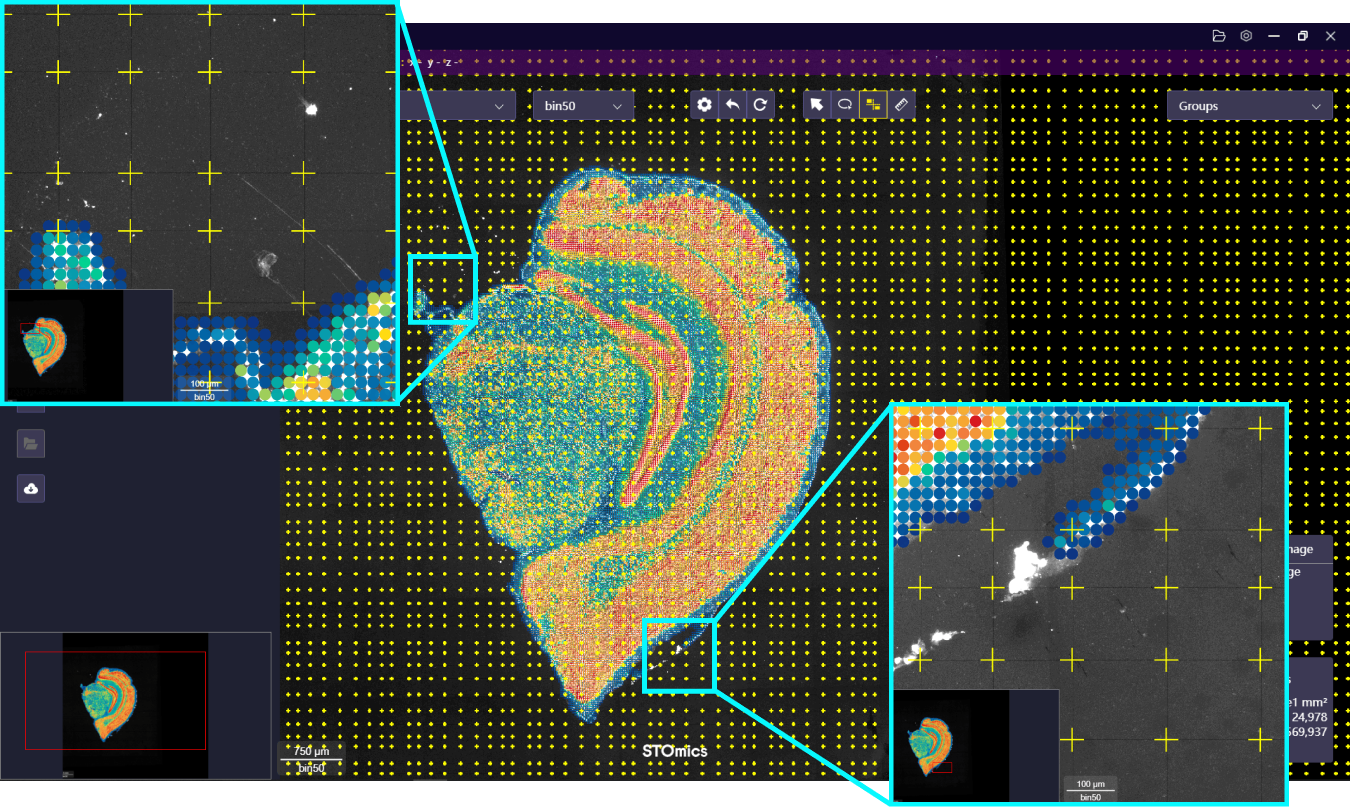
如果大部分轨迹线不重叠,您将需要手动重新对图像做配准。具体操作步骤可参考图像处理指南。
基因共表达分析
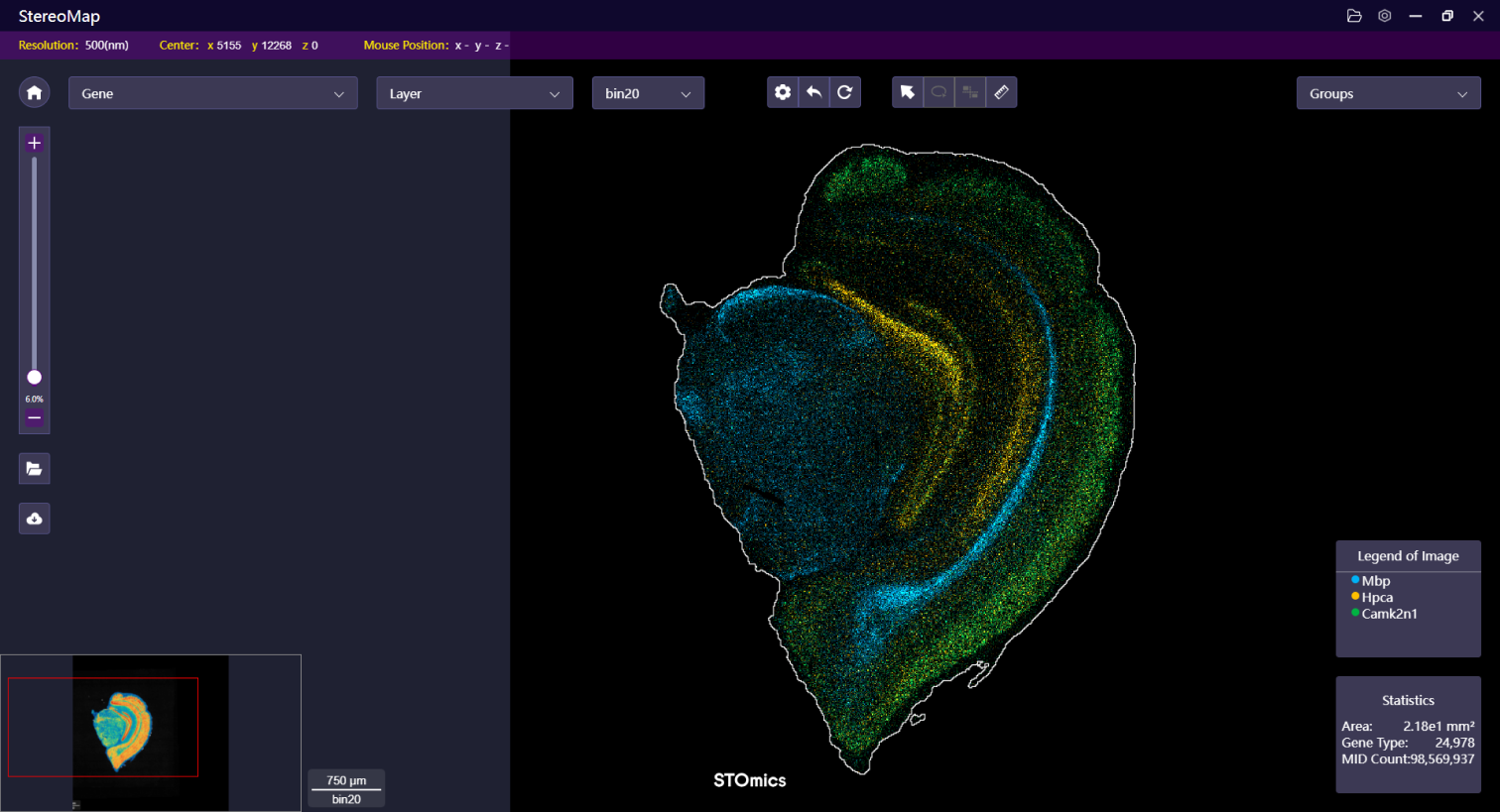
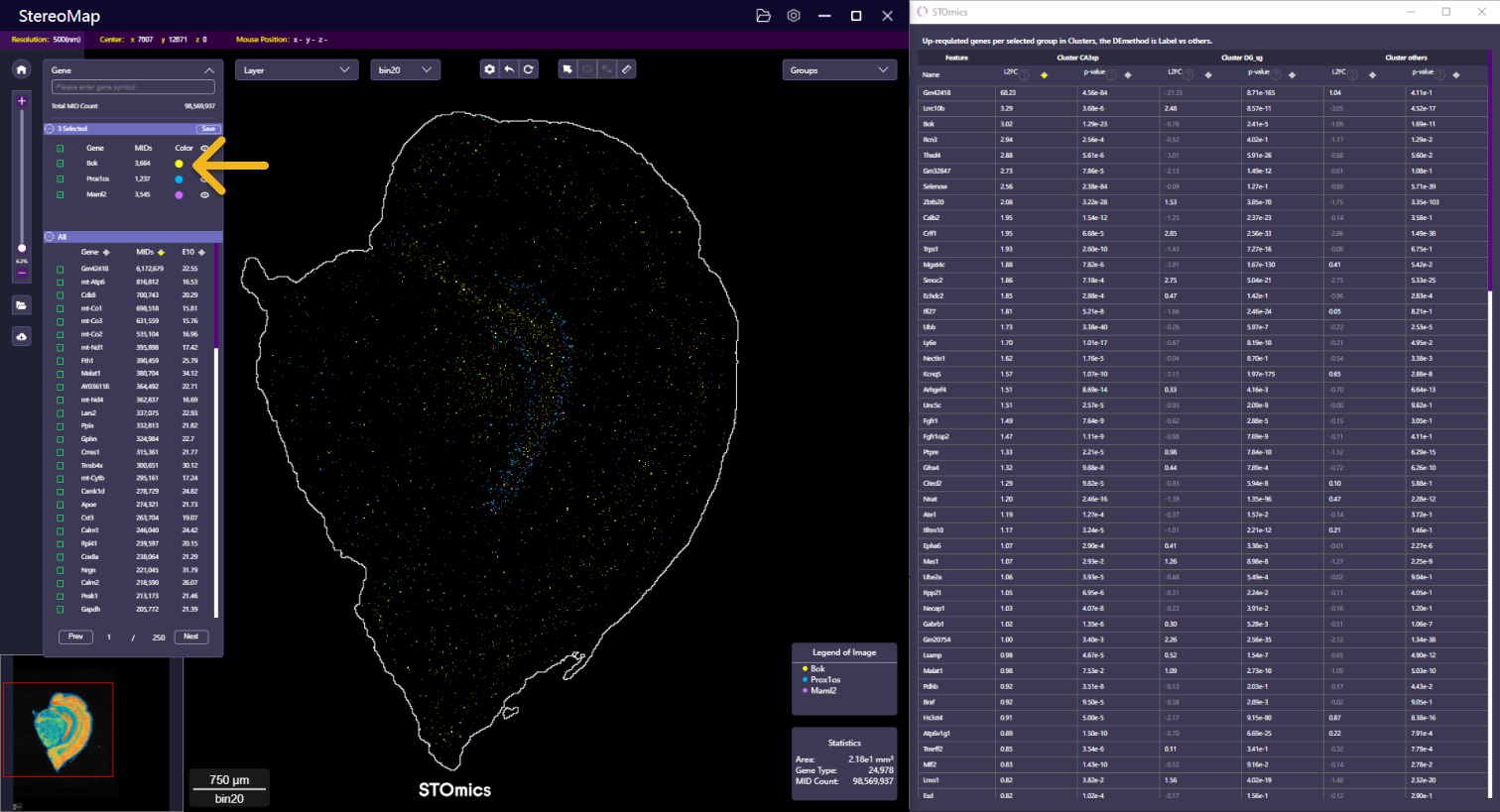
您可以使用不同的颜色来可视化不同的基因,分析基因的共表达。
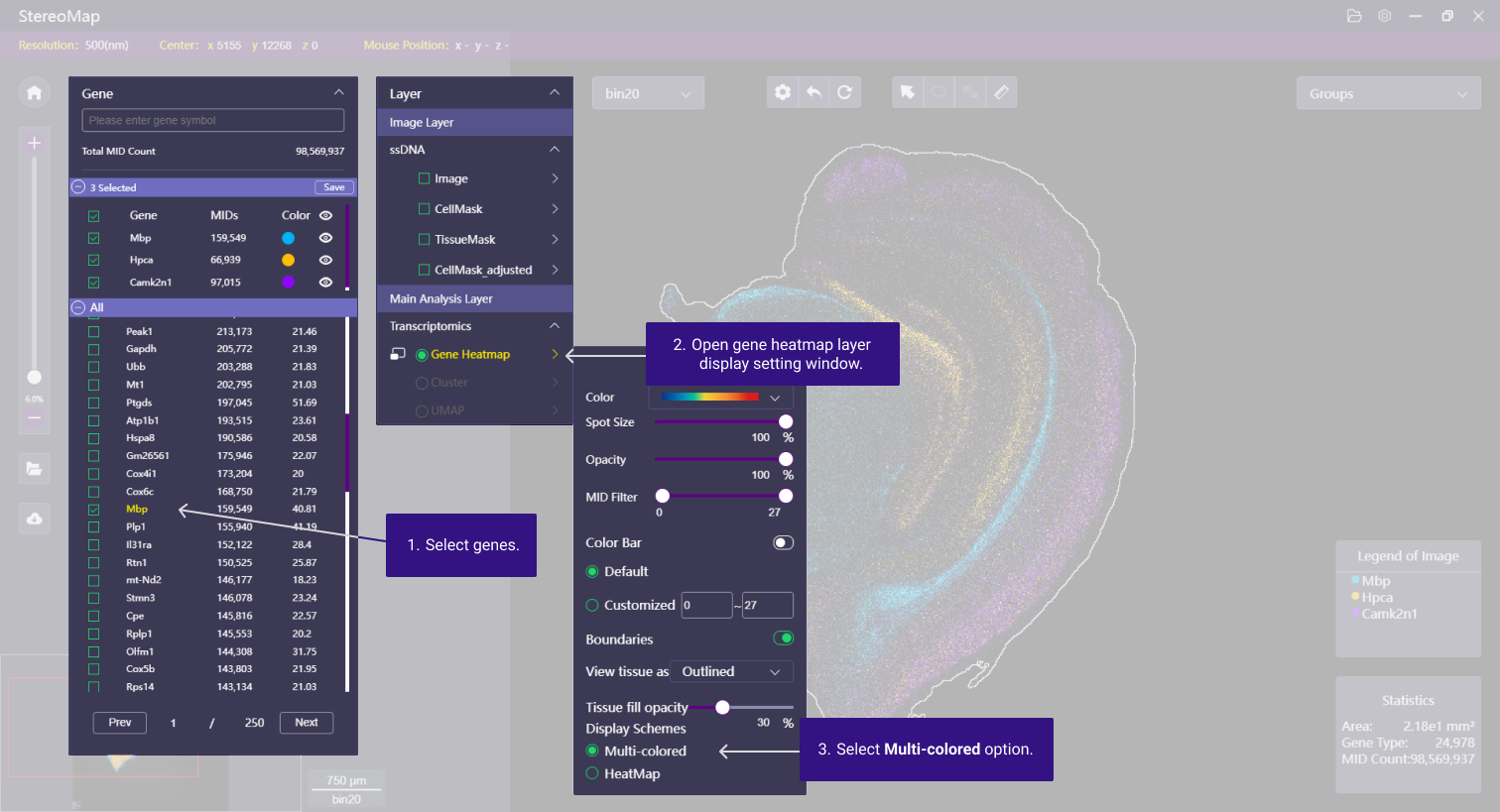
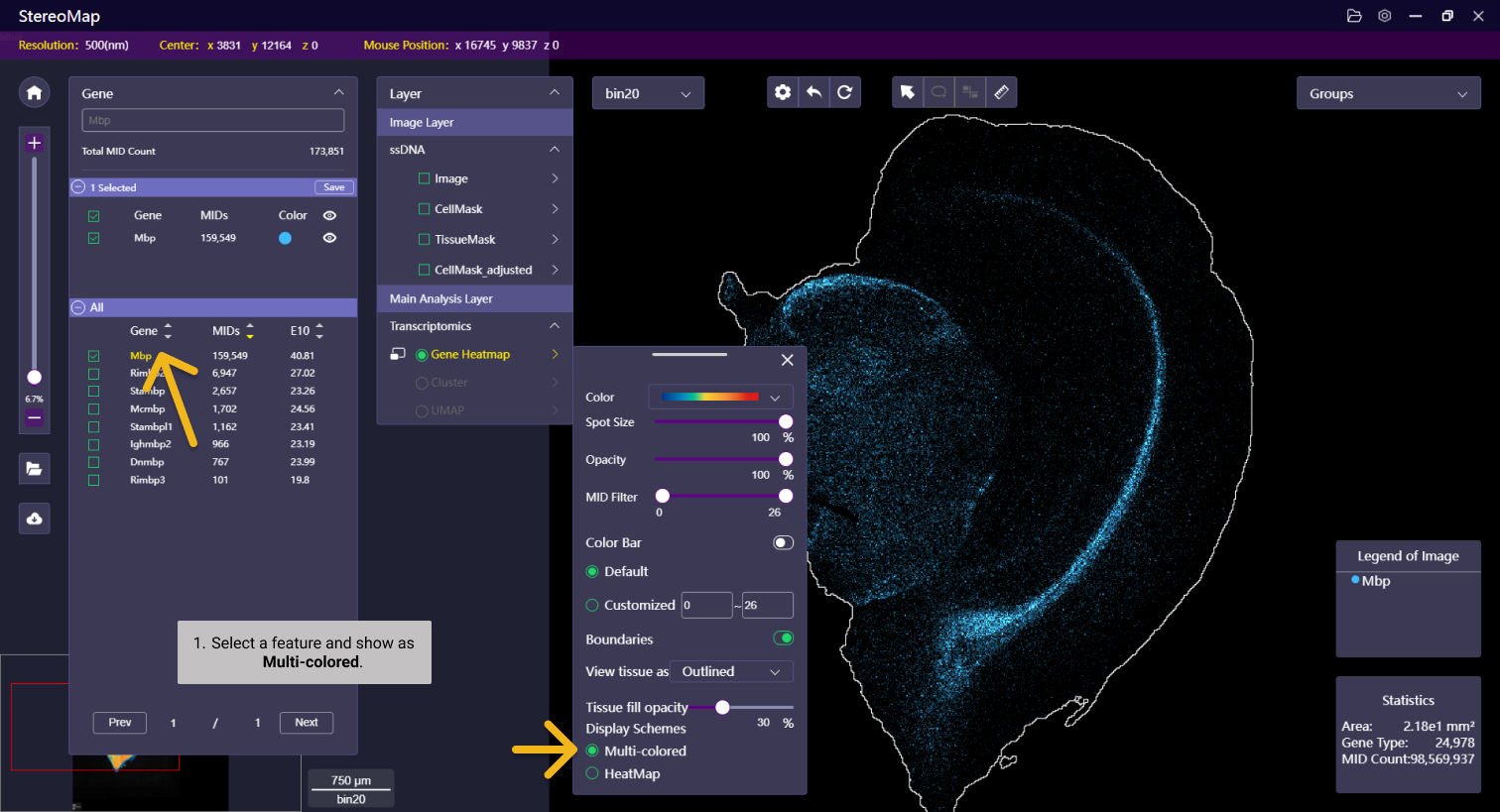
首先,在面板中选择感兴趣的基因,画布将展示该基因对应的表达热图。
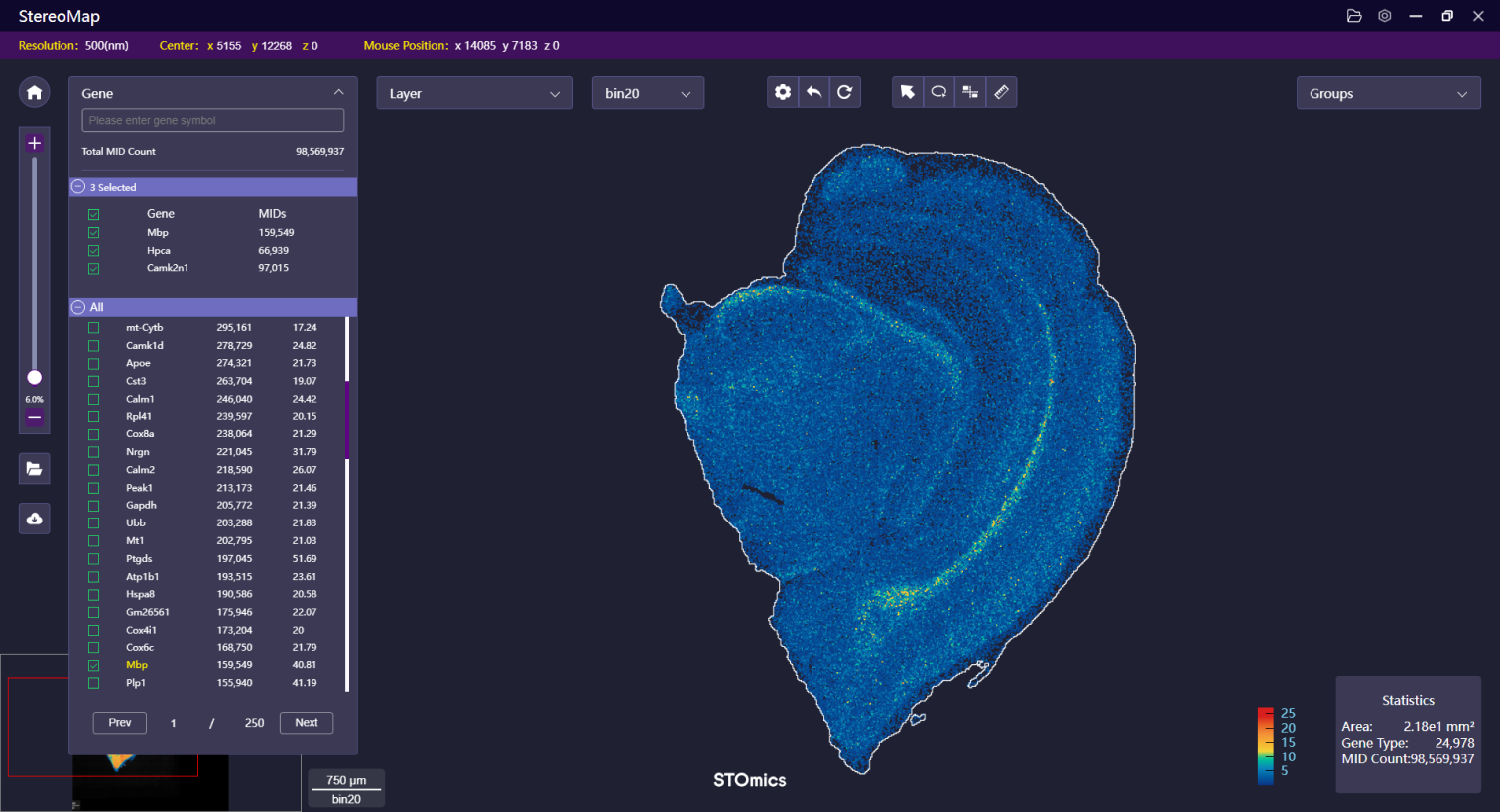
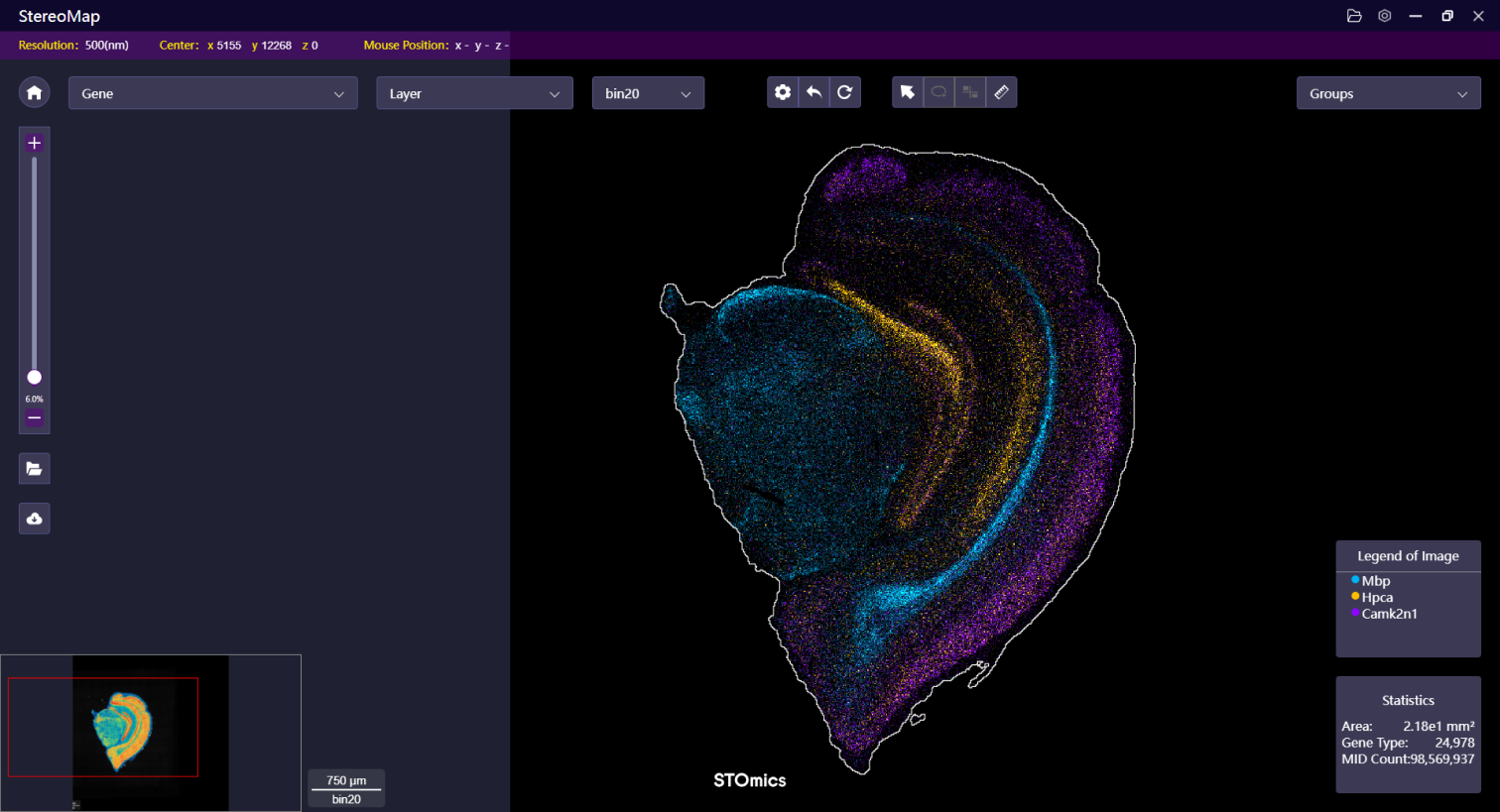
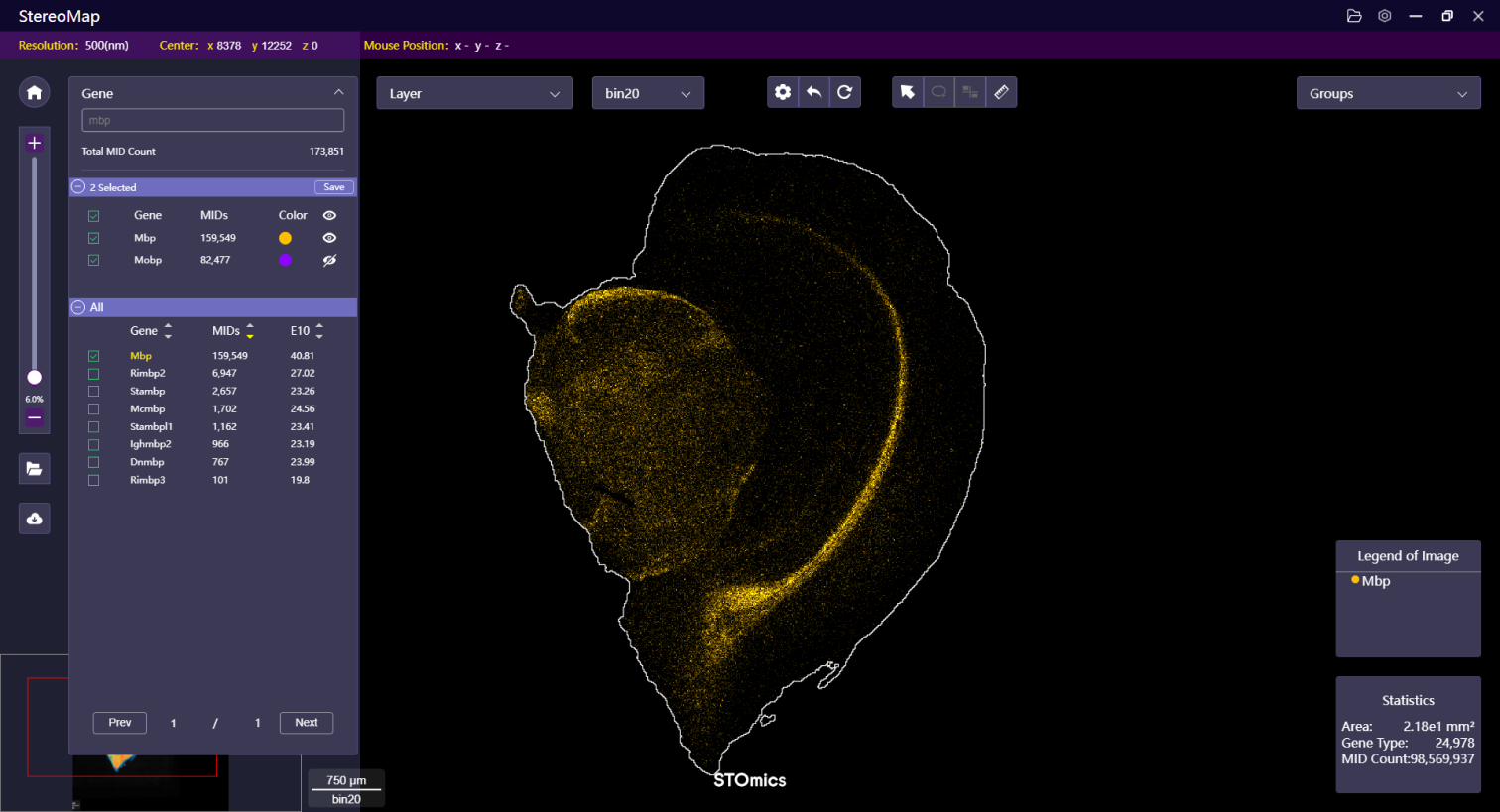
其次,点击 Layer menu 菜单,在 Display Schemes 下选择 Multi-colored ,就可以比较您所选择的基因是否存在共表达。说明:下图已选定的基因在该组织中没有共表达。
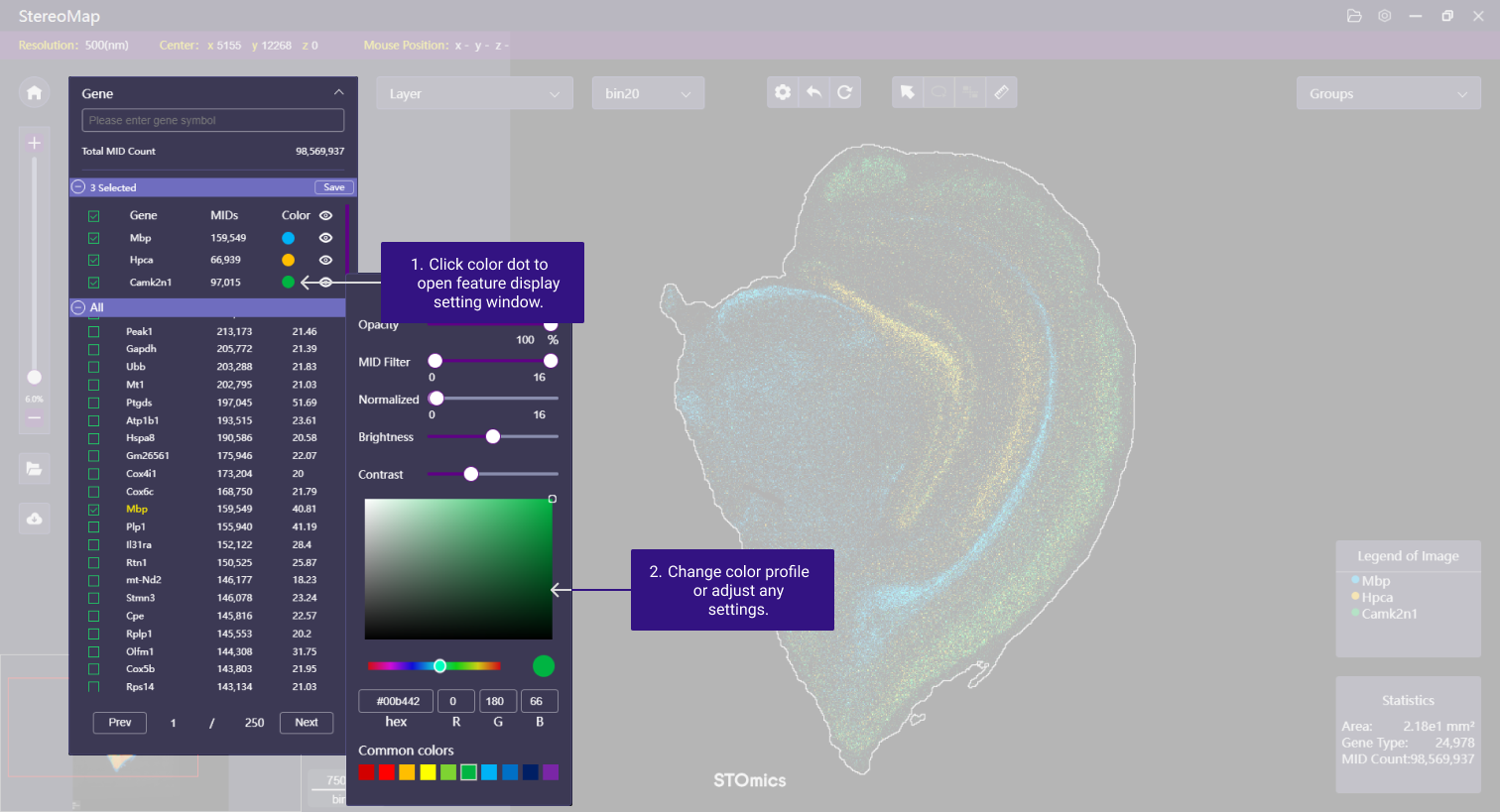
如果对于系统默认分配的颜色或者显示设置不满意,可以点击所选基因对应拾色器上的小圆圈/彩虹带,打开图层设置面板做调整。
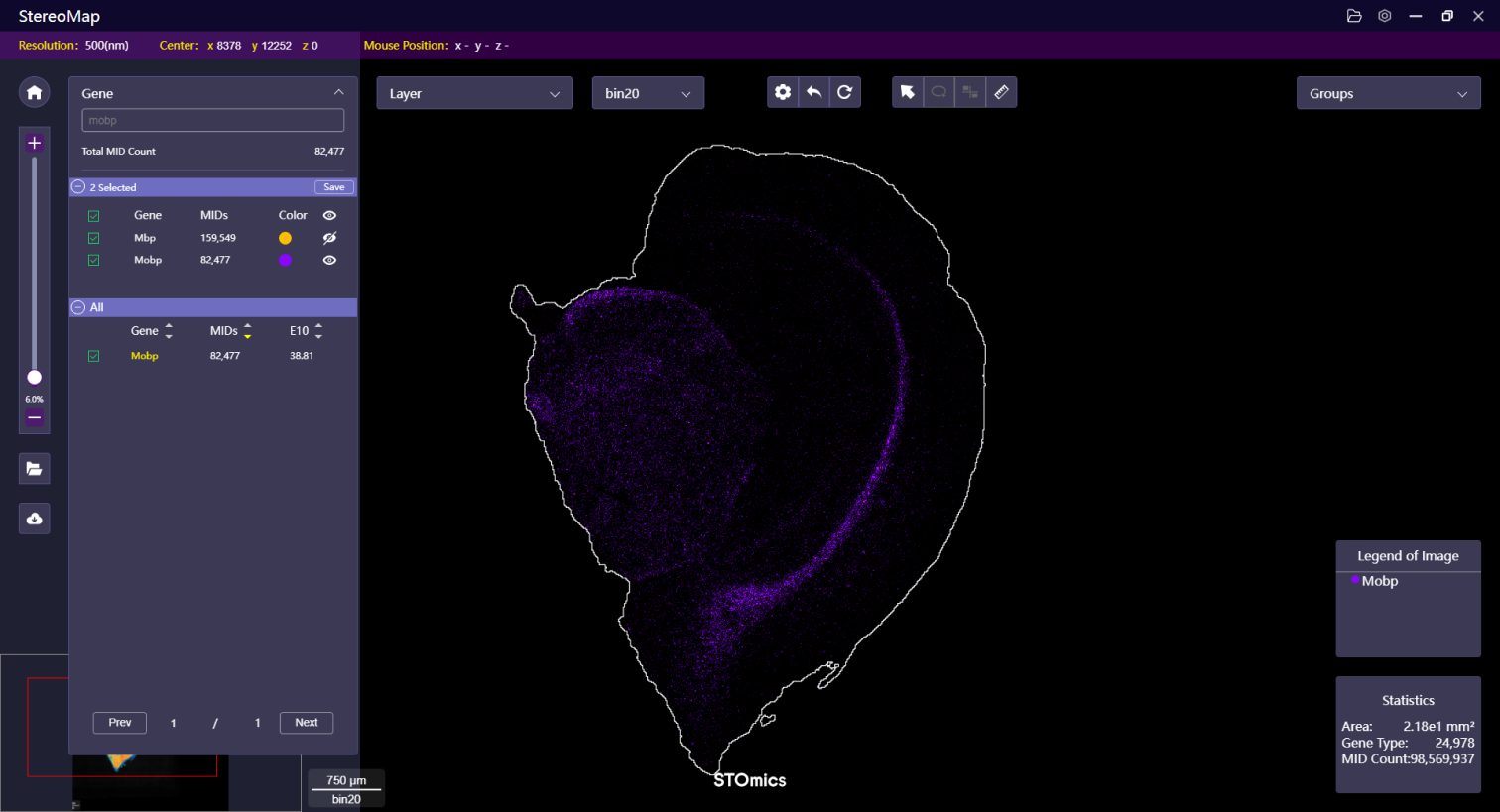
与之前的选择不同,这次我们选择了两个(黄色和紫色)共表达的基因如下图所示。
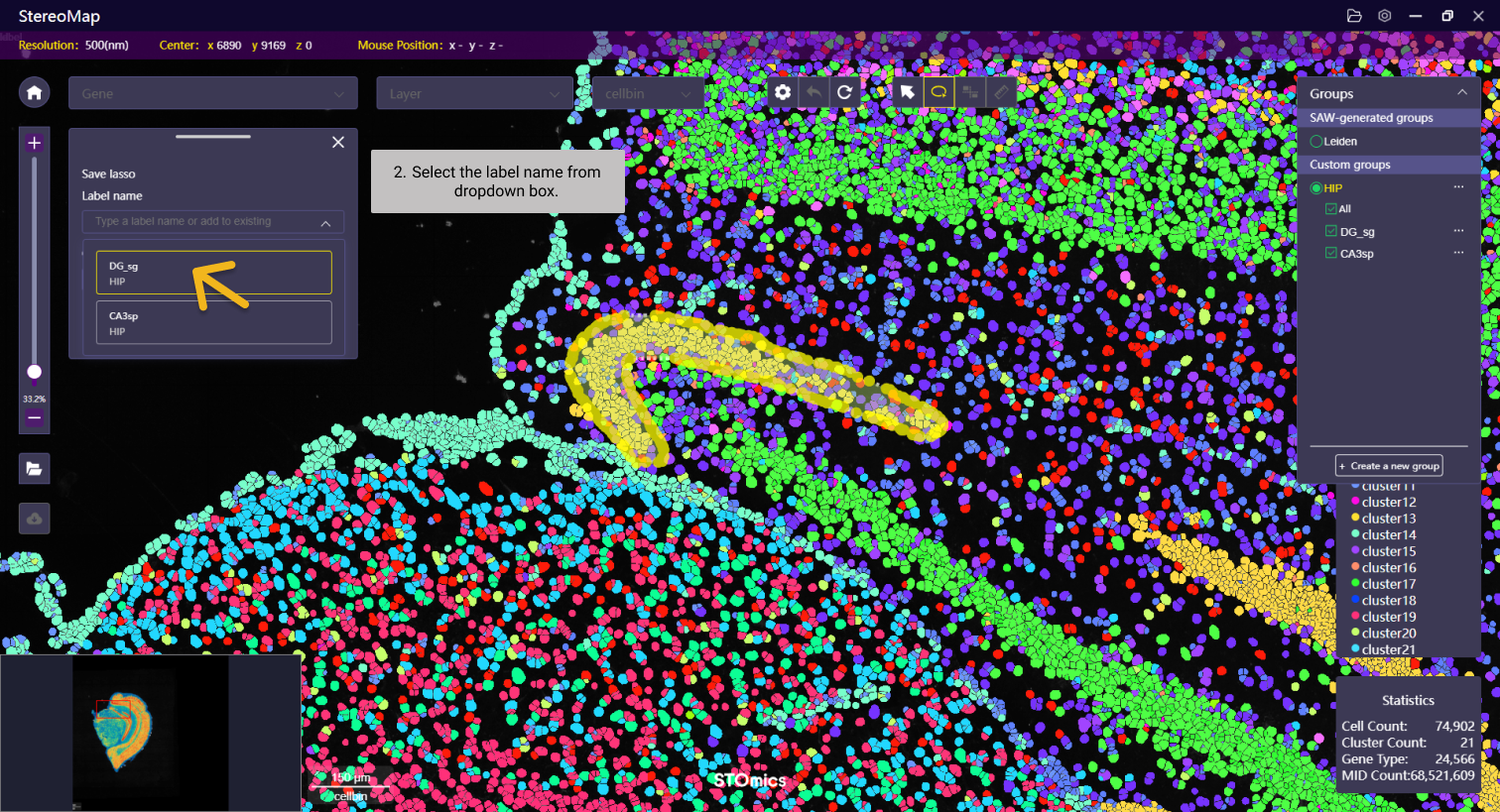
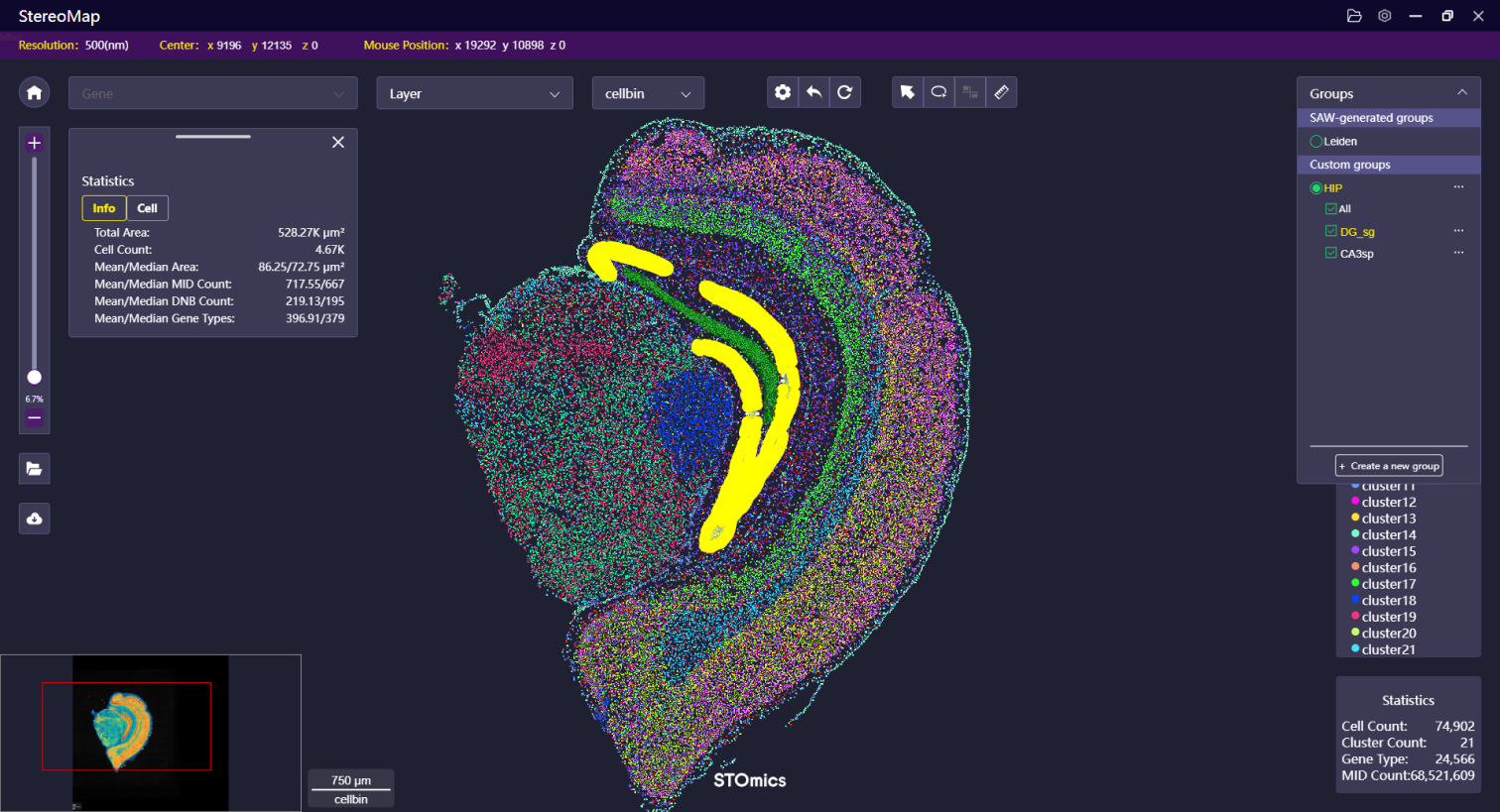
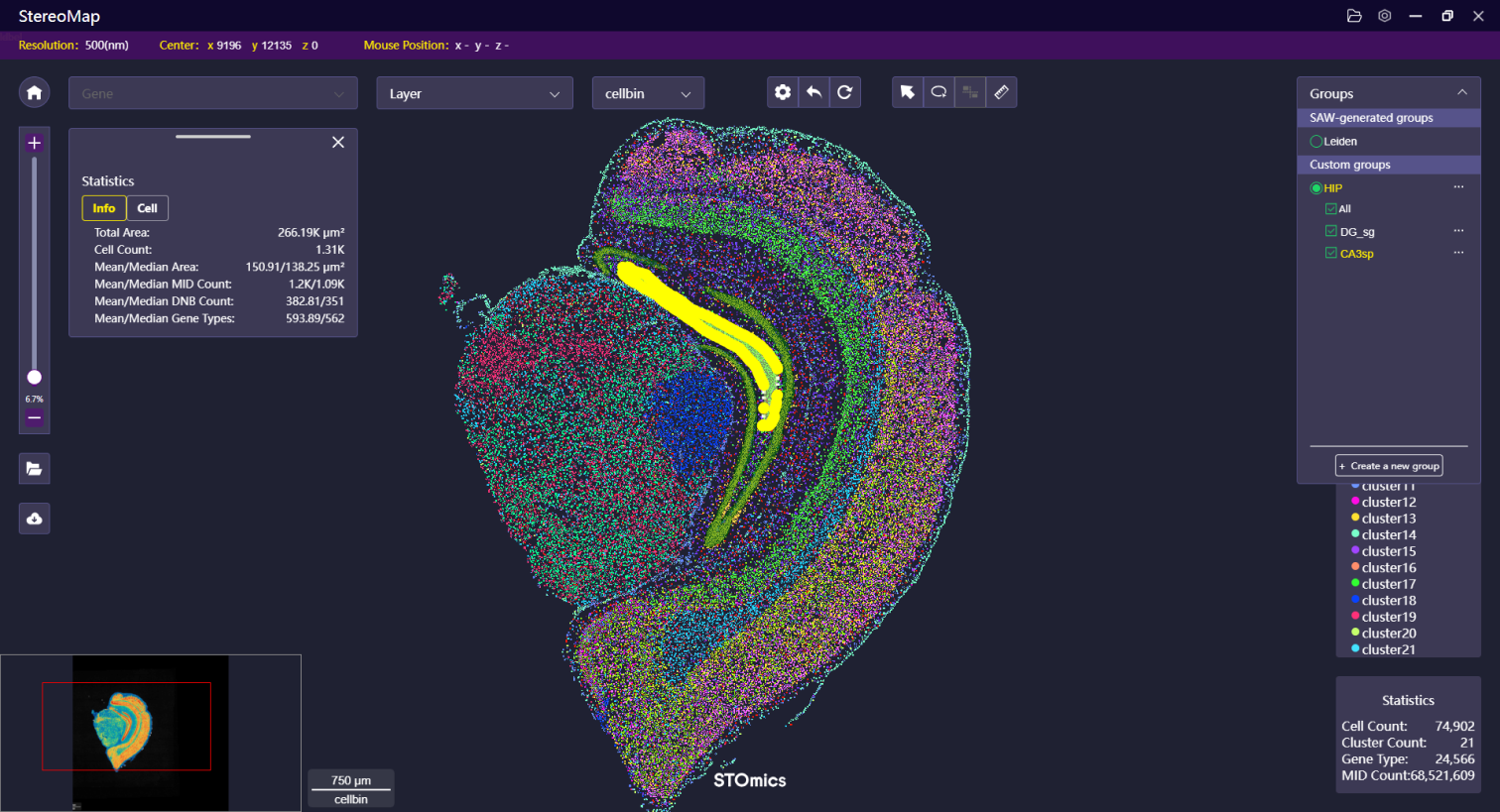
标注区域并生成新的热图
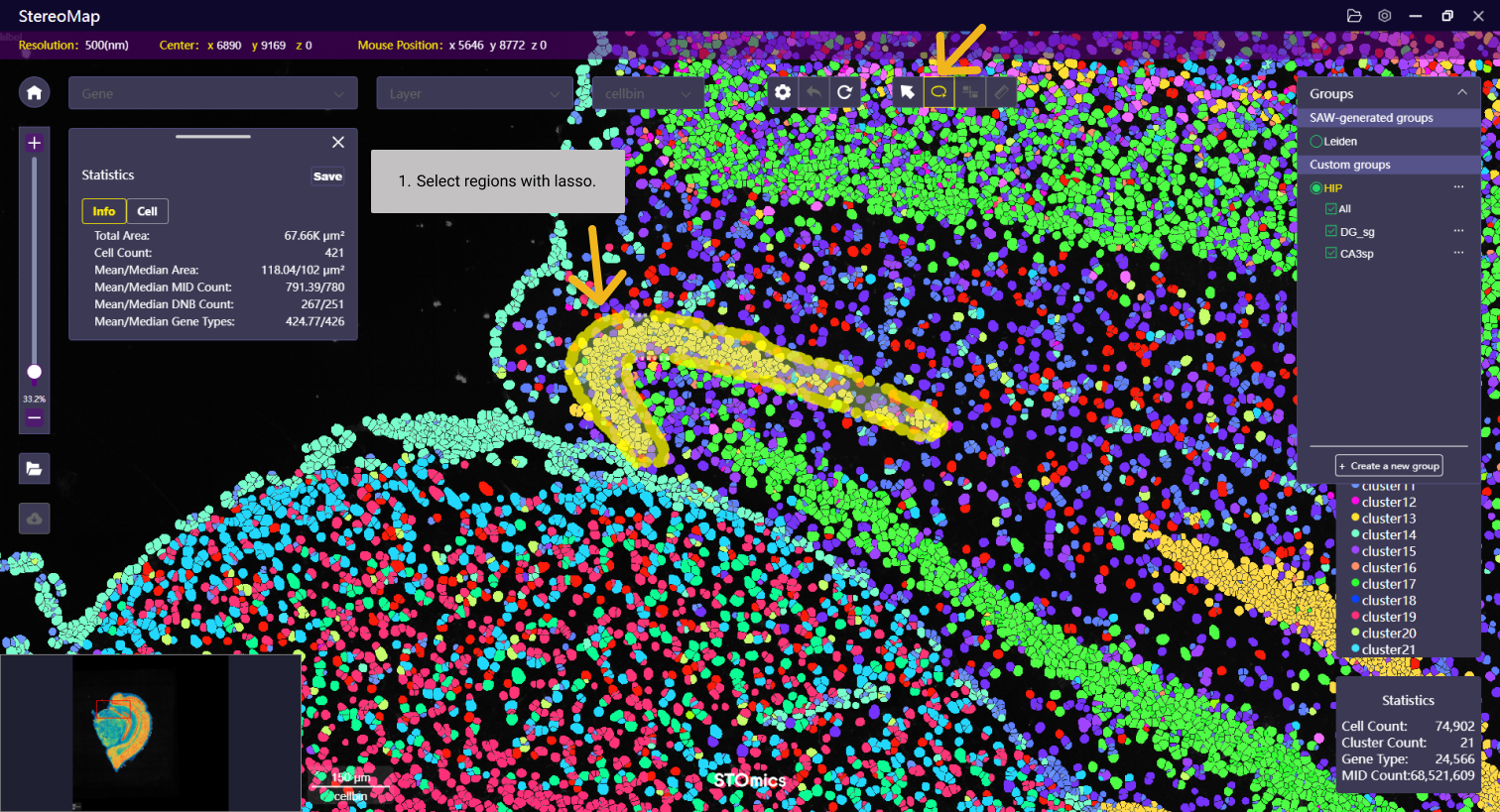
对组织样本中感兴趣区域的识别与分析,lasso 是一个很好用的工具,您可以在组织样本中手动勾画该区域,并将这些分散或者连续的区域归属在同一个组中。
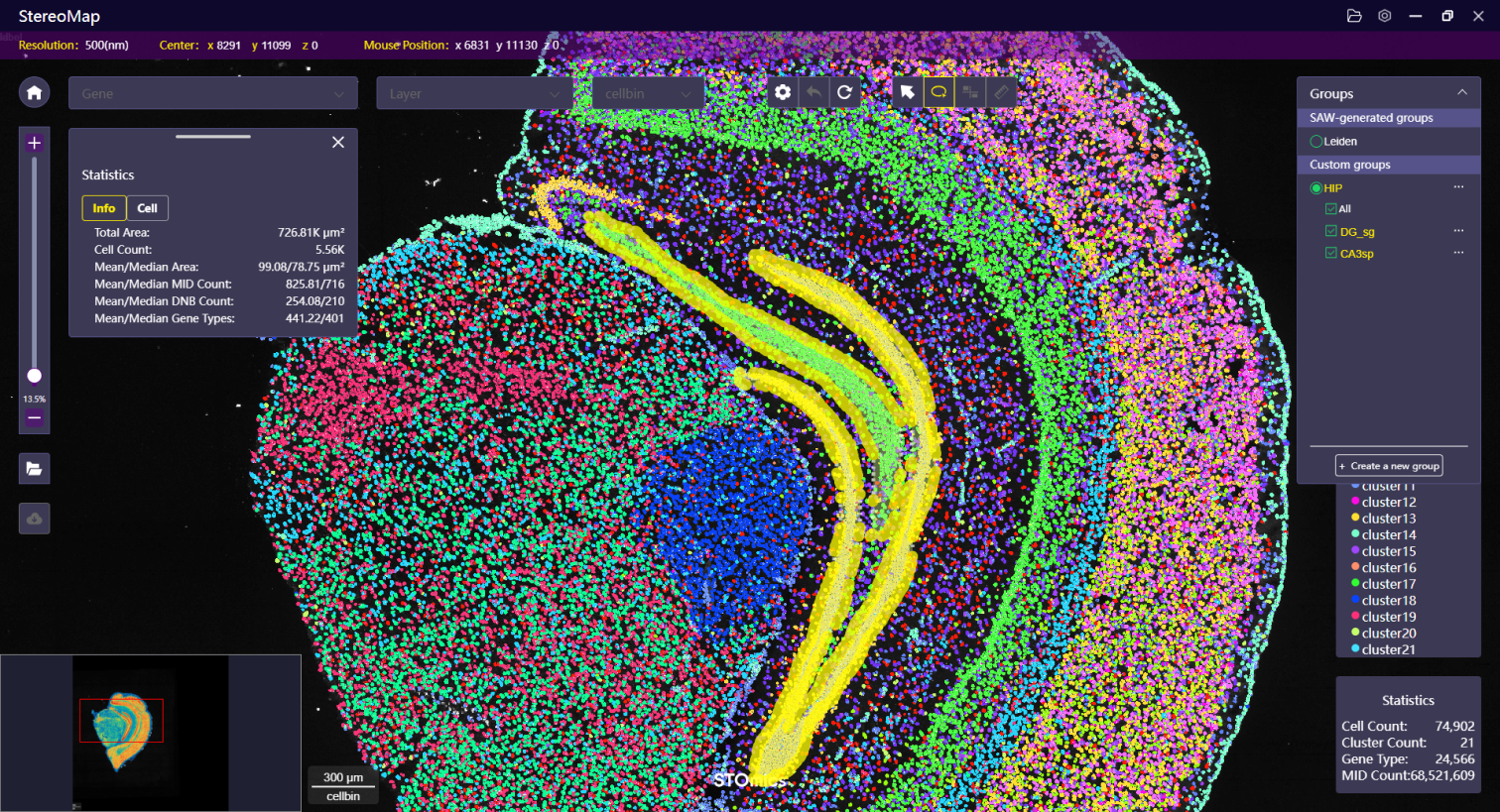
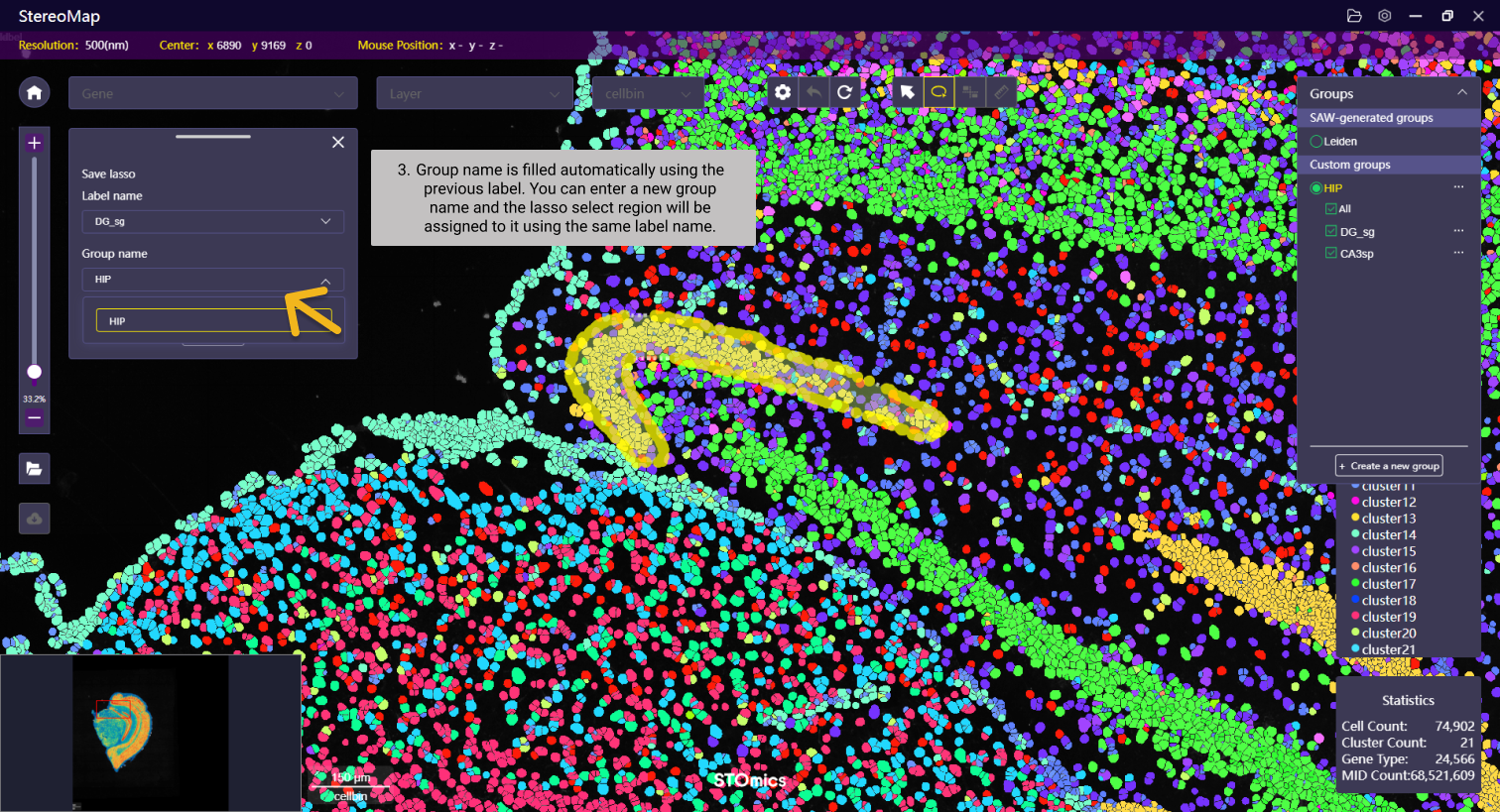
如果您需要将某个区域增加到已知的 Label 中,可以简单地使用相同的 Label 名称来命名,系统将自动合并两个区域。
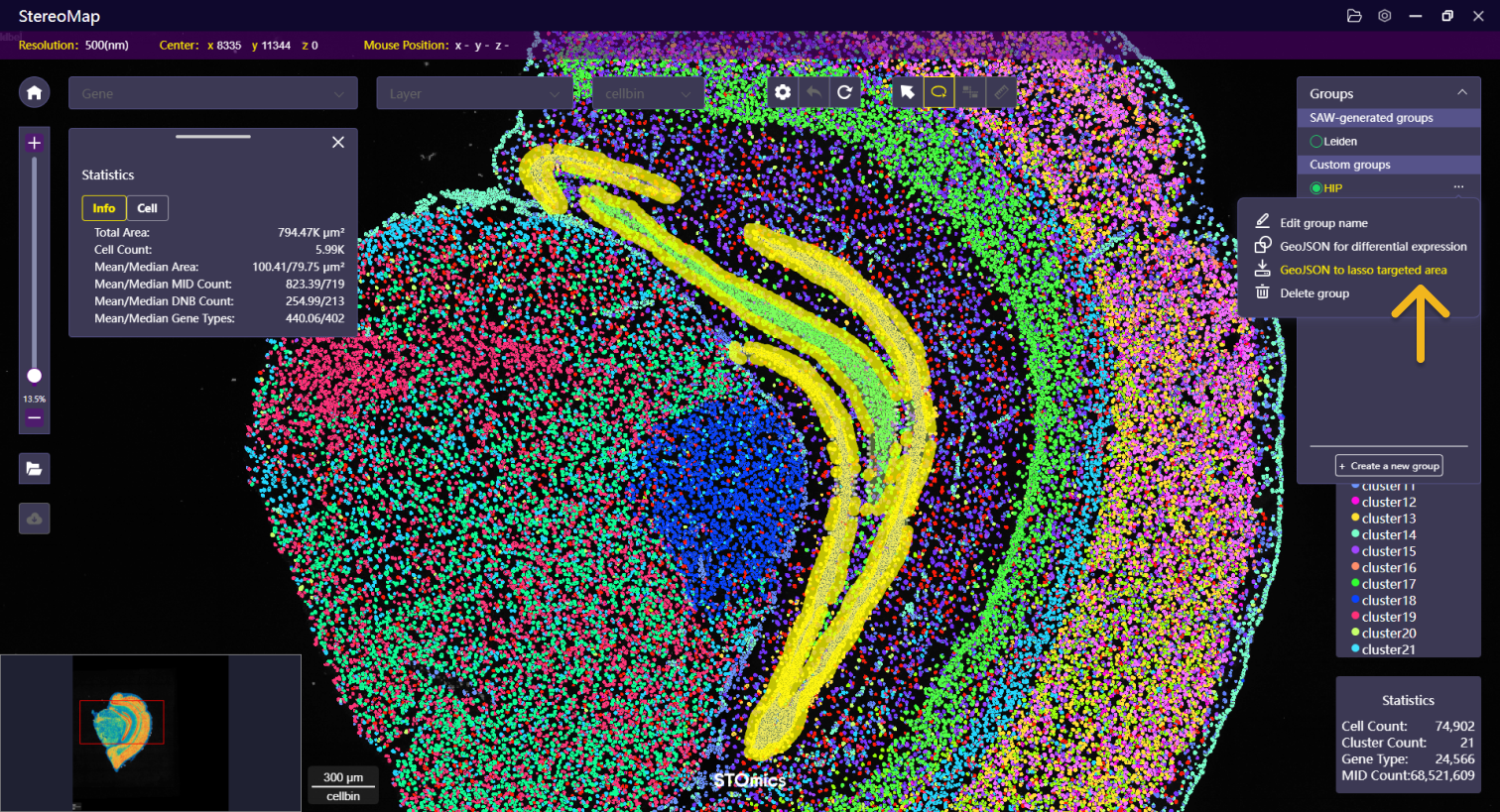
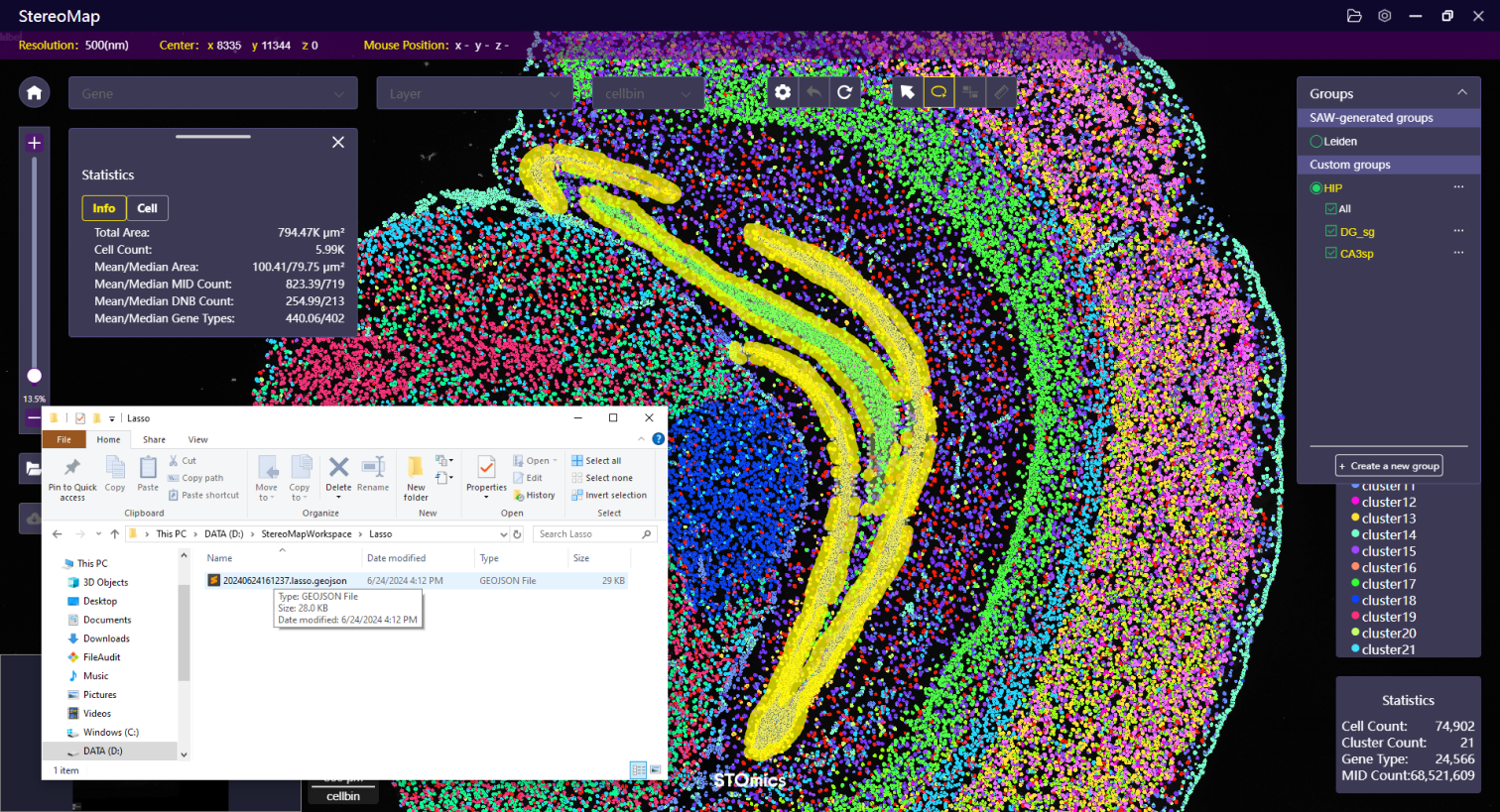
套索完成后,可以将坐标信息保存并传递给 SAW reanalyze lasso 重新分析,以获取所选区域的空间特征表达矩阵。单击Group 或 Label 名称右侧的按钮 ,选择 GeoJSON to lasso targeted area 导出套索的 GeoJSON 文件。选择后您的资源管理器将会打开,您可以选择想要保存输出的
,选择 GeoJSON to lasso targeted area 导出套索的 GeoJSON 文件。选择后您的资源管理器将会打开,您可以选择想要保存输出的 YYYYMMDDHHMMSS.lasso.geojson 文件的目录。
可以将套索后 GeoJSON 文件路径通过 --lasso-geojson 参数传递给 SAW reanalyze lasso流程,生成套索区域的空间特征表达矩阵。
为满足普通 bin 或 cell bin 下的计算,套索后的 GeoJSON 文件存储的是该区域下轮廓的坐标,而不是点的信息。
如果要在普通 bin 下生成新的矩阵,您可以在 --gef 参数下输入 .gef 文件的路径,并使用 --bin-size 指定 bin 的大小。
saw reanalyze lasso \
--gef=/path/to/input/GEF \
--lasso-geojson=/path/to/lasso/YYYYMMDDHHMMSS.lasso.geojson \
--bin-size=1,20,50,100,200 \
--output=/path/to/output/folder
如果要在 cellbin 下生成新的矩阵,您可以在 --cellbin-gef 参数下输入 .cellbin.gef 文件的路径。
saw reanalyze lasso \
--cellbin-gef=/path/to/input/cellbin/GEF \
--lasso-geojson=/path/to/lasso/YYYYMMDDHHMMSS.lasso.geojson \
--output=/path/to/output/folder
新生成的矩阵可用于后续进一步的分析。
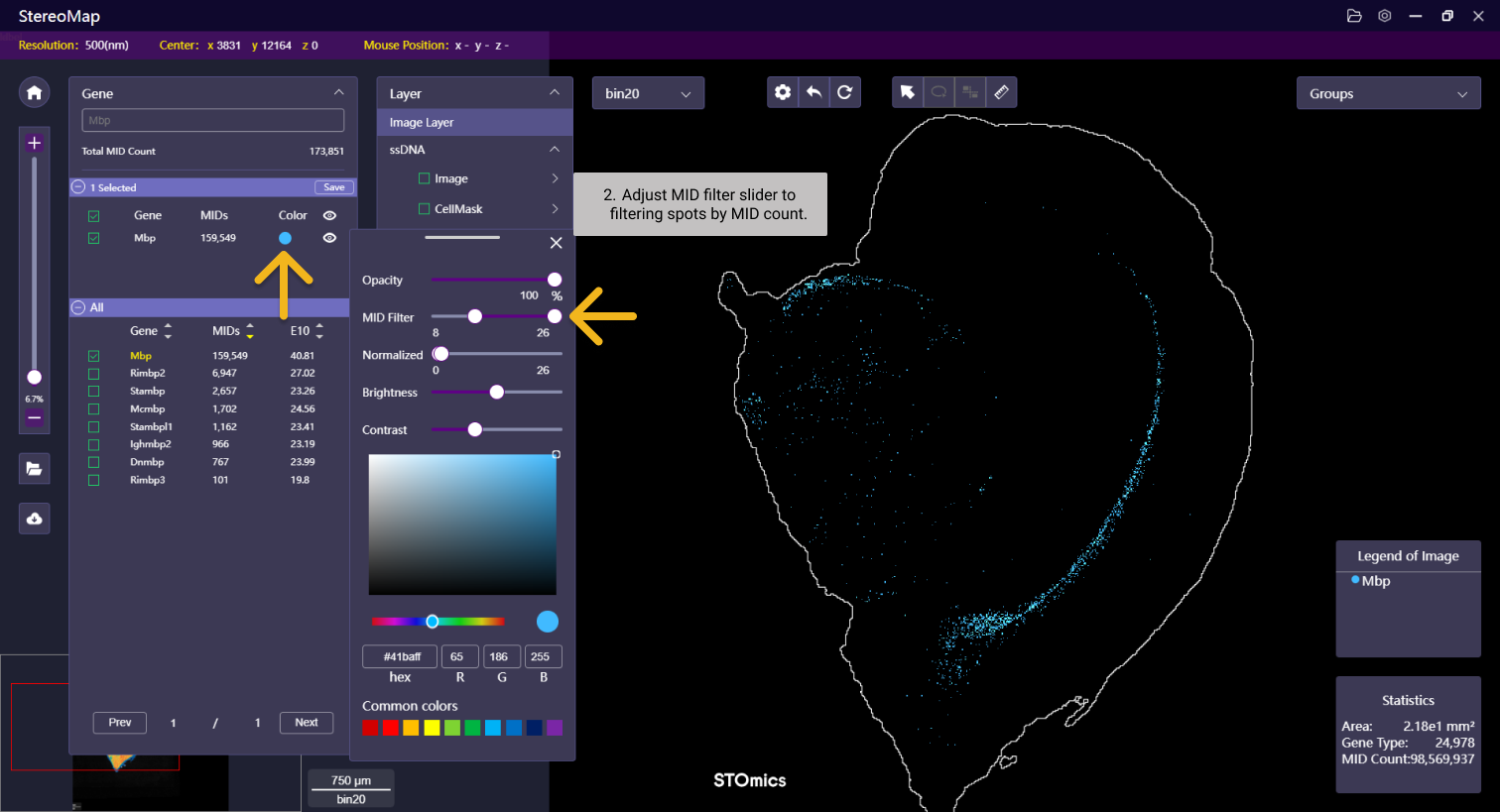
MID 过滤
在进行下游分析之前,对空间转录组数据进行预处理消除噪音至关重要,MID 过滤功能主要用于手动去除每个选定特征的高/低表达的点,聚焦于其空间的分布。
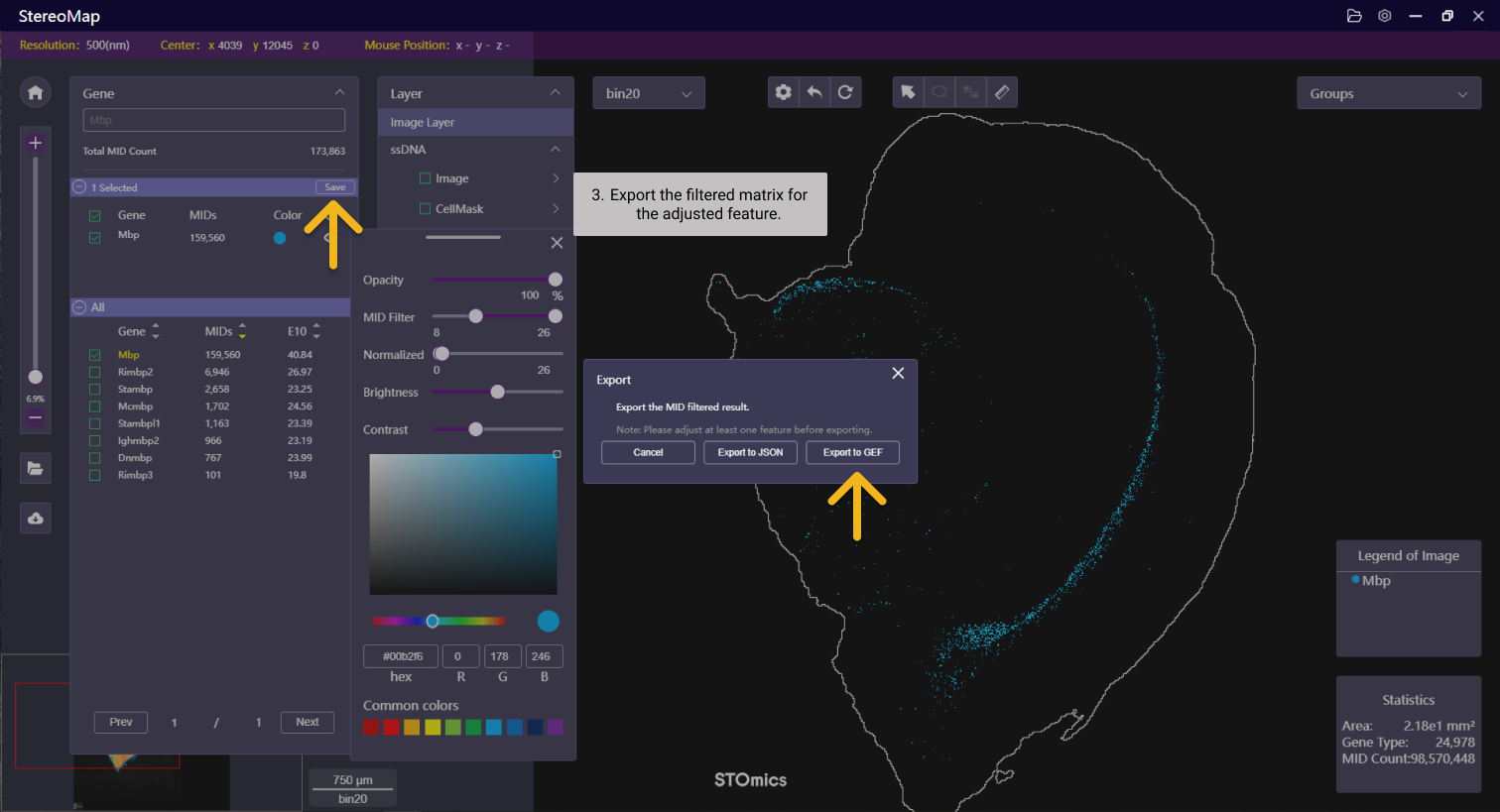
该过滤功能可以单独调整选定的特征点,设置不同的阈值。阈值的的大小代表了过滤的数据范围,会随着bin 的大小发生变化。因此建议您切换到后续计划分析中使用的 bin 大小再调整MID的阈值。输出的矩阵包含每个特征过滤后的结果,可用于下游分析。
考虑到计算资源的限制,如果您的数据是 Stereo-seq 定制化大芯片,强烈建议您使用 MID 过滤输出的 JSON 参数文件而不是 GEF 文件。如果是 JSON 文件,需要通过 --mid-json 参数传递给 SAW midFilter 流程提取矩阵文件。
saw reanalyze midFilter \
--gef=/path/to/input/GEF \
--mid-json=/path/to/MID/filtering/JSON \
--output=/path/to/output/mid_filtering
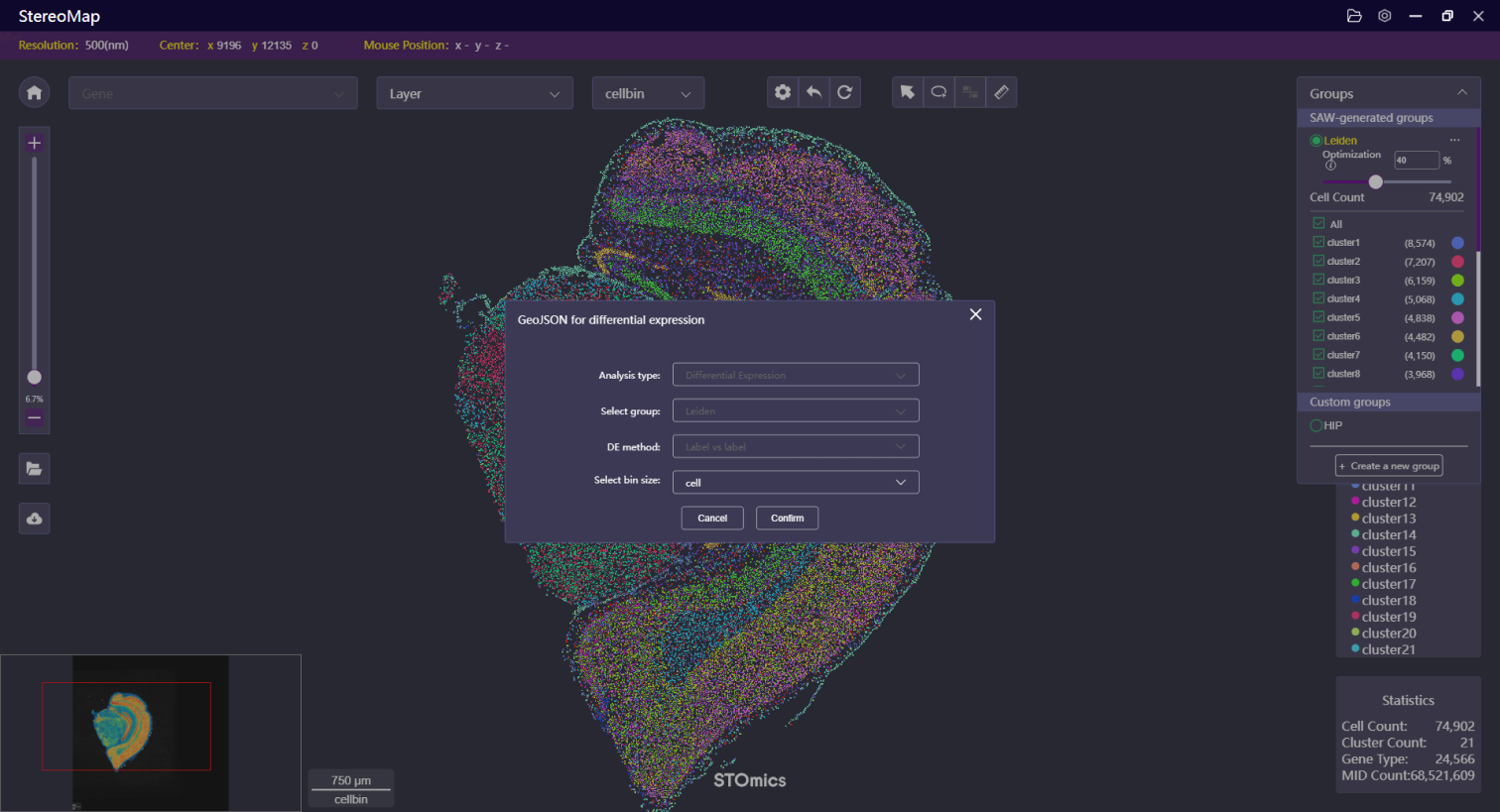
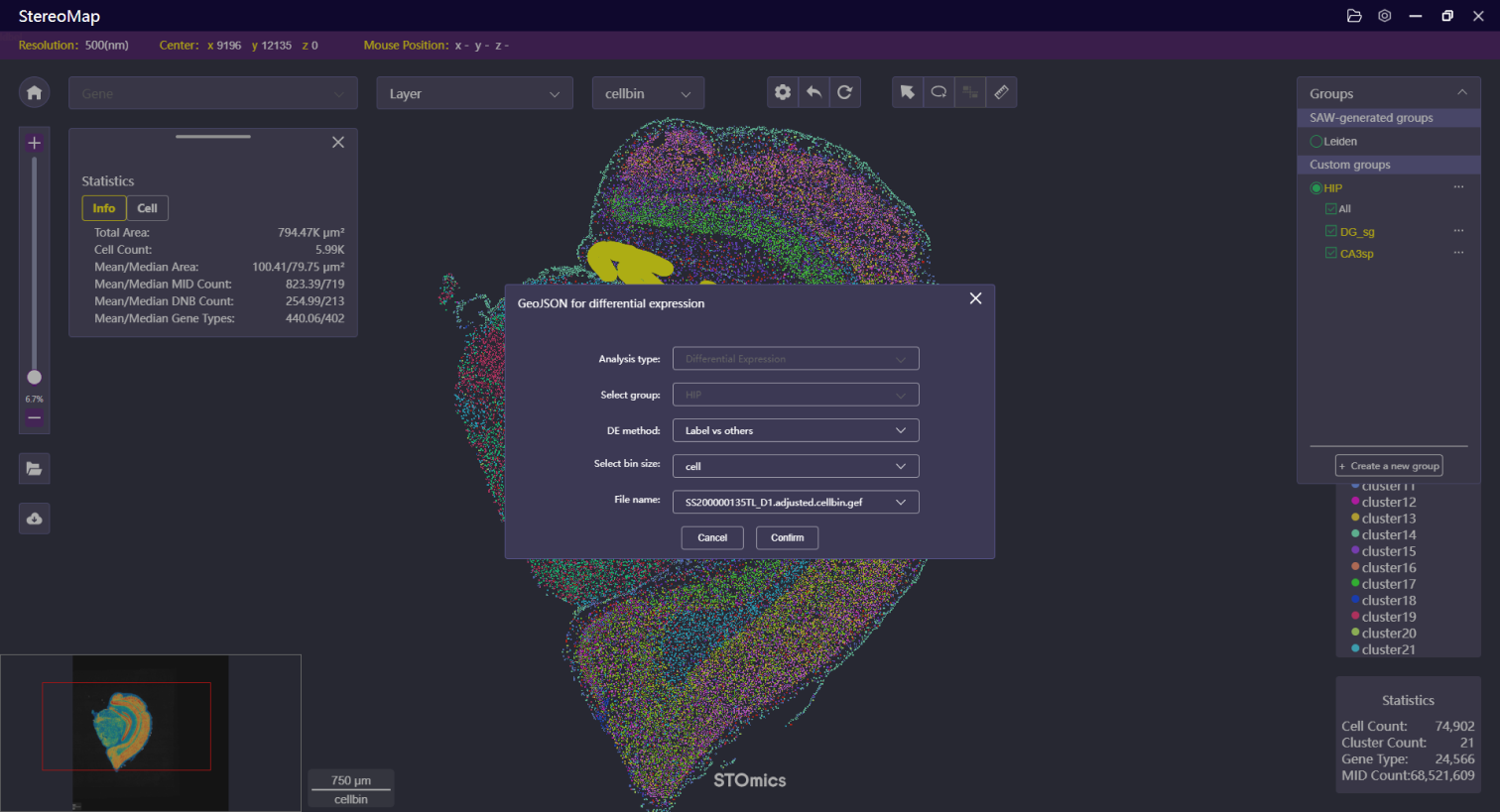
差异分析
新功能, 必须用 SAW >= V8.0 流程版本输出的可视化数据
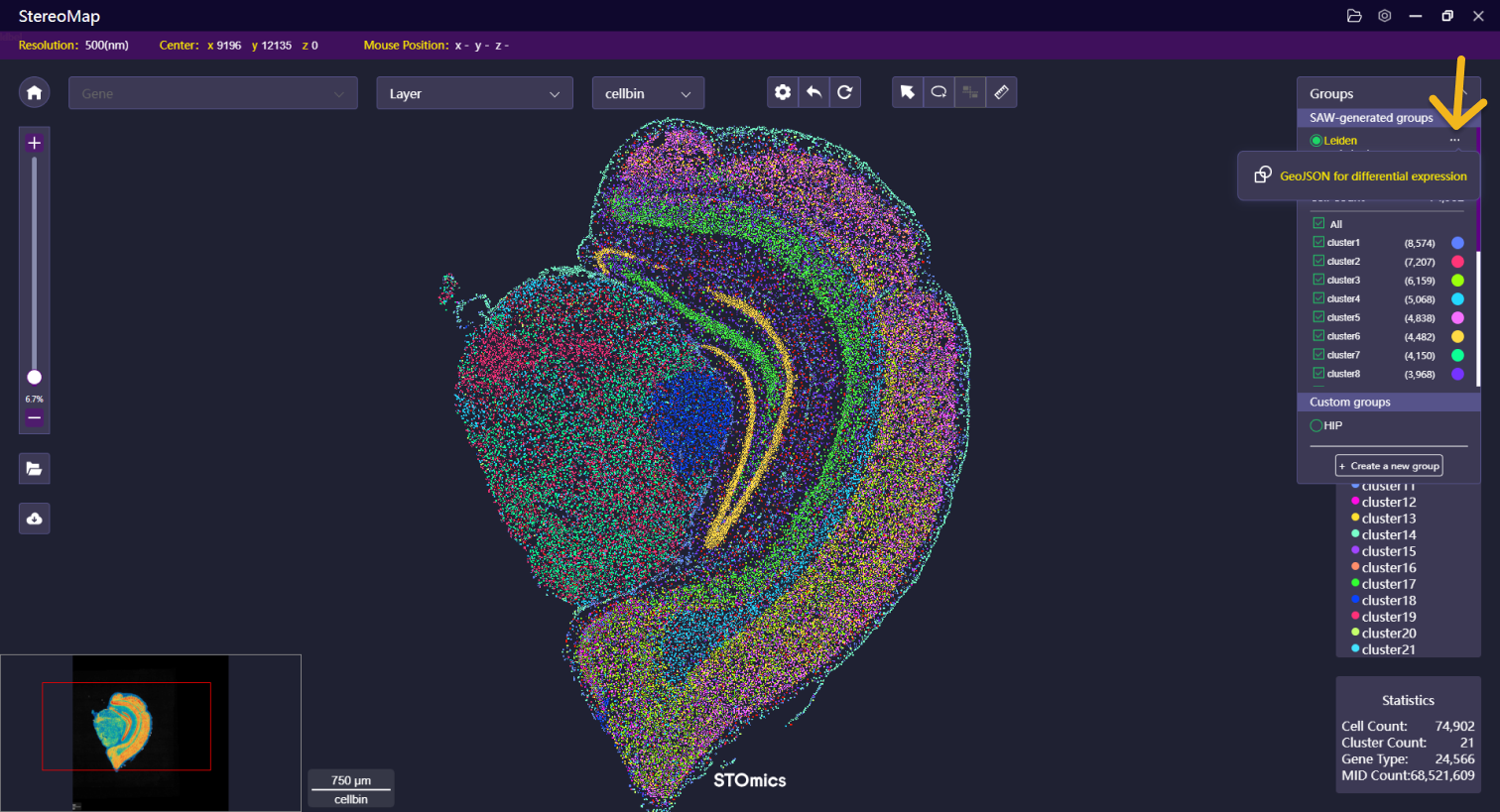
差异分析模块通常针对聚类文件、空间区域标记或者Lasso 某个区域等进行。
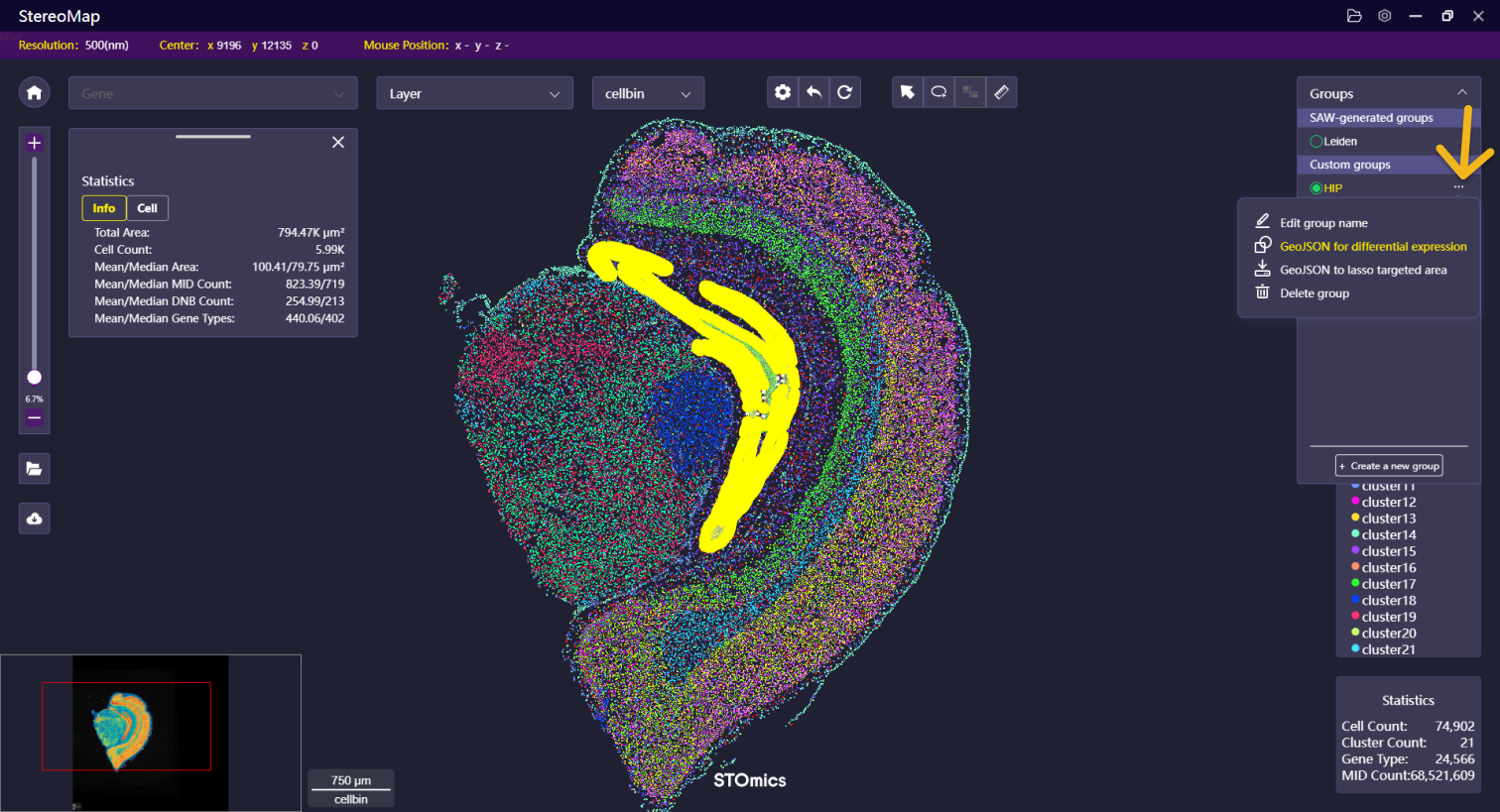
对于聚类文件,可以点击右侧Group面板的  选项,点击 GeoJSON for differential expression,填写必要的参数信息并确认导出后,输入到
选项,点击 GeoJSON for differential expression,填写必要的参数信息并确认导出后,输入到SAW reanalyze diffExp 模块分析结果。
对于lasso 圈选某个区域,您需要在某个组中至少创建 2 个 Label 区域。
其次,点击右侧 Group 面板的  选项,选择 GeoJSON for differential expression。填写必要的参数信息后,点击 Confirm 导出输入到
选项,选择 GeoJSON for differential expression。填写必要的参数信息后,点击 Confirm 导出输入到SAW reanalyze diffExp 模块分析结果。点击 Confirm 后您的资源管理器将会打开,您可以选择想要保存输出的 YYYYMMDDHHMMSS.diffexp.geojson 文件的目录。
两种差异分析方法说明:
- Label vs. others: 用来识别特定 label 或者聚类跟其它所有 label 或者聚类之间的差异分析。
- Label vs. label: 用于识别特定 label 或者聚类与已选择 label 或者聚类之间的差异分析。
您可将文件的路径通过 --de-geojson 参数传递给 SAW reanalyze diffExp流程做后续分析。
saw reanalyze diffExp \
--count-data=/path/to/previous/SAW/count/result/folder/id \
--diffexp-geojson=/path/to/StereoMap/diffexp/YYYYMMDDHHMMSS.diffexp.geojson \
--output=/path/to/output/folder
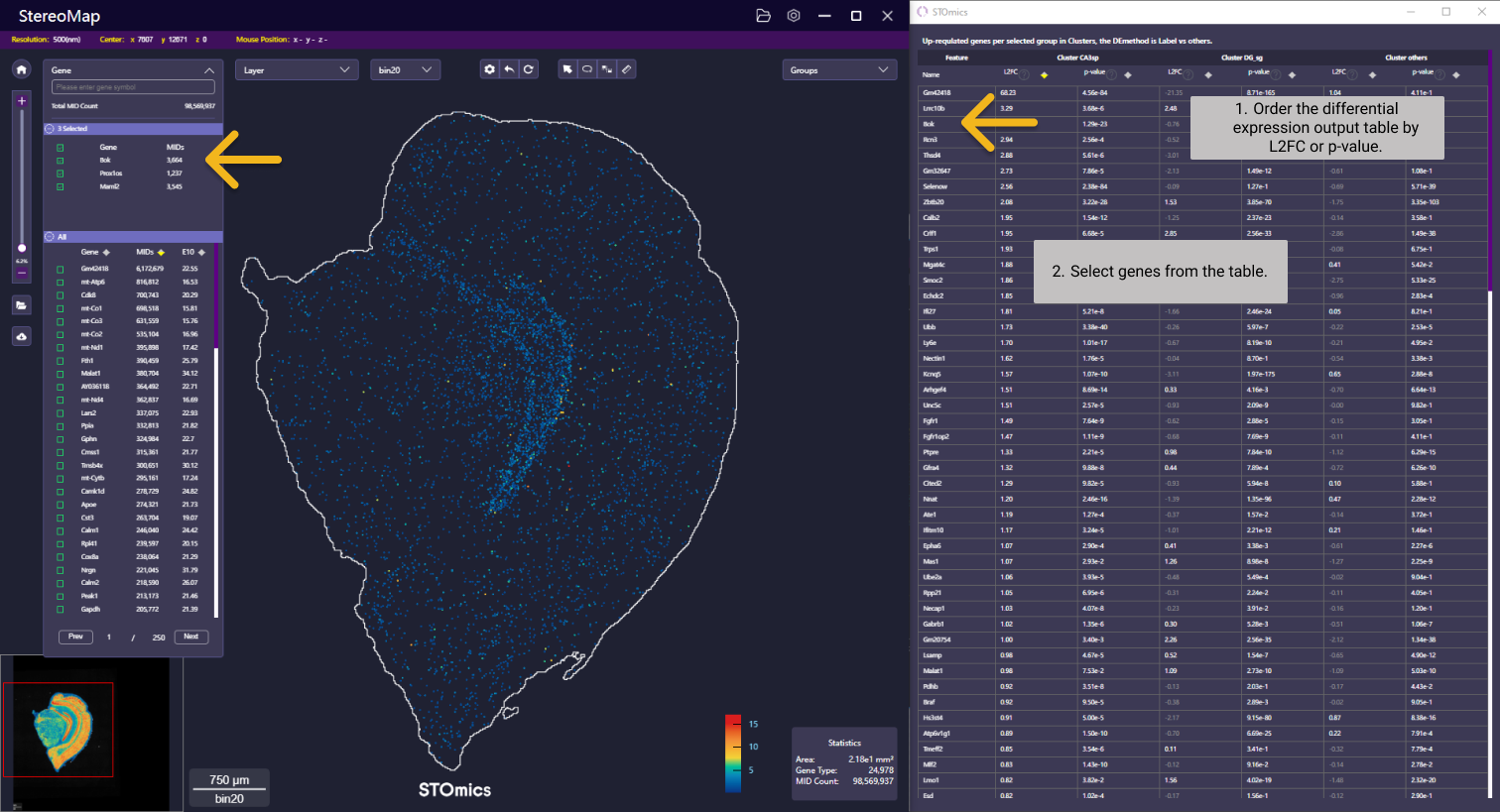
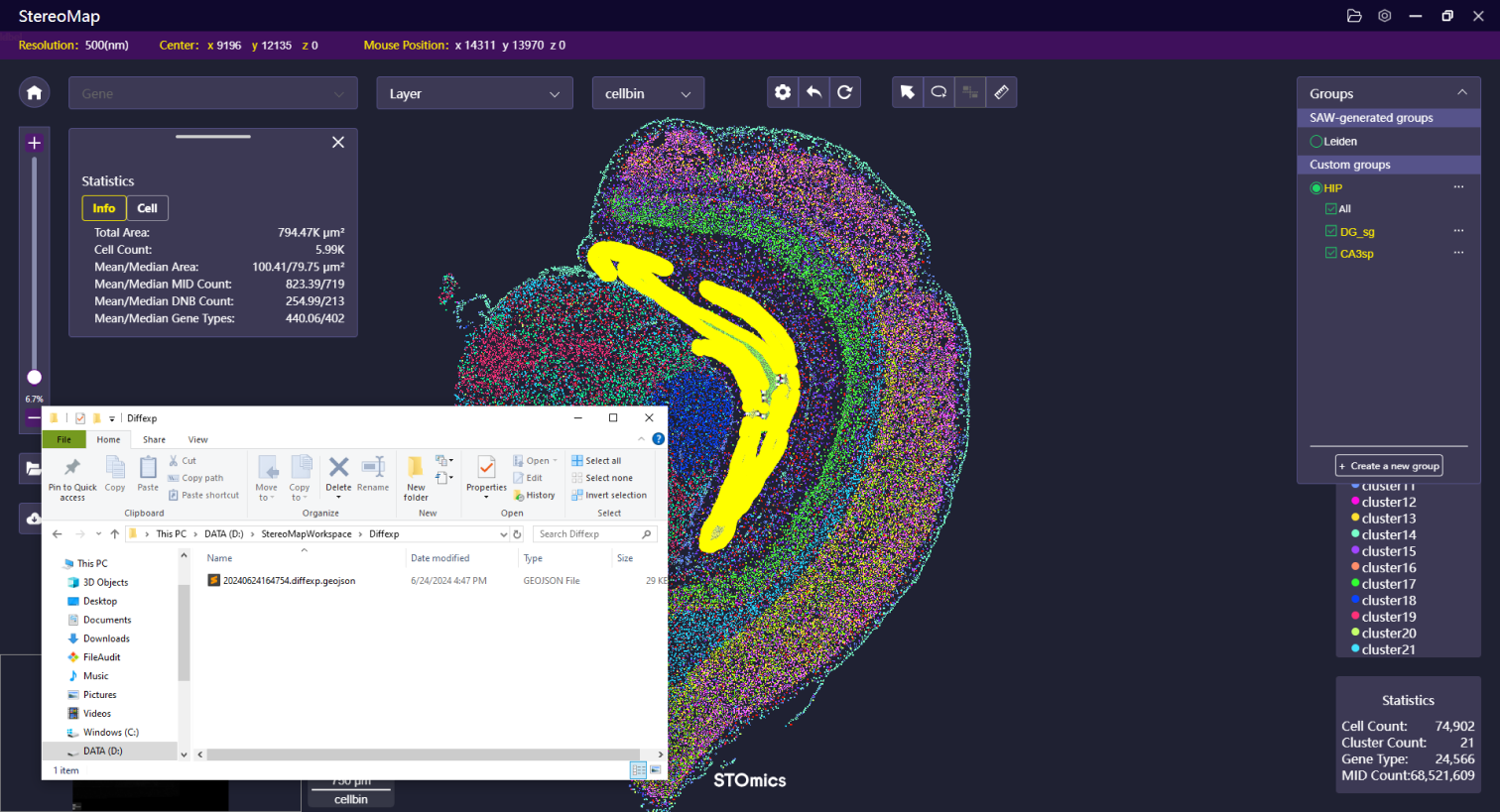
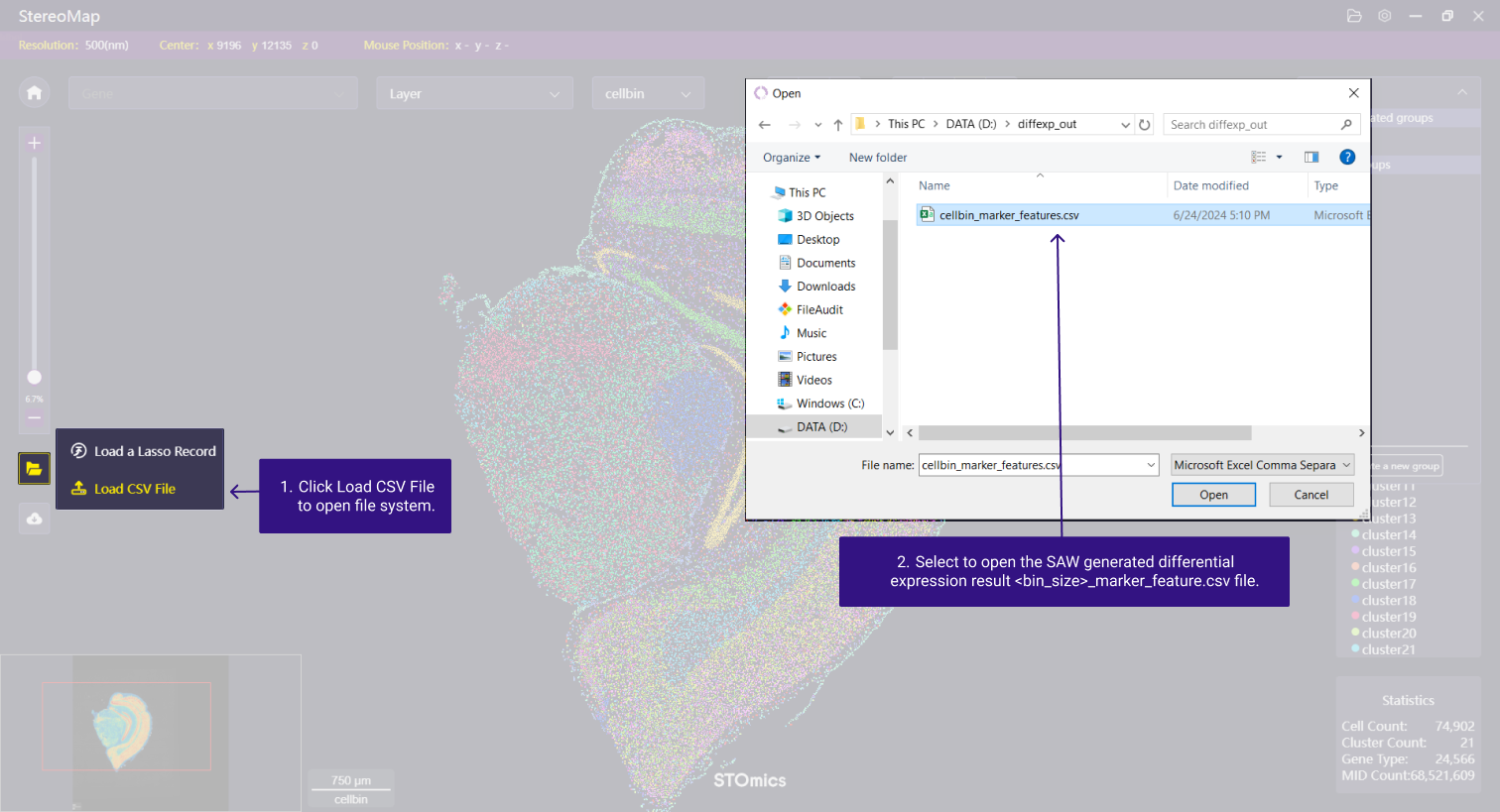
SAW 输出的 差异分析结果文件(<bin_size>_marker_feature.csv) ,可以点击 StereoMap 的 Load CSV file (参考 Feature 菜单-> Load and Save)展示查看。
差异分析结果文件将会在一个新窗口打开,用户可以点击 L2FC 或者 p-values 的排序箭头对表格进行重新排序,以查看其显著特征。
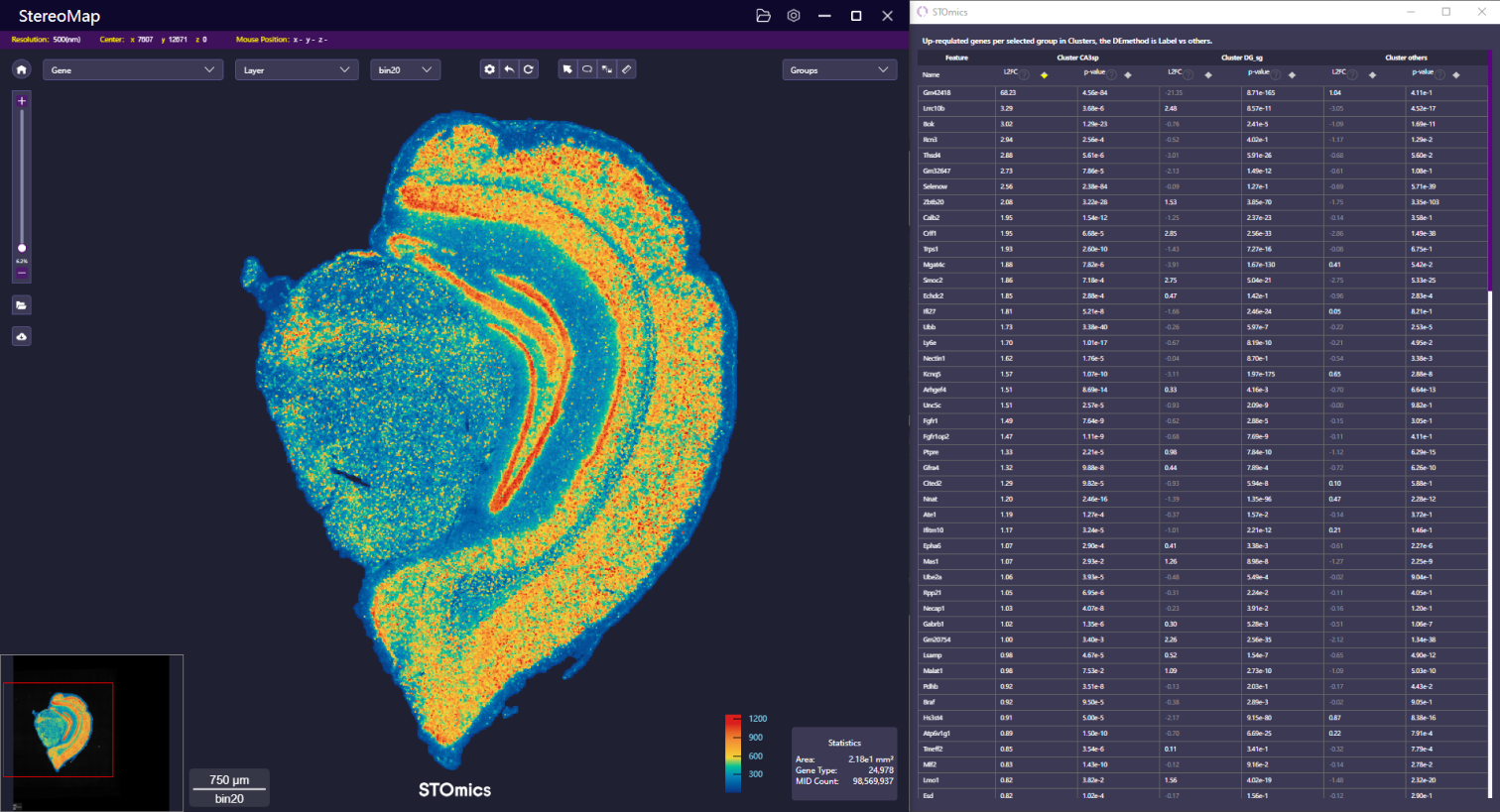
点击表格中的特征名称,画布将会展示该特征对应的表达热图。此外,对于多个特征的分析,您可以通过不同的颜色展示他们,从而做基因共表达分析。
.png)
.png)