手动图像处理
为什么要进行手动处理?
- 在某些情况下,
SAW count的输出结果不够完美,分析人员可能希望手动调整图像(需要结合表达矩阵),或使用第三方工具的算法处理结果,来得到令人满意的分析结果。 - 当数据的图像 QC 结果为不成功时,分析人员必须手动完成显微镜图像与空间特征表达矩阵之间的配准。
通常来说,细致的手动调整会让分析流程生成更准确的表达矩阵数据,用于下游生信分析。
SAW realign 概览
FF 和 FFPE 组织样本经手动处理过后都可以使用 SAW realign 重启分析, 在启动分析任务前,通常需要准备好以下文件:
- 相关联的
SAW count的输出目录; - 来自 StereoMap 的经过手动调整的图像
.tar.gz文件(包含手动处理信息)
来自 StereoMap 的重新对齐的压缩图像.tar.gz文件,保存了原始显微镜图像、图像 QC 信息和手动处理的信息记录。
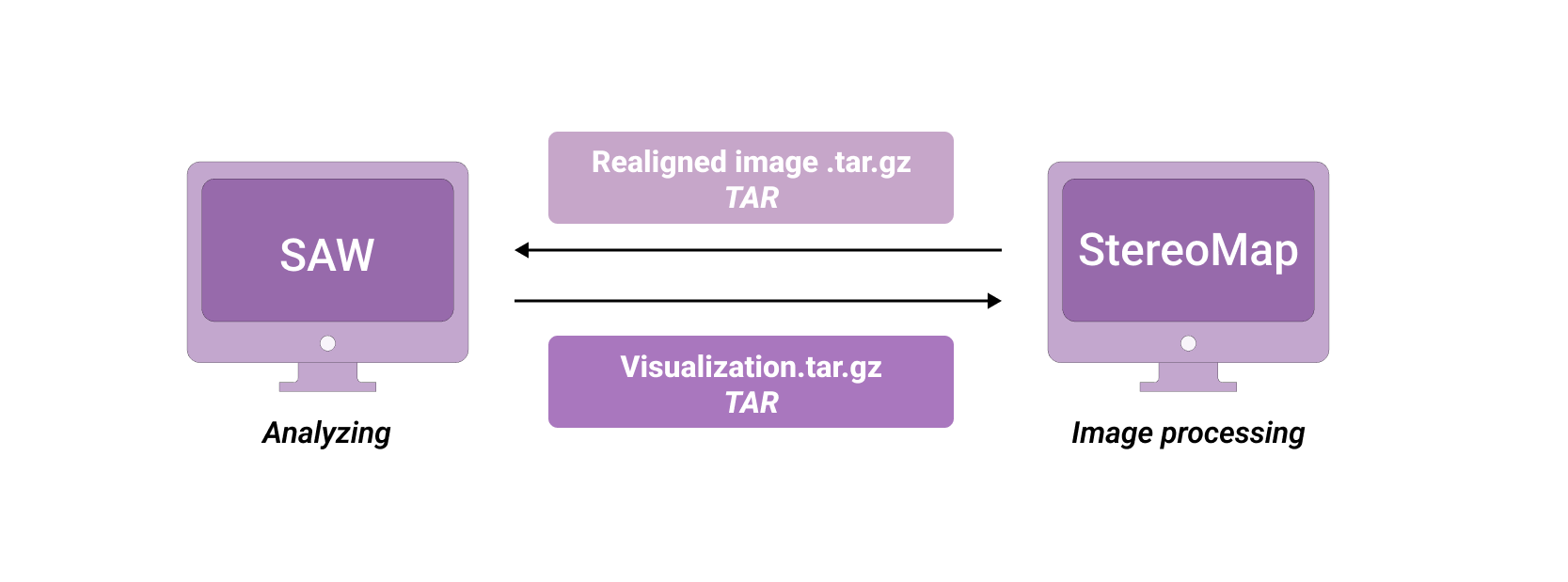
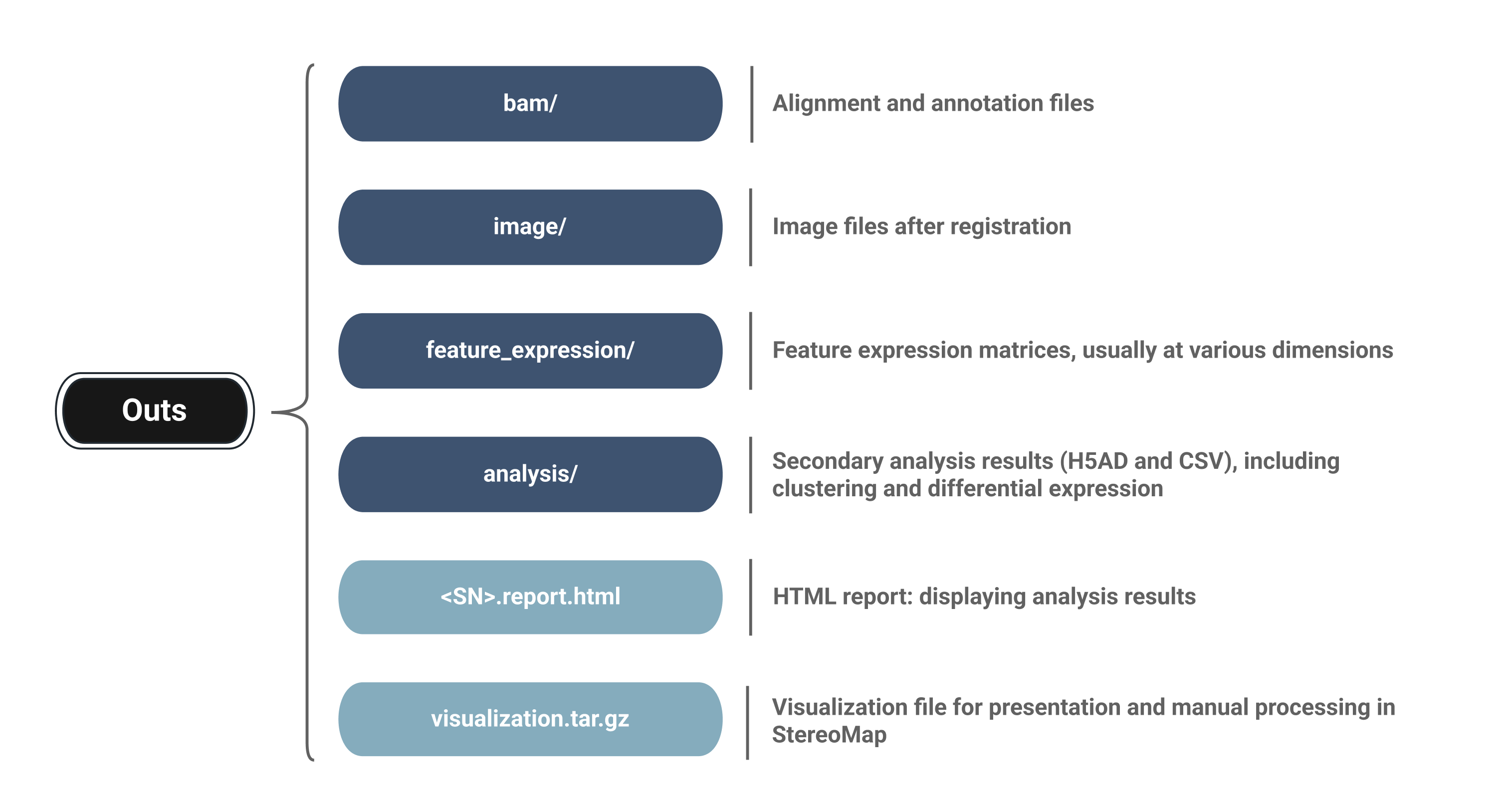
输出结果主要包括:
- 由关联
SAW count分析而来的表达矩阵相关数据; - 处理后的图像文件;
- 不同维度的基因表达矩阵;
- 聚类和差异表达分析结果;
- 可视化文件
visualization.tar.gz, 用于 StereoMap。
| Parameter | Description |
|---|---|
-id <ID> | (Optional, default to None) A unique task id ([a-zA-Z0-9_-]+) which will be displayed as the output folder name and the title of HTML report. If the parameter is absent, --sn will play the same role. |
--sn <SN> | (Required, default to None) SN (serial number) of the Stereo-seq chip. |
--count-data <PATH> | (Required, default to None) Output folder of the corresponding SAW count result, which mainly contains the expression matrices and other related datasets. |
--realigned-image-tar <TAR> | (Required, default to None) Compressed image file from StereoMap, which has been manually processed, including stitching, tissue segmentation, cell segmentation, calibration and registration. |
--lasso-geojson <GEOJSON> | (Optional, default to None) Lasso GeoJSON from StereoMap is used for tissue segmentation when the analysis is without images. It is incompatible with --realigned-image-tar. |
--adjusted-distance <INT> | (Optional, default to 10) Outspread distance based on the cellular contour of the cell segmentation image, in pixels. Default to 10. If --adjusted-distance=0, the pipeline will not expand the cell border. |
当你没有提供显微镜图像数据用于分析时, StereoMap 输出的 lasso GeoJSON 可用作于进行再一次的组织分割,以提取新的 .tissue.gef 矩阵。
--lasso-geojson与 --realigned-image-tar 不兼容。
第一次运行 SAW count
SAW count 的输入数据大致可分为三类:
- 图像 QC 结果为成功
- 包括在 StereoMap 图像处理步骤2中完成了打点配准
- 图像 QC 结果为失败
- 没有显微镜图像
以上三种情况,运行 SAW count 后,输出目录中都会包含下列文件:
Demo_Data
├── pipeline-logs
├── STEREO_ANALYSIS_WORKFLOW_PROCESSING
└── outs
├── analysis
├── bam
├── feature_expression
├── image
├── <SN>.report.tar.gz
└── visualization.tar.gz
visualization.tar.gz不仅用于可视化展示,还可在 StereoMap 中进行图像手动调整,调整结果将记录在压缩图像 .tar.gz 文件中。
对于 QC 失败图像或无图像的分析任务,组织分割是基于表达矩阵数据进行的,因此这两种情况下的任务不会调用标准分析流程中的自动图像算法。
手动处理
StereoMap 中可以实现手动调整,包括图像-矩阵配准、手动组织分割和手动细胞分割,所有这些操作都可以 StereoMap 的Image Processing模块完成,实时看到修改修过,所见即所得。
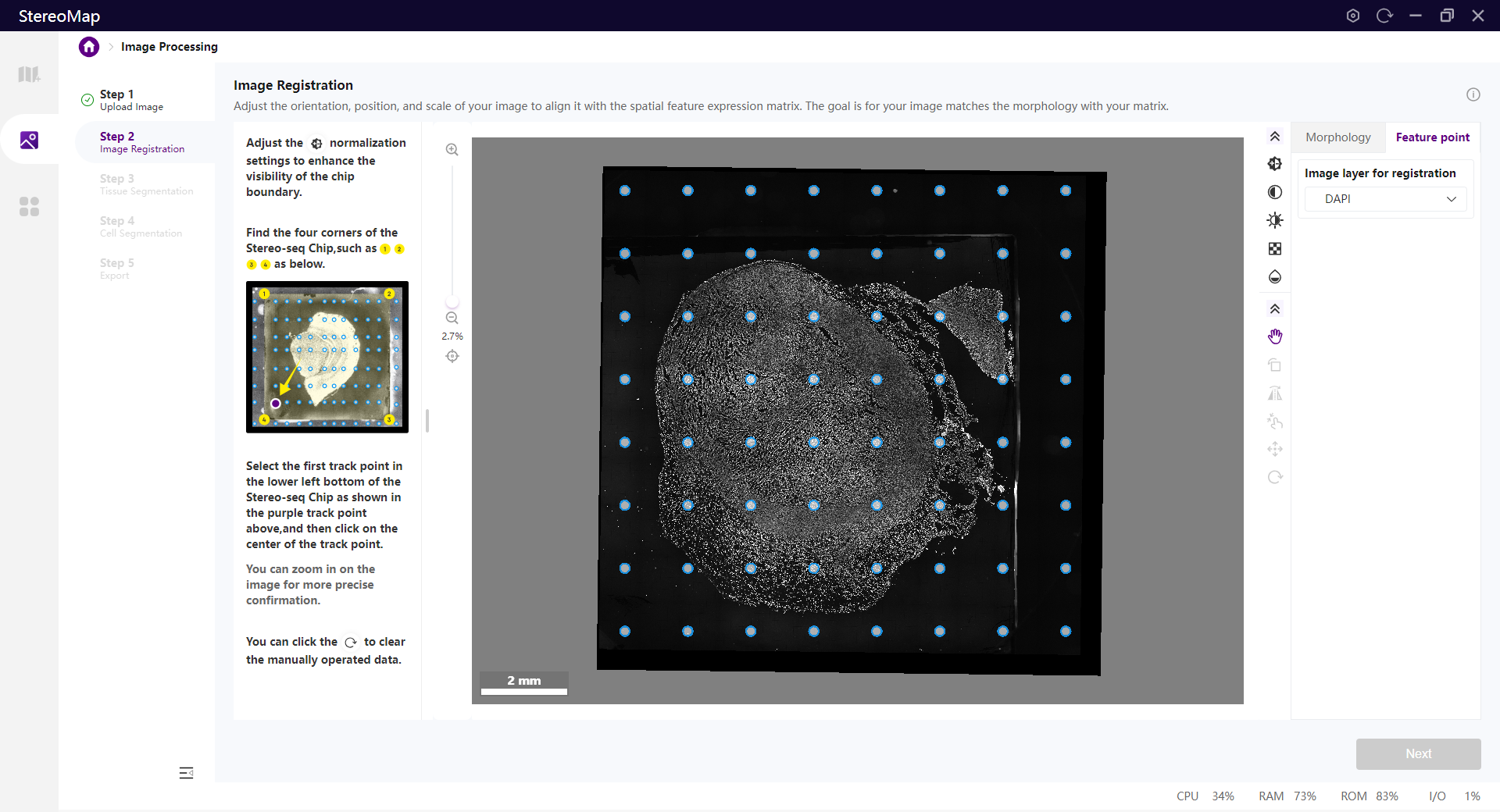
配准
形态学配准
图像形态学配准需要同时使用表达矩阵和显微镜图像,根据组织形态,分析人员可以将两个图像层靠拢对齐,直到配准效果达到分析人员的要求,配准操作对于后续步骤非常重要。
.png)
打点配准
打点配准操作只需要一张显微镜图像。StereoMap 会根据你给出的芯片左下角第一个特征点,以及预定义规则的芯片 mask 信息,自动计算影像图与矩阵配准参数。需要注意的是:
- 图像 QC 结果为成功,因为要确保 track 线是可见的。
- 有预定义规则的 Stereo-seq 芯片: Stereo-seq N FFPE V1.0, Stereo-seq T FF V1.3, or Stereo-CITE T V1.1 。
- 输入
.tar.gz或.stereo文件中对应的.tar.gz是由 StereoMap >= 4.1 生成的
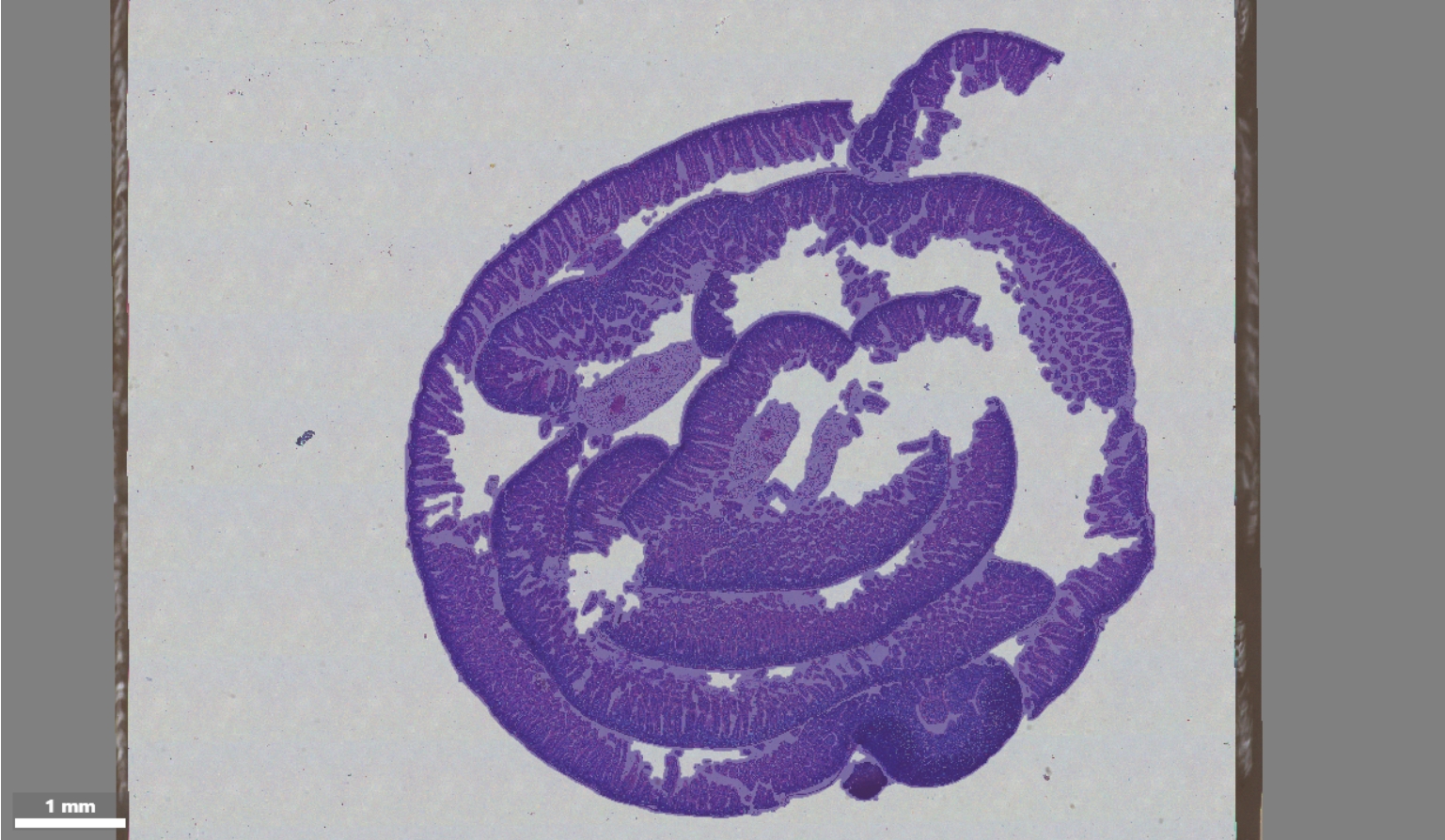
组织分割
组织分割是指通过识别样本的生物学组织形态来探测其组织区域,借助显微镜图像,分析人员可识别组织样本在 Stereo-seq 芯片上的相对位置。
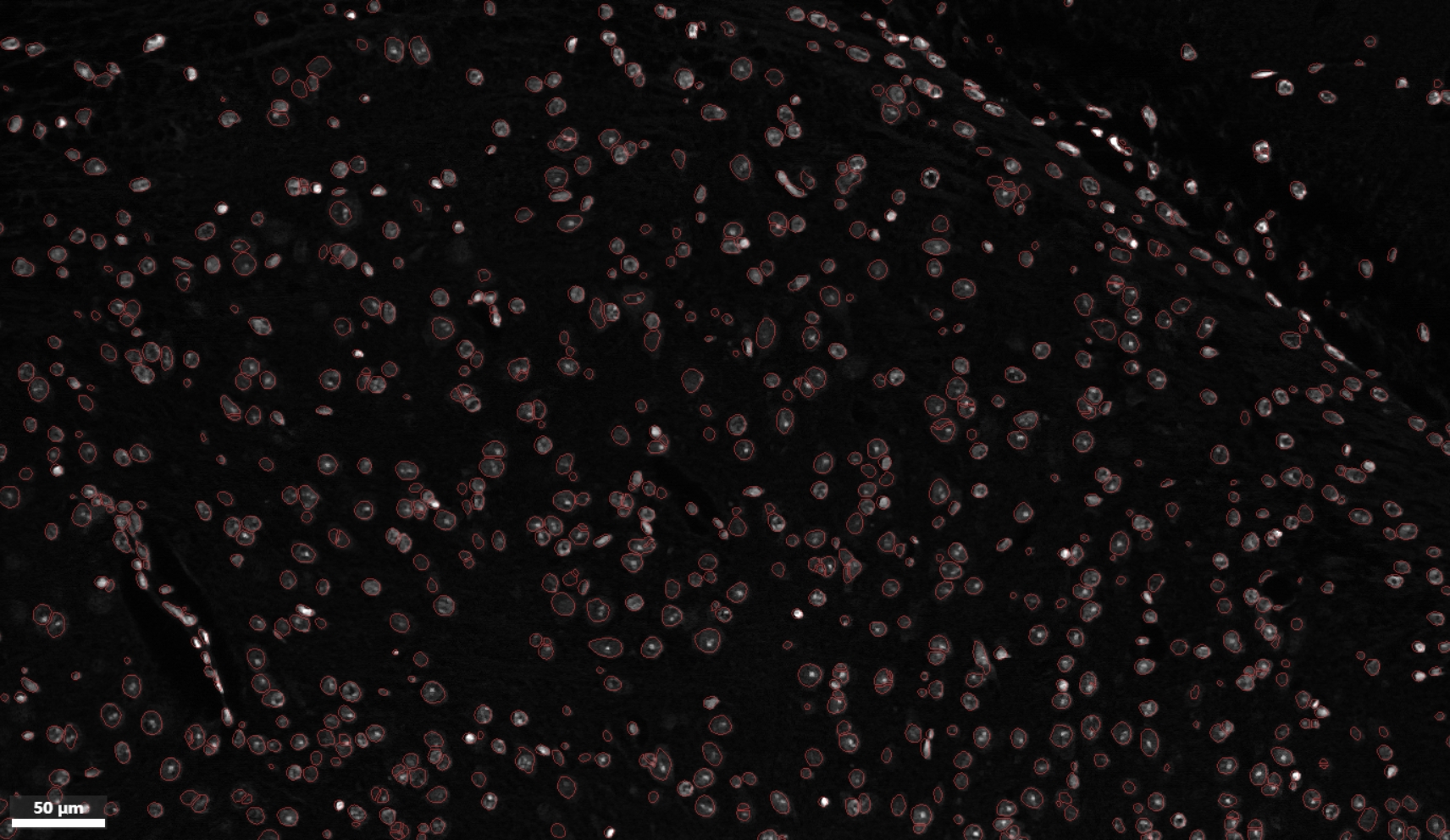
细胞分割
细胞分割是指通过识别细胞的形态来探测单个细胞的区域,根据细轮廓信息,可以进行细胞维度的深入分析和研究。
组织分割和细胞分割是生物信息学分析中的关键步骤,适当的分割结果对后续表达矩阵数据的提取和下游分析具有重要影响。
回到 SAW realign
使用 QC 成功的图像
本教程的第三部分至关重要,因为上一次 SAW count 分析任务的输出结果将帮助 SAW realign 以完成剩余工作,直到生成 HTML 报告。
cd /saw/runs
saw realign \
--id=Adjusted_Demo_Mouse_Brain \
--sn=SS200000135TL_D1 \
--count-data=/path/to/previous/SAW/count/task/folder \
--realigned-image-tar=/path/to/realigned/image/tar
SAW realign 和 SAW count 一样,会根据细胞分割结果自动对细胞轮廓进行外扩,默认为 10 个像素,可以通过设置 --adjusted-distance 来调整,可根据不同的组织样本和细胞类型来调整细胞轮廓的校正程度。
DNB 是 Stereo-seq 芯片上的捕获单元。
DNB 对应配准后图像的正方形像素块的顶点,如果细胞轮廓的扩展距离为 X 像素,则其物理距离估算为 X * 500 nm。
假如对细胞分割结果(手动处理或第三方工具生成的结果)非常满意,可以设置 --adjusted-distance=0 来关闭细胞修正功能。
如果只想获得手动调整后的压缩图像 .tar.gz 文件中的图像数据或表达矩阵矩阵,可以调用 --no-matrix 或 --no-report 参数。
运行分析,如下所示:
cd /saw/runs
saw realign \
--id=Demo_Mouse_Brain_Only_Images \
--sn=SS200000135TL_D1 \
--count-data=/path/to/previous/SAW/count/task/folder \
--realigned-image-tar=/path/to/realigned/image/tar \
--no-matrix
使用 QC 失败的图像
在重启分析之前,必须对图像 QC 失败的数据完成手动配准操作,需要结果显微镜图像与表达矩阵。
SAW 认为,经过手动配准操作(与表达矩阵)的 QC 失败的图像,可以通过调用流程中的自动图像算法进行分析。
cd /saw/runs
saw realign \
--id=Adjuated_Demo_Mouse_Brain \
--sn=SS200000135TL_D1 \
--count-data=/path/to/previous/SAW/count/task/folder \
--realigned-image-tar=/path/to/realigned/image/tar
无图场景
来自 StereoMap 的lasso GeoJSON 文件可以用于 SAW realign 分析流程,重新分割组织区域。
需要确认你使用的lasso GeoJSON文件中仅有一个 label 区域结果,因为SAW realign每次运行仅接收一个组织再分割结果。
仅在无图片输入的场景下有效,--lasso-json 无法与图像相关参数一起使用。
运行分析,如下所示:
cd /saw/runs
saw realign \
--id=Lasso_Demo_Mouse_Brain \
--sn=SS200000135TL_D1 \
--count-data=/path/to/previous/SAW/count/task/folder \
--lasso-geojson=/path/to/lasso/GeoJSON
SAW realign 输出结果
分析任务运行结束后,在工作目录下会生成一个名为 Adjusted_Demo_Mouse_Brain 的输出文件夹,它的命名取决于 --id 参数,当--id参数没有启用时取决于 --sn 参数的信息。
SAW count 分析任务通常在工作目录下开启,在该目录下,将找到一个名为 --id 或 --sn(当--id参数没有启用时)的文件夹。输出结果依据数据类型被分类,主要文件被保存在 /outs下。
下面列出了 SAW realign 的输出文件目录结构和内容:
Adjusted_Demo_Mouse_Brain
├── pipeline-logs
├── STEREO_ANALYSIS_WORKFLOW_PROCESSING
└── outs
├── analysis
├── bam
├── feature_expression
├── image
├── <SN>.report.tar.gz
└── visualization.tar.gz
进一步探究流程输出结果 :
- 跳转至HTML报告解读;
- 熟悉
visualization.tar.gz可视化文件; - 了解输出结果中的各种文件类型。